VI. Caractérisation des bactéries
lactiques
VI.1.Caractérisation
phénotypique
1- La morphologie des colonies et la coloration de Gram
L'observation microscopique est nécessaire pour
définir la forme des cellules bactériennes (coques ou bacilles).
La coloration de Gram des isolats après 72 h de culture sur MRS
modifié permet de s'assurer que nous sommes effectivement en
présence des bactéries gram positive (coloration violette).
2- Le test de catalase
La catalase est une enzyme contenant du fer, elle catalyse la
décomposition du peroxyde d'hydrogène en eau et en
oxygène. Le test de détection de cette enzyme dans une souche
bactérienne, consiste à ajouter du peroxyde d'hydrogène
aux cellules bactériennes, la présence de la catalase se marque
par la formation des bulles de gaz (oxygène).
3- Le test homo/hétérofermentaire
La souche est cultivée dans un bouillon MRS
modifié en bouillon. Le dégagement du gaz carbonique retenu par
la cloche de Durham met en évidence le caractère
hétérofermentaire.
(Sriranganathan et al., 1973).
VI.2.Caractérisation
moléculaire
1- Extraction de l'ADN avec le Kit Qiagen
1 ml de la culture bactérienne en phase exponentielle
de croissance est centrifugé à 7500 rpm pendant 10 minutes.
Après avoir éliminé le surnageant, le culot
bactérien est resuspendu avec 180ul de tampon de lyse (20mM Tris-HCl pH
8 ; 2mM EDTA ; 1,2% Triton X-100 ; 20 mg/ml lysozyme) suivi
d'une incubation à 37°C pendant une heure. 25ul de
protéinase K et 200ul de tampon AL sont ajoutés pour
dégrader toutes les protéines après une incubation
à 70°C pendant 30 minutes. Cette étape est suivie par
l'addition de 200 ul d'éthanol absolu. Le mélange est ensuite
transvasé sur une colonne, puis centrifugé à 8000 rpm
pendant une minute. L'éluant est éliminé et l'ADN est
récupéré sur une colonne dans un tube de 2 ml à
lequel on ajoute 500 ul de tampon AW1. On centrifuge à 800rpm pendant
une minute et on jette l'éluant et le tube. De nouveau, l'ADN est
récupéré dans un tube de 2 ml à lequel on ajoute
500 ul de tampon AW2. On centrifuge à 15 000 rpm pendant 3 minutes
et on jette l'éluant et le tube. On place la colonne sur un tube de
1,5ml et on ajoute cette fois-ci 100 ul de tampon AE directement sur la
membrane. On incube à température ambiante pendant une minute et
on centrifuge à 8 000 rpm pendant une minute. Cette démarche
est renouvelée puis, on jette la colonne et on conserve l'ADN pur
extrait à -20°C.
2- Quantification de l'ADN
Le but est d'obtenir une concentration d'ADN entre 20 et 100
ng/ul pour chaque échantillon avant de réaliser le PCR. La
quantification se fera grâce au spectrophotomètre UVIKON BIOTEK.
La concentration d'ADN sera calculée selon la formule suivante:
D.O à 260 nm x F x A
[ADN] ug/ ul =
---------------------------
1000
Où:
F= facteur de
dilution =10
A= Absorption
ADN= 50; Absorption ARN= 40
3- Les amorces
Les amorces oligonucléotidiques utilisées dans
cette étude sont obtenues d'Invitrogen
Tableau n°4 : Les amorces de
l'étude
|
Code
|
Localisation
|
Séquences oligonucléotidiques
Primers (5'?3')
|
T (°C)
Hybridation
|
Références
|
|
16S/2
|
16S
1389-1407 de E.coli
|
CTT GTA CAC ACC GCC CGT C
|
60°C
|
Gürttler et Stanisich 1996
|
|
23S/10
|
23S ARNr
456-474 de E.coli
|
CCT TTC CCT CAC GT ACT G
|
60°C
|
Idem
|
|
23S/7
|
23S ARNr
188-208 de E.coli
|
GGT ACT TAG ATG TTT CAG TTC
|
60°C
|
Idem
|
|
ARNtala
|
ITS
|
TAG CTC AGC TGG GAG AGC
|
60°C
|
Idem
|
|
Ls1
|
|
AAGTCGCCCAATTGATTCTTAGT
|
65°C
|
Zapparoli et al., 1997
|
|
Ls2
|
|
TTCACCCTAATCATCTGTCCCA
|
65°C
|
Zapparoli et al., 1997
|
|
TGGE (L1401)
|
V6-V8
|
GCG TGT GTA CAA GAC CC
|
56°C
|
Zoetendal et al., 1998
|
|
TGGE
(U968-GC)
|
V6-V8
|
CGC CCG GGG CGC GCC GGG GAA CGC GAA GAA CCT TAC
|
56°C
|
Vasquez et al., 2001
Zoetendal et al., 1998
|
4- La PCR
Ø Avec les primers 16S/2 et
23S/7
Le mélange final pour les réactions de PCR a un
volume de 25 ul et contient 1x PCR Mix du tampon Taq polymérase
sans MgCl2, 2,5 mM MgCl2 (Biolabs, New England),
1uM pour chaque amorce (Invitrogen), 0,25 mM pour chaque dNTP
(Eurogentec), 1U de Taq polymérase (Biolabs,
New England) et 20-50ng/ml d'ADN. La réaction du PCR s'effectue dans le
thermocycleur PTC-100 (MJ Research Inc., Watertown MA, USA). L'amplification
des ISRs comprend 35 cycles : 1 minute de dénaturation à
94°C, 1 minute d'annealing ou d'hybridation à 60°C et 1
minute d'extension à 72°C.
Ø Avec les primers tRNAala et
23S/10
La composition du mélange PCR et les conditions
d'amplifications sont les mêmes que celles qui sont décrites
ci-dessus. Cette deuxième réaction d'amplification est faite
après l'obtention des résultats de la première
amplification (présence de 2 ou 3 fragments).
Ø Avec des primers spécifiques des
espèces
La composition du mix PCR est la même que
précédemment. L'amplification des ISRs comprend 35 cycles avec
un changement au niveau de la température d'hybridation des amorces qui
est de 65°C.
Ø PCR sur colonies
C'est une méthode rapide et efficace pour la
détection et l'identification de la population des levains de
panification. Des colonies de diamètre compris entre 1-1,5 mm ont
été choisies et mélangées avec 5 ul de l'eau
stérile. Cette suspension sera incubée 5 minutes à
95°C. On centrifuge à l'aide d'une micro-centrifugeuse et 1-3,5 ul
sont utilisés pour la réaction de PCR. Les conditions
d'amplifications sont les mêmes que celles qui sont décrites plus
loin et la composition du mélange PCR reste le même à
laquelle on ajoute 0,1 x BSA.
5- L'électrophorèse sur le gel d'agarose
Les molécules placées dans un champ
électrique migrent vers l'anode. Dans le gel, la mobilité
électrophorétique dépend de la taille des molécules
et de leur conformation. Les molécules de forme linéaire migrent
d'autant plus vite qu'elles sont de petites tailles. Les produits
d'amplifications seront séparés sur un gel
d'électrophorèse à 1,5% d'agarose dans une solution tampon
TAE colorée au BET. La migration se fait sous un voltage de 100V.
L'observation de chaque gel se fera sur le transilluminateur à UV
grâce à la fluorescence sous UV du complexe ADN-UV. Afin de
déterminer la taille de chaque fragment, le marqueur de poids
moléculaire 100 pb DNA ladder sera utilisé.
1517 pb
1200 pb
1000 pb
900 pb
800 pb
700 pb
600 pb
500 pb
400 pb
300 pb
200 pb
100 pb

Figure
n°6 : Le marqueur 100 pb DNA Ladder-Biolabs
6- La restriction enzymatique (RFLP)
Les produits du PCR avec les primers tRNAala -
23S/10 sont digérés par des enzymes de restriction. Les enzymes
utilisées sont : HindIII, HinfI et
áTaqI (Biolabs). La réaction de enzymatique se
réalise avec un volume final de 20 ul contenant 1x du tampon 0,1 x BSA,
10U de l'enzyme et 16 ul du produit du PCR. Les produits de digestion sont
séparés sur un gel d'électrophorèse dans 2%
(wt/vol) agarose-résophore dans le TBE comme tampon (1x). Les gels sont
colorés par 0,5 ug/ml de BET et visualisés sous UV. Les
photographies sont prises par une vidéo caméra (Sony, Clichy,
France) et sauvegardés. Ils seront analysés par le logiciel Bio
Profile 1D++ (Vilbert Lourmat, Marne La Vallée, France).
TTGE
Ø Extraction et purification de l'ADN total
bactérien
L'extraction de l'ADN total bactérien est décrit
par Meroth et al., (2002) avec des petites modifications. 10g de
levain sont homogénéisés pour 5 minutes par un stomacher
dans un sac contenant 90 ml d'une solution de tryptone-sel. Un aliquote (50 ml)
est centrifugé à 4°C pour 5 minutes à 100 rpm. Le
surnageant est centrifugé pour 15 minutes à 4000 rpm et le culot
cellulaire est stocké à -20°C. Le culot est ensuite
déposé sur glace et lavé 3 fois par 1 ml d'une solution
tampon de phosphate saline et une fois par 1 ml de l'eau. Puis on le resuspend
dans 130 ul d'un tapon lyse ( 6 ,5% sucrose, 50mM Tris HCl pH 8, 0,1 mM
EDTA, 20 mg/ml lysozyme et 100ug/ml RNase). Après une incubation d'une
heure à 37°C, on ajoute 30 ul du NaCl (5M) et le tout est
incubé pour 10 minutes à 65°C. Ensuite, on ajoute
successivement 20 ul de protéinase K (15mg/ml) et 10 ul de sodium
dodécyle sulfate (20%). Le nouveau mélange est incubé
pendant 30 minutes à 60°C. Enfin, 200 ul de phénol
(65°C, pH =7) ont été ajoutés à la mixture et
incubée pour 6 minutes à 60°C. Après le
refroidissement sur glace, on procède 2 fois à l'extraction et
avec des volumes égales de phénol-chloroforme-isoamyl alcool
(25 :24 :1) et 2 fois par du chloroforme. Après la
précipitation par de l'éthanol, l'ADN est gardé dans 100ul
de Tris-HCl (10 mM, pH =8).
La PCR-TTGE est obtenue en utilisant les deux primers
amplifiant les régions variables V6-V8 du gène ARNr. La PCR est
effectuée dans un volume total de 50 ul contenant 1x tampon PCR sans le
chlorure de magnésium, 2,5 mM MgCl2, 1 ug/ml ADN, 0,3 uM pou
chaque primer, 0,25 mM dNTP et 1U de Taq polymérase. On a les
mêmes conditions des PCR précédentes. Les produits du PCR
sont séparés sur 1,5 % (wt/vol) du gel d'agarose (Eurobio,
France) et consécutivement visualisés sous la lumière UV
après une coloration par le BET.
Ø Les analyses du TTGE
10 ul du produit amplifié par PCR sont analysés
par la TTGE sur le gel du polyacrylamide (composition et préparation
dans l'Annexe 1). L'analyse du TTGE est effectuée en utilisant DCode
Universal Mutation Detection System (Bio Rad, Marnes la coquette, France). Le
gel du polyacrylamide 16 cm/16cm/1 mm est utilisé avec le tampon 1x TAE
(2M tris base, 1M acide acétique glacial, 50mM EDTA). Les gels ont
été préparés avec 11 % (wt/vol) de la solution
d'acrylamide (acrylamide-bisacrylamide ; 37,5 :1) et une
concentration d'urée finale de 9,7M. Pour séparer les
bactéries étudiées, les conditions suivantes ont
été admises : voltage -55V, température initiale
-65,5°C, température finale - 70,5°C, gradient de
température 0,2°C/h et le temps de migration est de 18 heures. 10
ul du produit de PCR sont introduites dans les pores. En outre, le gel est
coloré pour 15 minutes par le colorant SYBR Green I
(Molecular Probes, Eugene OR, USA) et photographié sur
la table disposant d'un transilluminateur à UV.
L'objectif de cette étude est de montrer l'influence de
la matière première mise en oeuvre sur la flore lactique d'un
levain et notamment sur la diversité des espèces du genre
Lactobacillus. Pour cela deux échantillons de levains
ont été analysés : BF -70 entretenu sur
farine de type 70, et BF-FC rafraîchi sur farine de
blé concassé.
I. Détermination du pH et du TTA
Les résultats sont consignés dans le tableau
n°5 :
Tableau
n°5 : pH et TTA des échantillons des levains
BF
|
Echantillon
|
pH
|
TTA ml/N/10/10g
|
|
BF-70
|
4.03
|
8.04
|
|
BF-FC
|
4.08
|
9.30
|
L'acidité plus élevée du levain BF-FC
peut-être mise en relation avec le taux de cendres supérieur et la
quantité plus importante de parties périphériques du grain
dans le cas du blé concassé.
II. Identification des bactéries lactiques des
levains Français
II.1. Dénombrement des
bactéries lactiques et des levures :
Les dénombrements sont effectués sur les milieux
MRS modifié agar et SDB agar pour les bactéries lactiques et sur
le milieu Sabouraud glucose agar avec chloramphénicol pour la flore
levurienne (Tableau n°6).
Tableau n°6:
Dénombrement des levures et des bactéries lactiques dans les
levains BF
|
Echantillons
|
Bactéries lactiques
|
Levures
|
|
MRS m
UFC/g
|
SDB UFC/g
|
Sabouraud glucose agar
UFC/g
|
|
BF-70
|
1,4.109
|
5,1.109
|
1,5 .108
|
|
BF-FC
|
1,9.109
|
3,1.109
|
1,5.108
|
La population bactérienne observée sur SDB agar
est plus importante que sur MRS modifié agar. Les colonies sont plus
petites sur SDB agar et elles présentent pratiquement la même
morphologie. Les populations obtenues ne présentent pas de
différences significatives entre les deux levains analysés.
Selon les analyses antérieures sur ce levain, Lb.
sanfranciscensis est la flore lactique dominante du levain BF. Le milieu
SDB, de part sa composition (pH qui vaut 5,6 et présence d'extrait frais
de levure) est favorable à la croissance et au dénombrement cette
espèce bactérienne (Kline et Sugihara, 1971). A l'inverse, le
milieu MRS m est moins favorable et ne permet pas le dénombrement de la
totalité des flores présentes dans le levain.
II.2. Identification phénotypique des
bactéries lactiques :
Les étapes d'identification consistent à
réaliser les différents tests phénotypiques comme la
coloration de Gram, le test catalase et le test
homo-hétérofermentaire.
A partir du milieu MRS m, 20 isolats de chaque
échantillon ont été purifiés et soumis à la
coloration de Gram et au test catalase. Les 20 isolats provenant de la farine
BF-70 sont des bacilles Gram positifs, catalases négatives, 18 sont
hétérofermentaires et 2 homofermentaires. Sur la farine
concassée, le même travail a été entrepris. Les 20
isolats obtenus sont des bacilles Gram positif, catalase négative et
tous sont hétérofermentaires.
Tableau n°7 : Ratio
des isolats homo/hétérofermentaire dans les levains
|
Echantillons de levains
|
Bacilles hétérofermentaires
|
Bacilles homofermentaires
|
|
BF-70
|
98 %
|
2%
|
|
BF-FC
|
100 %
|
0 %
|
II.3. Identification moléculaire des
bactéries lactiques :
II.3.1. Identification des isolats du
BF-70 :
L'amplification par PCR avec les amorces 16S/2-23S/7 des 16
isolats hétérofermentaires du BF-70 montre un profil de 3 bandes
sur le gel d'agarose. Ce profil correspond à l'espèce Lb.
sanfranciscensis type de notre étude. En effet selon Valcheva
et al., 2006 la présence de ces 3 fragments est uniquement
observée chez Lb. sanfranciscensis alors que toutes les autres
espèces de Lactobacillus présentent seulement 2 types
d'opérons ribosomiques.
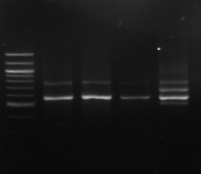
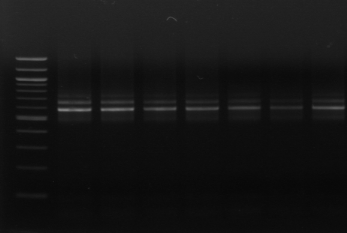
M 1 2 3 4 M
M1 M2 M3 M4 M5 M6 M7
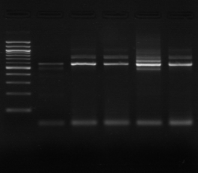
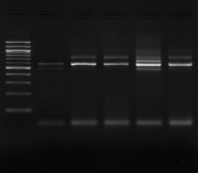
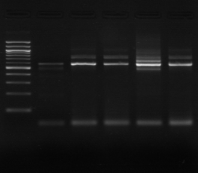
M M8 M9 M10 M11 M M12 M13
M M14 M15 M16
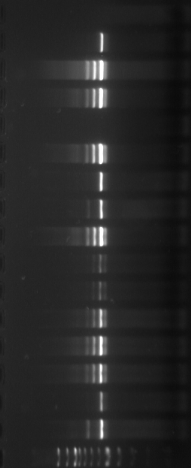

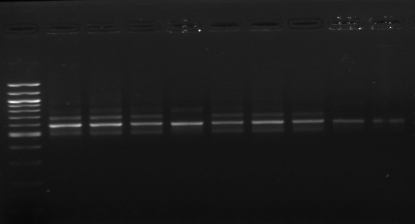
Figure n°7: PCR avec les amorces
16S/2-23S/7 des 16 isolats de BF-70.
M-marqueur 100 pb DNA ladder (biolabs) et
les numéros 1 à 4 correspondent à
l'amplification des souches de
référence utilisées dans cette étude :
1-Lb.
paralimentarius, 2-
Lb.spicheri,3-Lb. kimchii, 4-Lb. sanfranciscensis
ATCC
43322.
M1 à M16 : isolats BF-70.
Après avoir déterminé de manière
probable l'espèce qui domine dans l'échantillon BF-70, nous avons
confirmé ce résultat en procédant à
l'amplification de la région tRNAala -23S/10 par les amorces
correspondantes à cette séquence nucléique dans le but de
faire ensuite la restriction enzymatique .On observe donc la présence
d'un seul fragment d'amplification sur la figure n°8 :
M R M1
M8 M16
740 pb
Figure n°8: Amplification par
les amorces tRNAala -23S/10 (souches : 1,8 et 16)
M-marqueur 100 pb DNA
(Biolabs).
R : souche de
référence Lb. sanfranciscensis ATCC 43322.
On observe ainsi que toutes les souches présentent un
seul fragment de taille identique de 740 pb environ. L'étape suivante va
consister à générer des profils de restriction en
utilisant des enzymes permettant de différencier Lb.
sanfranciscensis. Les 3 enzymes choisis dans notre étude
sont : Hind III, Hinf I et á Taq I.
M M
M M

Figure
n°9: RFLP par Hind III, Hinf I et á Taq I
Pour tous les isolats il génère le même
profil connu et caractéristique pour Lb. sanfranciscensis ATCC
44322.
Seul l'enzyme á Taq I donne un profil unique pour cette
souche de Lb. sanfranciscensis ATCC 43322. Les tailles des fragments
digérés par l'enzyme á Taq I sont les suivantes :
110,140, 160, 310 et 380 pb.
Les autres enzymes génèrent des profils communs
à Lb. paralimentarius DSM13238, Lb. alimentarius CIP
102986T et Lb. delbrueckii subsp. delbrueckii DSM 20074.
Les tailles des fragments des fragments digérés par Hinf I
sont : 60, 210 et 470 pb et par Hind III sont : 320 et 420 pb.
La taille des fragments a été obtenue à
l'aide du logiciel Bio Profile 1D++ (Vilbert Lourmat). Lors de cette
étude, nous avons pris les valeurs moyennes arrondies de tous les essais
par souche et par enzyme de restriction. L'intervalle de confiance
estimé à environ 7% pour chaque fragment à l'aide du
logiciel Bio 1D++.
Pour la confirmation finale des résultats on a choisi
de faire la PCR avec des primers spécifiques de Lb.
sanfranciscensis (Zapparoli et al., 1997) sur les isolats. Elle
permet l'identification définitive de Lb. sanfranciscensis
ATCC 43322 pour les isolats étudiés comme le montre la figure
n°10 où on oberve un seul fragment de 1460 pb spécifique
de cette espèce (Zapparoli et al., 1997) .
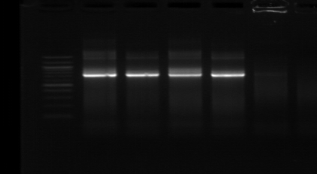
M R
M1 M8 M16
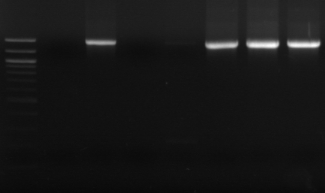
Figure n°10: PCR avec
primers spécifiques pour Lb. sanfranciscensis
M- marqueur 100 pb DNA ladder
(Biolabs)
R : souche de
référence
M1, M8 et M10 : isolats BF-70.
II.3.2. Identification des isolats du
BF-FC :
On a procédé tout d'abord à
l'amplification de la région ITS par les amorces 16S/2-23S/7. Sur 20
isolats, seules 12 souches ont été amplifiées. Les isolats
proviennent du milieu MRS modifié.

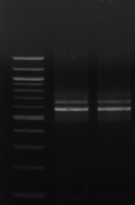
M M1 M2 M3 M4 M7 M9 M10 M11
M12 M15 M M18 M20
Figure n°11: PCR avec
les amorces 16S/2-23S/7 de 12 isolats de BF-FC
On recueille un profil de 3 bandes pour toutes les souches
étudiées (figure n°11). Lb. sanfranciscensis
représenterait encore une fois 100% de la flore lactique de la farine
concassée. On procède aux mêmes démarches que celles
utilisées pour l'étude de la flore lactique de BF-70.
Après une amplification de la région
tRNAala -23S/10 et une restriction enzymatique par les enzymes de
restriction utilisés pour les isolats du BF-70, on observe sur la figure
n°12 pour l'isolat M9 choisi comme exemple que celui-ci comme les autres
isolats de ce levain présente un profil de restriction identique
à celui de Lb. sanfranciscensis ATCC 44322. .
PCR Hinf I
Hind III á Taq I




Figure n°12: PCR
tala/10 et RFLP par Hinf I, Hind III et á Taq I de l'isolat
M9
En outre, pour confirmer que les isolats appartiennent
à l'espèce Lb. sanfranciscensis, une PCR avec les
amorces spécifiques a été faite. La figure n°13
ci-dessous montre le résultat d'amplification des 3 isolats avec les
amorces spécifiques.
R
M1 M9 M20
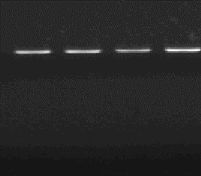
1460 pb
Figure n°13 : PCR avec
primers spécifiques de l'espèce Lb. sanfranciscensis
ATCC 43322
R : souche de référence Lb.
sanfranciscensis ATCC 43322 et les isolats M1,
M9 et M20.
II.3.3.PCR sur colonies:
Les approches classiques pour la caractérisation et
l'identification des bactéries lactiques des levains prennent beaucoup
de travail et du temps.
Dans cette étude, nous avons essayé de
développer la PCR sur colonie comme méthode pour la
détection rapide de la population des bactéries lactiques.
Différents protocoles avec différentes modifications ont
été employés. Les petites colonies ont été
sélectionnées et inoculées dans 5 ul de l'eau
stérile. Ensuite elles seront incubées pendant 5 minutes à
95°C et 2 à 3 ul de la suspension est utilisée pour y
être amplifiée. Des modifications au niveau du mix PCR ont
été faites.
Cette méthode permet de détecter rapidement la
population et le pourcentage de Lb. sanfranciscensis dans
l`échantillon de levains. Dans notre cas, la PCR sur colonies a
confirmé que ces isolats provenant des deux échantillons
appartenaient à l'espèce Lb. sanfranciscensis.
M 1 2 M
3 M 4 5 6
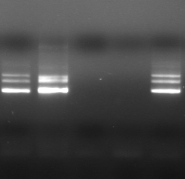
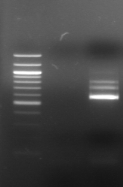


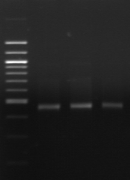
M 7 8 M 9
M 10

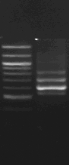

Figure n°14:
Amplification ITS avec la PCR sur colonies
M-Marqueur DNA ladder
(Biolab)
1 à 5 colonies de
BF-70 et 6 à 10 colonies de BF-FC
III. La TTGE
Cette technique est choisie pour étudier le suivi
dynamique des communautés bactériennes. Ainsi, chaque
communauté est représentée par un profil où chaque
bande électrophorétique correspond à une espèce
définie. L'identification des espèces se fait grâce
à un référentiel préalablement établi
à partir de fragments d'ADN de souches pures.
L'extraction d'ADN bactérien est effectuée
directement de la matrice à analyser. Dans notre cas on a choisi
à amplifier la région V6-V8 du 16S rDNA des deux
échantillons.
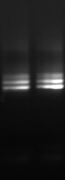
M R
1 2
Figure
n°15 : Amplification de la région V6-V8 du 16S
rDNA.
M: Marqueur 100 pb
DNA ladder (Biolab)
R: souche de
référence Lb. sanfranciscensis ATCC 44322
1- BF-70
2-BF-FC
1 2
3 4

Figure n°16 : TTGE.
1- 3 :Lb. sanfranciscensis ATCC 43322
2- BF-70 4-BF-FC
Le gel ci-dessus montre le profil des échantillons BF. Les
fragments correspondent 100 % à Lb. sanfranciscensis ATCC
44322.
Il faut noter que la TTGE ne permet pas d'identifier les
populations sous-dominantes, qui peuvent avoir un intérêt.
Ce projet avait pour objectifs : d'une part de montrer
l'influence de la matière première mise en oeuvre sur la flore
lactique d'un levain et notamment sur la diversité des espèces de
Lactobacillus et d'autre part de valider les méthodes
moléculaires de caractérisation des flores lactiques. Les
principaux résultats obtenus sont les suivants :
- L'acidité plus élevée obtenue à
partir du levain BF-FC est intéressante.
- Les populations obtenues ne présentent pas de
différences significatives entre les deux levains analysés.
- Les 20 isolats provenant de la farine BF-70
sont des Lactobacillus, 18 d'entre eux sont
hétérofermentaires et 2 homofermentaires. Sur la farine
concassée, les 20 isolats obtenus sont des Lactobacillus
hétérofermentaires.
- L'amplification par PCR avec les amorces
16S/2-23S/7 a mis en évidence la présence de 3 fragments
d'amplification et a conduit à l'identfication présumée de
Lb. sanfranciscensis pour les 16 isolats
hétérofermentaires du BF-70 et les 12 isolats BF-FC .
- L'étape suivante (RFLP) consistant
à générer des profils de restriction en utilisant des
enzymes Hind III, Hinf I et á Taq I a permis d'identifier
spécifiquement Lb. sanfranciscensis et l'enzyme
Taq I s'est révélée être la plus
discriminante.
- Enfin l'utilisation des des primers
spécifiques de Lb. sanfranciscensis (Zapparoli et al.,
1997) a permis de conclure que tous les isolats
hétérofermentaires appartenaient à l'espèce
Lb. sanfranciscensis .
En conclusion, dans les levains de panification
réalisées à partir de la farine concassée, la flore
lactique semble être constituée uniquement de l'espèce
Lb. sanfranciscensis alors que la biodiversité
bactérienne semble être plus importante dans les levains
fabriqués à partir de la farine BF-70 (une faible pourcentage de
lactobacillus homofermentaires).
Les travaux futurs viseront à :
? Compléter l'étude de la biodiversité
des flores lactiques des levains français de panification en
identifiant les espèces de bactéries non encore
identifiées par d'autres méthodes moléculaires telles
que la PCR multiplex et la RAPD.
? Valider les méthodes moléculaires de
caractérisation et de détection des flores
céréaliers par PCR-RFLP et PCR spécifiques sur les
régions intergéniques 16S-23S sur un échantillon plus
large de levains et constituer une banque de profils avec les souches de
référence et les souches sauvages des levains.
? Contribuer à une meilleure connaissance de la
dynamique des écosystèmes par des méthodes
moléculaires cultures indépendantes comme la TTGE.
? Utiliser des souches pour reconstituer des levains pour
obtenir des propriétés technologiques et nutritionnelles
intéressantes.
? Analyser quantitativement les populations
bactériennes par des méthodes moléculaires comme la PCR
quantitative (Real Time PCR).
REFERENCES BIBLIOGRAPHIQUES
|
1- Brevas E., 1991. Mise au point des levains
bactériens pour la panification. Thèse de doctorat.
Université Blaise Pascal (Clermont II). 179 pages.
2- Brochoire G., Netter A., Onno B., Rio I., Roussel
Ph., Stephan C., 1996. Les nouvelles de la boulangerie
pâtisserie, supplément technique.
3- Cai Y., Okada H., Mori H., Benno Y., Nakase T.,
1999. Lactobacillus paralimentarius sp. nov.,
isolated from sourdough. International Journal of Systematic
Bacteriology, 49: 1451-1455.
4- Caillez J-C 2004 La biologie
moléculaire, 1ère édition, Ellipses-238p.
5- Cocolin L., Manzano M., Cantoni C., Comi
G., 2000. Development of a rapid method for the identification of
Lactobacillus ssp. isolated from fermented Italian sausages using a
polymerase chain reaction-temperature gradient gel electrophoresis. Letters in
Applied Microbiology, 30:126-129.
6- Corsetti A., Lavermicocca P., Morea M., Tosti N.,
Gobbeti M., 2001. Phenoypic and molecular identification and
clustering of lactic acid bacteria and yeasts from wheat (species Triticum
durum & Triticum aestivum) sourdoughs of Southern Italy.
International Journal of Food Microbiology, 64: 95-104.
7- Czerny M., Schieberle P., 2002. Important
aroma compounds in freshly ground wholemeal and white wheat
flour-identification and quantitive changes during sourdoughs fermentation.
Journal of Agricultural and Food Chemistry, 50: 6835-6840.
8- Desmazeaud M., 1998. Bactéries
lactiques et qualité des fromages. Laboratoire de recherches
laitières, INRA.
9- DeVuyst L., Schrijvers V., Paramithiotis S., Hoste
B., Vancanneyt M., Swings J., Kalantzopoulos G., Tsakalidou E., Messens W.,
2002. The biodiversity of lactic acid bacteria in Greek traitionnal
wheat sourdoughs is reflected in boh composition and metabolite formation.
Applied & Environnemental Microbiology, 68, N°12:
6059-6069.
10- Di Cagno R., De Angelis M., Lavermicocca P., De
Vincenzi M., Giovannini C. Faccia M., Gobbetti M., 2002. Proteolysis
by sourdough lactic acid bacteria: Effects on wheat flour protein fractions and
gliadin peptides involved in human cereal intolerance. Applied and
Environnemental Microbiology, 68, N°2: 623-633.
11- Duthoit F., Godon J-J., Montel M-C.,
2003. Bacterial community dynamics during production of registered
designation of origin salers cheese as evaluated by 16S rRNA gene single-strand
conformation polymorphism analysis. Applied & Environmental Microbiology,
69, N°7: 3840-3848.
12- Ehrmann M., Müller M., Vogel R.,
2003. Molecular analyses of sourdough reveals Lactobacillus
mindensis sp. nov., .International Journal of Systematic and
Evolutionary Microbiology, 53:7-13.
13- Gobbetti M., Corsetti A., 1997.
Lactobacillus sanfrancisco a key sourdough lactic acid bacterium. Food
Microbiology, 14: 175-187.
14- Gobbetti M., De Angelis M., Corsetti A., Di Cagno
R., 2005 Biochemistry and physiology of sourdough lactic acid
bacteria. Trends in Food Sciences and Technology, 16:57-69.
15- Gobbetti M., Smacchi E., Corsetti A.,
1996. The proteolytic system of Lactobacillus sanfrancisco
CB1: purification and characterization of a proteinase, a dipeptidase, and an
aminopeptidase. Applied and Environmental Microbiology, 62,
N°9: 3220-3226.
16- Gürtler V., Stanisich VA., 1996. New
approaches to typing and identification of bacteria using the 16S-23S rDNA
spacer region. Microbiology, 142: 3-16.
17- Ignatova M. 2005.
Identification moléculaire des levains de panification. Mémoire
de stage. ENITIAA. 41p.
18- Kabadjova P., Dousset X., Le Cam V., Prevost H.,
2002. Differentiation of closely related Carnobacterium food
isolates based on 16S-23S ribosomal DNA Intergenic Spacer Region polymorphism.
Applied and Environmental Microbiology, 68, N°11:
5358-5366.
19- Kitahara M., Sakata S., Benno Y., 2005.
Biodivesity of Lactobacillus sanfranciscensis strains isolated from
five sourdoughs. Letters in Applied Microbiology,
40:353-357.
20- Kline L., Sugihara T.F., 1971.
Microorganisms of the San Francisco sourdough bread process. Applied
Microbiology, 21, N°3: 439-465.
21- Larpent J-P., 1992. La microbiologie de
la fermentation panaire. Agro-alimentaire Information CDIUPA. Université
Blaise Pascal-Clermont II.
22- Lavermicocca P. Valerio F., Visconti A.,
2003. Antifungal activity of phenyllactic acid against molds isolated from
bakery products. Applied and Environmental Microbiology, 69,
N°1: 634-640.
23- Le Jeune C., Lonvaud-Funel A.,
1997. Sequence of DNA 16S/23S spacer region of Leuconostoc
oenos (Oenococcus oeni): application to strain differentiation.
Res. Microbiol., 148:79-86.
24- Meroth C., Walter J., Hertel C., Brandt M., Hammes
W., 2003. Monitoring the bacterial population dynamics in sourdough
fermentation process by using PCR-denaturing gradient gel
electrophoresis. Applied and Environmental Microbiology, 69,
N°1: 475-482.
25- Müller M., Ehrmann M., Vogel R.,
2000. Lactobacillus frumenti sp. nov., a new lactic acid
bacterium isolated from rye-bran fermentations with a long fermentation period.
International journal of systematic and Evolutionary Microbiology,
50: 2127-2133.
26- Müller M., Ehrmann M., Vogel R.,
2000. Multiplex PCR fort he detection of Lactobacillus pontis and two
related species in a sourdough fermentation. Applied and Environmental
Microbiology, 66, N°5: 2113-2116.
27- Müyzer G., Smalla K., 1998.
Application of denaturing gel electrophoresis (DGGE) and temperature gradient
gel electrophoresis (TGGE) in microbial ecology. Antoine Van
Leuwenhoek, 73: 127-141.
28- Nour M., 1998. 16S-23S and 23S-5S
Intergenic spacer regions of lactobacilli: nucleotide sequence, secondary
structure and comparative analysis. Res. Microbiol. 149:
433-448.
29- Oheix N., 2003. Caractérisation
moléculaire des bactéries lactiques des levains de panification.
Mémoire d'ingénieur. ENITIAA. 81p.
30- Onno B., Roussel P., 1994. Technologie et
microbiologie de la panification au levain. Bactéries lactiques Volume
II. Edition Lorica. Pages 293-321.
31- Ogier J-C., Son O., Gruss A., Tailliez P.,
Delacroix-Buchet A., 2002. Identification of the Bacterial Microflora
in Dairy Products by Temporal Temperature Gradient Gel Electrophoresis. Applied
and Environmental Microbiology, 68, N°8: 3691-3701.
32- Rachman C., 2004. Développement
d'outils moléculaires d'identification et d'analyse de la
biodiversité de l'écosystème bactérien du saumon
fumé. Thèse de Doctorat. ENITIAA. 230 p.
33- Rachman C., Kabadjova P., Valcheva R., Prevost H.,
Dousset X., 2004. Identification of Carnobacterium species by
restriction fragment length polymorphism of the 16S-23S rRNA gene intergenic
spacer region and species-specific PCR. Applied and Environmental Microbiology,
70, N°8: 4468-4477.
34- Randazzo C.L., Heilig H., Giudici P., Caggia C.,
2005. Bacterial population in traditionnal sourdough evaluated by
molecular methods. Journal of Applied Microbiology, 99,
251-258.
35- Rohrlich M., 1953. Beiträge zur
kenntnu der biologie und biochemider sauerteiggärung II Mit: Untersuchum
gen uber denbiochemischen Mechanismus der Barkteriellen sauerteiggärung,
z. lebens. Unkers. Forsch. 96, p.24-40.
36- Rollan G., De Angelis M., Gobbetti M., Valdez
G.F, 2005. Proteolytic activity and reduction of gliadin-like
fractions by sourdough lactobacilli. Journal of Applied Microbiology,
99: 1495-1502.
37- Roussel Ph., Chiron H., 2002. Les pains
Français, 1 ère édition,
MAÉ-ERTI-433p.
38- Ruiz A., Poblet M., Mas A., Guillamon J.,
2000. Identification of acetic acid bacteria by RFLP of PCR-amplified
16S and 16S-23S rDNA Intergenic Spacer. International Journal of Systematic and
Evolutionary Microbiology, 50: 1981-1987.
39- Settanni L., Van Sinderen D., Rossi J., Corsetti
A., 2005. Rapid differentiation and in situ detection of 16 sourdough
Lactobacillus species by multiplex PCR. Applied and Environmental
Microbiology, 71, N°6: 3049-3059.
40- Spicher G., 1987. Die Mikroflora des
sauerteiges XXII Mitt: Die in Weizensauerteign Vorkommenden lactobacillen;
z.lebens.Unters.Forsch. 184, p.300-303.
41-Sriranganathan N., Seidler R., Sandine W., Elliker
PR., 1973. Cytological and deoxyribonucleic acid-deoxyribonucleic acid
hybridization studies on Lactobacillus isolates from San Francisco
sourdough. Applied Microbiology, 25, N°3: 461-470.
42- Stiles M., Holzapfel W., 1997. Lactic
acid bacteria of foods and their current taxonomy. Areview J. Food Microbiol,
36: 1-29.
43- Tannock GW., Timisjarvi A., Rodtong S., NG J.,
Munro K., Alatossava T., 1999. Identification of Lactobacillus
isolates from the gastrointestinal tract, silage, and yoghurt by 16S-23S rRNA
gene Intergenic spacer region sequence comparisons. Applied and Environmental
Microbiology, 65, N°9: 4264-4267.
44- Thiele C., Grassl S., Gänzle M.,
2004. Gluten hydrolysis and depolymerization during sourdough
fermentation. Journal of agricultural and food chemistry, 52:
1307-1314.
45- Torriani S., Zapparoli G., Dellaglio F.,
1999. Use of PCR-based methods for rapid differentiation of
Lactobacillus delbrueckii subsp. bulgaricus and L. delbrueckii
subsp.lactis.
Applied and Environmental Microbiology,
Vol.65, N°10: 4351-4356.
46- Valcheva R., Korakli M., Onno B., Prevost H.,
Ivanova I., Ehrmann M., Dousset X., Gänzle M., Vogel R., 2005.
Lactobacillus hammesii sp. nov., isolated from french sourdough.
International Journal of Systematic and Evolutionary
Microbiology, 55: 763-767.
47- Valcheva R., Ferchichi M., Korakli M., Ivanova I.,
Gänzle M., Vogel R., Prévost H., Onno B., Dousset X. 2006.
Lactobacillus nantensis sp. nov., isolated from French wheat
sourdough. International Journal of Systematic and Evolutionary Microbiology,
56: 587-591.
48- Vasquez A., Ahrné S., Pettersson B., Molin
G., 2001. Temporal temperature gradient gel electrophoresis (TTGE) as
a tool for identification of Lactobacillus casei, Lactobacillus
paracasei, Lactobacillus zeae and Lactobacillus
rhamnosus. Letters in Applied Microbiology, 32:
215-219.
49- Vogel R., Knorr R., Muler M., 1999.
Non-dairy lactic fermentations: the cereal world.
50- Wiese B., Strohmar W., Rainey F., Diekmann
H., 1996. Lactobacillus panis sp. nov., from sourdough with a
long fermentation period. International Journal of Systematic
Bacteriology, 46, N°2: 449-453.
51- Zapparoli G., Torriani S., 1997. Rapid
identification and detection of Lactobacillus sanfrancisco in
sourdough by species-specific PCR with 16S rRNA-targeted primers. System. Appl.
Microbiol. 20: 640-644.
52- Zavaleta A., Martinez A., Rodriguez F.,
1996. 16S-23S rDNA intergenic sequences indicate that Leuconostoc
oenos is phylogenetically homogeneous. Microbiology, 142:
2105-2114.
53- Zoetendal E., Akkermans A., De Vos W.,
1998. Temperature gradient gel electrophoresis analysis of 16S rRNA from human
faecal samples reveals stable and host-specific communities of active bacteria.
Applied and Environmental Microbiology, 64, N°10:
3854-3859.
Tableau n°1: La diversité des
flores des levains naturels
2
Tableau n°2: Exemples de quelques
enzymes de restriction et leurs sites de reconnaissances
8
Tableau n°3: Liste des souches de
référence
11
Tableau n°4: Les amorces de
l'étude
13
Tableau n°5: pH et TTA des
échantillons des levains BF
16
Tableau n°6: Dénombrement des
levures et des bactéries lactiques dans les levains BF 16
Tableau n°7: Ratio des isolats
homo/hétérofermentaires dans les levains
17
Figure n°1 : Positions des
régions conservées de l'opéron ribosomique 16S-23S chez
Escherichia coli
6
Figure n°2 : Organisation de la
région intergénique 16S-23S de l'ARNr chez les bactéries
lactiques
7
Figure n°3 : La technique TTGE
9
Figure n°4 : Formation d'un brin
d'ADN ramifié grâce à la présence de GC clamp
9
Figure n°5 : Schéma
générale de l'étude
10
Figure n°6 : Le marqueur 100pb DNA
Ladder-Biolabs
15
Figure n°7 : PCR avec les amorces
16S/2-23S/7 de 16 isolats de BF-70 18
Figure n°8 : Amplification par les
amorces tRNAala -23S/10 (souches : 1,8 et 16)
19
Figure n°9: RFLP par Hind III, Hinf I et
á Taq I
19
Figure n°10 : PCR avec primers
spécifiques pour Lb. sanfranciscensis
20
Figure n°11 : PCR avec les amorces
16S/2-23S/7 de 12 isolats de BF-FC 21
Figure n°12 : PCR
tala/10 et RFLP par Hinf I, Hind III et á Taq I de la souche
M9 21
Figure n°13 : PCR avec primers
spécifiques de l'espèce Lb. sanfranciscensis
ATCC 43322
22
Figure n°14 : Amplification ITS
avec la PCR sur colonies
23
| 


