Il s'est agi d'une étude
observationnelle à visée descriptive et analytique,
privilégiant une approche pluridisciplinaire qui associe
l'épidémiologie et la biologie moléculaire
.3.1.1. Type et période d'étude
Cette étude a été réalisée
entre Avril 2022 et Septembre 2022 au sein de l'Unité
d'Epidémiologie, d'Evolution et de Résistance Parasitaire
(UNEEREP) du Centre Interdisciplinaire de Recherche Médicale de
Franceville (CIRMF). Les échantillons analysés provenaient d'un
recueil effectué entre 2013 et 2014, dans la région de
Lastourville, incluant le centre médical de Lastourville, la ville de
Lastourville, les villages de Matsatsa, Mikouyi, Malende et Mana-Mana, dans le
département de Mulundu (province de l'Ogooué-Lolo, Sud-Est du
Gabon).
3.1.2. Site de l'étude
Les prélèvements sanguins pour les patients
symptomatiques ont été réalisés sur des enfants
reçus en consultation au Centre Médical de Lastourville. Quant
aux enfants asymptomatiques, leurs prélèvements ont
été réalisés dans les écoles au sein des
différents villages inclus dans l'étude.
Le diagnostic microscopique et immunochromatographique (TDR)
de l'infection plasmodiale a été réalisé sur les
sites de récoltes d'échantillons tandis que les analyses
moléculaires ont été réalisées au Centre
Interdisciplinaire de Recherches Médicales de Franceville (CIRMF).
3.1.3. Population d'étude
Les sujets inclus dans cette étude étaient des
enfants âgés de 6 mois à 15 ans, présentant un
syndrome fébrile ou non, recrutés dans les sites
mentionnés ci-dessus.
3.1.4. Critères d'inclusion et de non
inclusion
Pour les patients fébriles, les critères
d'inclusion étaient les suivants :avoir consulté le service de
pédiatrie externe pendant la période de l'étude , avoir
une température corporelle supérieure ou égale à
37,5°C ou une histoire de fièvre durant les 24 heures
précédents, être âgé de 6 à 180 mois ,
avoir un test diagnostique rapide (TDR) positif, et présenter au moins
un critère spécifique au paludisme selon les recommandations de
l'OMS (notamment la fièvre). Pour les asymptomatiques, les individus ne
devaient présenter aucun signe de fièvre durant les 7 jours
précédent le prélèvement. Pour les mineurs le
consentement éclairé a été signé par un
tuteur. Les enfants dont les tuteurs n'ont pas donné leur consentement
n'ont pas été inclus dans l'étude.
3.2. Analyses Biologiques
3.2.1. Diagnostic de
l'infection plasmodiale
3.2.1.1. Le prélèvement :
Le prélèvement sanguin a été
réalisé au pli du coude dans un tube contenant de l'EDTA ou par
piqure franche au bout du doigt (pulpe du doigt). Après
réalisation des différents tests de diagnostics du paludisme, les
prélèvements ont été séparés par
centrifugation à 1500 tours/ minutes afin d'obtenir le culot globulaire
et le plasma. Les culots globulaires ont été
transférés dans des tubes Eppendorff puis conservés
à -20°C, pour l'extraction de d'ADN.
Le diagnostic du paludisme a été
réalisé par la technique de la Goutte épaisse, le test de
diagnostic rapide du paludisme (TDR, kit optimal-It). Le diagnostic des
différentes espèces plasmodiales a été
réalisé par analyse moléculaire.
3.2.1.2. Diagnostic par TDR
Le test de diagnostic rapide du paludisme (TDR), est un test
immuno-chromatographique qui utilise des anticorps monoclonaux dirigés
contre la lactate déshydrogénase spécifique à
P. falciparum (pfLDH) et une LDH commune aux espèces de
Plasmodium (pan-LDH) [22]. Ce test contient un dispositif prêt
à l'emploi, constitué d'une cassette (contenant deux puits, puits
lavage et puis conjugué) et d'une bandelette. La réalisation de
celui-ci consiste à mettre une goutte de tampon dans le premier puits
(puits conjugué), 4 gouttes dans le deuxième puits (puits
lavage), qu'on laisse reposer 1 minute. Dix microlitres (10 ìl) de sang
ont été ensuite prélevé à l'aide de la
pipette et déposé dans le premier puits. Après
homogénéisation, le produit a été laissé au
repos pendant 1 minute, puis, le support de la bandelette a été
retiré et l'extrémité de la bandelette été
mise dans le puits conjugué. Après 10 minutes d'incubation, la
bandelette a ensuite été transférée dans le
puits-lavage, et a été à nouveau incubée pendant 10
minutes. Une fois le lavage terminé, la bandelette est mise dans la
pièce plastique transparente pour effectuer la lecture. Le test est
validé lorsque la bande contrôle apparait et
l'interprétation est fonction du nombre de bande qui apparait sur la
bandelette (voir Figure 6).
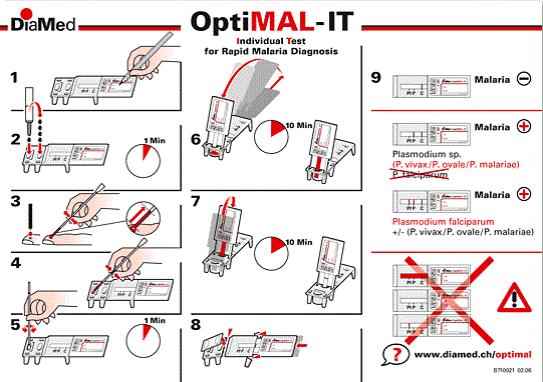
Figure 6:
Représentation schématique de la réalisation d'un TDR(
kit-optimal-it)
(1) -identification de bandelette, (2) - remplissage des puits
avec le tampon de lyse, (3) -prélèvement sanguin à la
pulpe du doigt, (4) - dépôt du sang dans le tampon, (5)
-homogénéisation et lyse, (6) - migration chromatographique (10
minutes) (7) - lavage 10 minutes, (8) mise de la bandelette dans la
pièce plastique, (9) - interprétation des résultats.
3.1.2.3. Diagnostic par microscopie: Goutte
épaisse
La goutte épaisse permet la mise en évidence des
parasites dans le sang mais aussi leur quantification. Elle a été
réalisée selon la méthode de Lambaréné qui
consiste à étaler sur une lame porte-objet propre et
dégraissée 10ìl de sang sur une surface de 18×10
mm2. Après étalement, la lame est séchée
puis colorée pendant 25 minutes au Giemsa à 20%. La lame est
ensuite lue au microscope optique à l'objectif ×100 en utilisant de
l'huile à immersion. La parasitémie a été
déterminée en nombre de formes asexuées de
Plasmodium et calculée en utilisant la formule suivante :
Parasitémie = X × 500 parasites / ìl de sang. X= moyenne de
parasites obtenus à partir de 10 champs microscopiques, 500= facteur
correcteur (varie en fonction du microscope).
3.2. Analyses moléculaires
3.2.1. L'extraction d'ADN :
L'extraction d'ADN par le kit Blood DNA
E.Z.N.A® est basée sur la chromatographie
d'affinité, elle consiste à mélanger, 250ìL du
culot globulaire, 25ìL d'OB protéase (20mg/ml), 250ìL de
tampon de lyse (BL) dans un tube Eppendorff. Le mélange est
incubé à 65°C pendant 10 minutes au minimum (on
homogénéise toutes les 3 minutes). A la fin de l'incubation,
260ìL d'éthanol absolu sont ajoutés, puis le
mélange est homogénéisé. Une colonne d'extraction
est ensuite équilibrée avec 100ìL de tampon
d'équilibration pendant 4 minutes à température ambiante
et centrifugée brièvement pendant 20 secondes. Le lysat est
transféré dans la colonne puis centrifugé pendant une
minute à 10000g. La colonne est transférée dans un nouveau
tube collecteur et 500ìL de tampon HB sont ajoutés puis
centrifugée pendant 1 minute à 10000g. Après
centrifugation, la colonne est à nouveau transférée sur un
tube collecteur et lavée deux fois avec 700ìL de DNA Wash buffer
reconstitué avec de l'éthanol à 70%. Elle est ensuite
centrifugée pendant une minute à 10000g, puis
séchée par centrifugation pendant 2 minutes à 13000g. La
colonne est ensuite placée dans un tube Eppendorff stérile de
1,5ml. Pour l'élution de l'ADN, 90ìL d'eau distillé
préchauffée à 65°C, ont été
déposé sur la colonne, que l'on laisse reposer 5 minutes à
température ambiante. La colonne est ensuite centrifugée pendant
une minute à 10000g, pour l'élution de l'ADN, l'élution
est répétée une seconde fois. L'ADN recueilli dans le tube
Eppendorff est conservé à -20°C.
| 


