I.1.2.2. Les éléments SINEs :
Ils sont des rétroposons non autonomes
dérivés principalement des ARN de transfert ou de l'ARN
cytoplasmique 7SL (Figure 2), le SINE le plus abondant chez l'homme
est appelé Alu, parce qu'il contient un site de clivage pour
l'enzyme de restriction Alu1. Un élément Alu
complet est d'environ 200 nucléotides, et contient deux
répétitions d'environ 120pb encadrant une séquence de
60pb. Le mécanisme de déplacement des SINEs, n'est pas
encore élucidé : dans la mesure où ils ne codent pas pour
les fonctions nécessaires à leur rétrotransposition, les
SINEs pourraient utiliser la Reverse Transcriptase des
éléments LINEs. Ces « ADN égoïstes
» sont avérés des outils très efficaces en
systématique moléculaire : en effet, l'insertion d'un SINE
à un site donné est un événement unique et non
réversible à l'échelle des génomes (Huchon et
al., 2002 ; Renault et al., 1997 ; in Griffiths et
al., 2004).
I.1.2.3. Les RTEs :
Ils sont des rétroposons autonomes d'environ 3,3 kb et
possèdent de larges répétitions d'une centaine de paires
de bases. Contrairement aux autres rétroposons, l'élément
RTE ne code que pour l'ORF2 (Figure 2), ce qui en fait le plus petit
rétroposon autonome connu actuellement (In Tempel, 2007).
I.2. Les éléments de la Classe II: Les
Transposons
Ces éléments transposent d'un site chromosomique
à un autre grâce à l'activité d'une transposase, une
enzyme codée par l'élément sur un ou plusieurs ORF
(Anxolabéhère et al, 2000). Ils sont Essentiellement
caractérisés par la présence des ITR (Inverted Terminal
Repeat ou répétition terminal inversé) en orientation
inverse qui flanquent les transposons à chaque extrémité.
(Renault et al, 1997).
Au cours des 30 dernières années, le nombre des
transposons chez les procaryotes et les eucaryotes identifiés n'a
cessé à croître, avec une grande diversité dans leur
structure et une remarquable conservation des mécanismes qui assurent
leur mobilité (Merlin et Toussaint, 1999).
I.2.1. Structure et diversité des transposons
procaryotes:
La classification des transposons procaryotes se repose sur des
critères fonctionnels et structuraux. De ce fait, ils sont
regroupés en deux classes principales:
· Les séquences d'insertions (IS)
· Les transposons composites (Tn) I.2.1.1.
Les séquences d'insertions: (IS)
La première séquence d'insertion était
découverte chez E-coli pendant les années 60 à
l'occasion d'une mutation qui apparaissait dans les gènes constituant
l'opéron lactose grâce à l'insertion de cette
séquence.
Les séquences d'insertions sont les
éléments transposables les plus simples, de petite taille
(généralement inférieure à 2500pb) (Merlin et
Toussaint, 1999) et bordées à
chaque extrémité par des ITR (< 50pb) (In
Griffiths et al., 2004) indispensables pour le phénomène
de transposition et qui sont reconnus spécifiquement par la transposase
( figure 3) (In Singer, Berg, 1992).
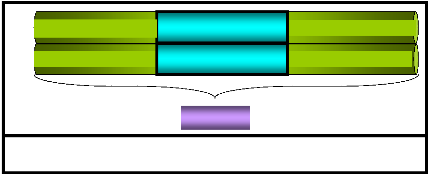
Figure 3 : La structure d'une séquence
d'insertion (IS1) (d'après Singer, Berg.,
1992).
GGTGATGCTGCCAACTTACTGAT
CCACTACGACGGTTGAATGACTA
722pb
ORF
ORF
ATCAATAAGTTGGAGTCATTACC
TAGTTATTCAACCTCAGTAATGG
Selon les différences dans leurs séquences, les
IS ont été cataloguées en 17 familles dont les
membres des 12 entre elles contiennent un seul ORF qui code pour la
transposase, à partir de ce même cadre ouvert de lecture, les
IS de certaines familles codent pour une autre protéine, une
version tronquée de la transposase mais douée d'une
activité régulatrice pour la transposition. Pour les membres de
la famille IS3, la synthèse de la transposase (ORF AB)
résulte d'un déphasage programmé de la traduction entre
deux cadres de lecture ouvert qui codent pour une protéine
régulatrice de la transposition (ORF A) et une protéine de
fonction inconnue (ORF B) (Merlin et Toussaint., 1999). La séquence du
site d'insertion varie pour une même IS mais pas sa longueur,
aussi, certains éléments manifestent une
spécificité pour une séquence en palindrome (par exemple
elle est 5'-CTAG pour IS5), lors de l'intégration d'une IS une
duplication du site cible se produit, ce qui conduit à en trouver une
copie à chaque extrémité de l'élément
(Merlin et Toussaint., 1999; In Singer et Berg., 1992; Renault et al.,
1997) ( figure 3.1).
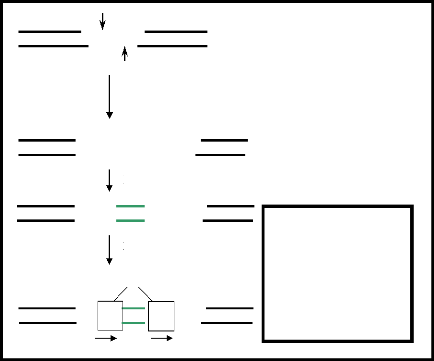
AGGTAAGGTA
AGG TCCATTCC
TAAGGTAG
ATC
TCCATTCCATC
La Transposase coupe le site cible
L'insertion de l'élément transposable
Figure 3.1 : La
duplication du site cible lors de l'insertion d'un
élément transposable par la coupure cohésive
effectuée par la transposase. (d'après Griffiths et al.,
2004)
AGG TCCATTCC
TAAGGTAG
ATC
AGGTAAGG
TCCATTCC
TAAGGTAG ATTCCATC
L'hôte répare les brèches
Un ITR de 5 pb flanque l'élément
| 


