I.1.1.b2.2. La famille des LARDs :
Les LARDs identifiés dans l'orge et d'autre
membre de Triticaeae ont des LTRs de 4.5kb et un domaine interne
non-codant de 3.5kb. Le mécanisme de déplacement de ces
éléments est mal-connu, mais en se basant sur les similitudes des
séquences, il semble que ces éléments se mobilisent sous
l'action d'autres éléments tels que les métavirus
Erika-1 de Triticum monococcum et RIRE3 du riz
(Figure1.1) (Havecker et al., 2004).
I.1.1.c. Cycle de vie d'un rétrotransposon :
Les rétrotransposons à LTR suivent un cycle de
vie analogue à celui des rétrovirus décrit par Echalier en
1989 (In Rivière, 2000), la transcription de l'élément en
ARNm débute dans la région 5'-R à la région R-3'des
LTR, de sorte que l'ARN bicistronique (génère deux
protéines) soit une version tronquée de l'ADN en raison de
l'absence des régions U3 en 5 `et U5 en 3'. Par la suite, cet ARN est
traduit aux différentes protéines des gènes pol
et gag (plus des protéines env dans le cas des
Errantivirus), la polyprotéine pol est clivée par la
protéase « PR », en INT, RH, PR et RT, les protéines
gag se polymérisent pour former les particules viraux, ensuite
l'ARN est assemblé dans ces particules où commence la
transcription inverse par le couple RT-RH, la reverse transcriptase
synthétise l'ADN en utilisant l'ARN comme matrice avec un taux d'erreur
de (2.5×10-5 erreurs/nucléotide/cycle) et la
Ribonucléase H lyse l'ARN matricielle après l'achèvement
de la transcription inverse. Enfin, l'ADN double brin synthétisé
est couplé à l'intégrase INT (Sabot et Schulman, 2006)
(qui est caractérisée par un motif catalytique de type DDE
analogue à celui des enzymes des éléments
mariner) sur les LTRs, ce complexe migre dans le noyau dont INT assure
l'intégration du rétrotransposon dans un nouvel site hôte
(Figure1.2) (Kapitonov et Jurka, 2006 ; Sabot et Shl 2006)

PR
Figure 1.2: Cycle de vie d'un
rétrotransposon. (D'après Sabot et Schulman, 2006)
I.1.2. Les rétrotransposons sans LTR : ou
rétroposons
Ces éléments ont également une phase ARN
dans leur cycle de rétrotransposition, ils ne possèdent pas des
LTR et sont subdivisés en deux familles selon leur autonomité,
les éléments autonomes « LINEs (Long
Interspersed Nuclear Elements) », et les éléments non
autonomes « SINEs (Short Interspersed Nuclear Elements)
», actuellement une nouvelle famille appelée RTEs
(RetroTransposable Elements) est connue (Figure 2) (In Tempel,
2007).
I.1.2.1. Les LINEs :
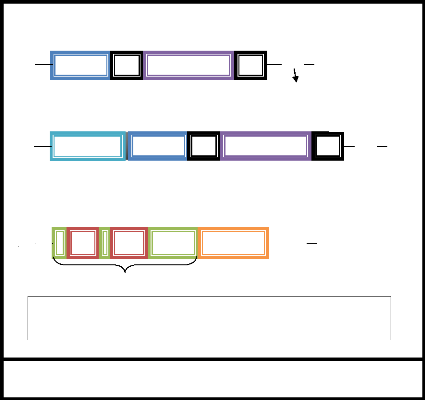
Figure 2 : Organisation structurelle des
rétroposons. (In Tempel, 2007 ; Huchon et
al., 2002)
5'
5'
5
Légende : ENDO : endonucléase, RT : Reverse
Transcriptase,
An : queue poly A ; A.B : sites de fixation à
l'ARN polymérase Ø
ENDO
ORF1
A
Domaine ARNt
B
ENDO
ORF2
RT
ORF2
O
RT
Queue poly A
An 3'
An
SINEs
RTEs
3'
LINEs
An
3'
Les éléments autonomes de type LINEs sont
représentés majoritairement dans les génomes des
mammifères y compris l'homme, ils constituent 40% du génome
humain, et il existe plusieurs éléments
LINEs : L1, L2, L3 le premier étant le seul
élément actif décrit dans le génome humain. Un
élément L1 complet est une séquence d'environ
6kpb, les éléments incomplets présentent des troncations
en 5', de réarrangement interne ou de mutations. Un TSD de 9-19pb soit
présent, ou non selon les espèces, à chaque
extrémité de l'élément. L1 possède
en 3' une séquence riche en A (poly A), il se trouve des
séquences allant de séries d'A presque ininterrompues
jusqu'à une répétition en tandem de la séquence
TAAA ou de séquences similaires (Gilbert, non publié ; in Singer
et Berg, 1992), il existe aussi deux ORF, l'ORF1 code une protéine de
liaison à l'ARN de 40kDa, qui se lie avec l'ARN du L1 dans le
cytoplasme pour former un complexe ribonucléoprotéine « RNPs
(RiboNucleoprotein Particles) >>, tandis que l'ORF2 code une
protéine multifonctionnelle présentant des activités
Endonucléase et Reverse Transcriptase (RT), l'endonucléase
génère des trous dans l'ADN chromosomique, et la RT transcrit
inversement l'ARNm en ADN en utilisant le résidu 3' hydroxyle
libéré comme amorce, il semble que la transposition d'un
élément L1 s'effectue par un processus appelé
« TPRT (Target-Primed Reverse Transcription) >> (In Zhan, 2007)
c'està-dire que l'endonucléase réalise une coupure simple
brin dans l'ADN de l'hôte, et la queue poly A de l'ARN de
rétroposon se fixe à une région poly T. D'une façon
plus générale la rétrotransposition du L1 peut se
résumer dans deux étapes. La première est la transcription
et la traduction d'un élément L1 actif pour former le
RNP. La deuxième est la transcription inverse et l'intégration
qui sont concomitantes à un nouveau site chromosomique (Figue2.1)
(Gilbert, non publié).
Figure 2.1 : Modèle de
rétrotransposition de l'élément L1 (d'après
Gilbert,
non publié)
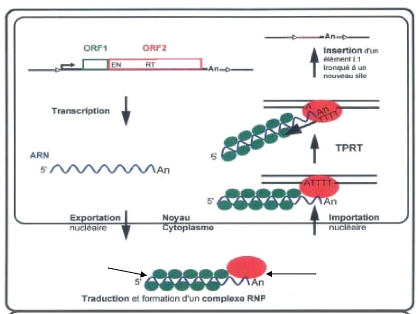
Protéine de liaison
à l'ARN La protéine
multifonctionnelle
| 


