I.2.1.2. les transposons composites: "Tn"
Ils sont des entités avec des structures complexes
(peuvent compter jusqu'à plusieurs milliers de pair de base), ils
contiennent une variété des gènes encadrés par deux
IS en direction directe ou inverse (figure 3.2) (In Watson et
al., 1994; In Singer et Berg, 1992), très souvent seule une des
deux séquences d'insertion code une transposase fonctionnelle, tandis
que l'autre code majoritairement pour un régulateur de la transposition,
le choix de transposition d'un Tn entier ou d'une seul IS se
fait en fonction de la taille de la séquence située entre les
deux IS c'est-à-dire que plus un Tn est court plus qu'il a
tendance à être mobilisé en entier (Merlin et Toussaint,
1999; Renault et al., 1997). Le segment d'ADN bordé par les
deux IS peut coder pour n'importe quelle fonction comme la
résistance aux antibiotiques (kanamycine chez Tn5,
tétracycline chez
Tn10), ou une fonction métabolique (catabolisme
du citrate chez Tn3411) (Tableau 1) (Merlin et Toussaint, 1999).

Figure 3.2: La structure du transposon Tn10,
les IS sont orientées en sens inverse et
forme des ITR
(d'après GRIFFITHS et al., 2004).
IS10 IS10
Résistance à la
tétracycline
Tranposon Tn10
Tableau 1: Transposons caractéristiques
d'E.coli (Tableau d'après Singer et Berg,
1992)
|
Transposon
|
Taille (pb)
|
Extrémités IS
|
Orientation des IS
|
Réplique terminale inverse
(pb)
|
Duplication du site
cible
|
Gènes
portés
|
|
Tn3
|
4957
|
Aucun
|
|
38
|
5
|
Ampr
|
|
Tn9
|
2638
|
IS1
|
Identique
|
23
|
9
|
Camr
|
|
Tn10
|
9300
|
IS10
|
Inverse
|
1329
|
9
|
tetr
|
Ces transposons peuvent passer du chromosome bactérien
au génome d'un phage ou dans un plasmide conjugatif. Pour ces raisons,
ces transposons peuvent être transmis à d'autres bactéries.
Ce type de transposon est une cause naturelle d'acquisition de
résistance aux antibiotiques pour les bactéries (in Halaimia
Toumi, 2006).
I.2.1.3. Les mécanismes de transposition: Deux
modèles de transposition des IS et donc des Tn ont été
observés
v' La transposition réplicative:
(appelé aussi la co-intégration ou
copier-coller)
Ce processus implique la formation d'une structure
moléculaire de type "crossing-over" appelée
co-intégrat. Cette structure résulte de la fusion entre un
réplicon (molécule circulaire capable de se répliquer)
donneur et un réplicon récepteur avec une copie du transposon
à chacune des deux jonctions entre ces deux molécules (In Singer
et Berg, 1992; Renault et al., 1997), généralement cette
structure ne persiste pas et une recombinaison entre les deux copies du
transposons résout le co-intégrat. La transposase produit une
coupure à un site bien précis de chaque côté du
transposon ainsi qu'au site d'insertion, ensuite ces extrémités
sont liées et une réplication se produit en utilisant les
fonctions réplicative de la cellule hôte pour former le
co-intégrat qui est détaché par une résolvase
codée par le transposon et qui agit au niveau du site res
également porté par le transposon. Dans certains cas, une
intégrase achève la résolution du co-intégrat en
agissant sur un site appelé att (figure 3.3) (Merlin et
Toussaint, 1999; In Singer et Berg, 1992; Renault et al., 1997).
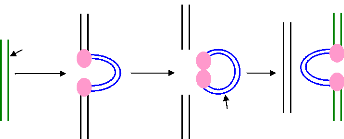
v' La transposition conservative: "non
réplicative" "couper-coller" cut-paste":
Ce modèle correspond à la simple insertion, la
transposase se fixe aux ITRs pour former une structure compacte appelée
complexe synaptique ou transpososome, qui rapproche les ITRs, ensuite une
coupure double brin est effectuée par la transposase conduisant à
la formation de l'intermédiaire de transposition (une molécule de
transposase fortement associée au transposon). En présence d'ions
calcium ou magnésium, la transposase coupe au site cible d'une
façon décalée et une structure branchée de type
crossing-over est formée entre les brins d'ADN du transposons
donneur et la séquence cible. La réparation des
extrémités décalées conduit à la duplication
de la séquence cible. Cette transposition se distingue de la
précédente par le fait que l'intégration du transposon
dans le site cible se fait sans le passage par une étape de
réplication (Figure 3.4) (In Singer et Berg, 1992; Renault et
al., 1997; In Watson et al., 2004).

31
Figure 3.3: la transposition
réplicative
Transposon
Résolution par la résolvase ou
l'intégrase
La transposase coupe le réplicon donneur
Fusion entre les deux réplicons et
réplication
du transposon
Le réplicon donneur


1
2
3
4
Transpososome
7 8
5
6
Intermédiaire de
transposition
9


Figure 3.4: Le mécanisme
général de la transposition conservative: 1: la transposase, 2:
l'ADN donneur, 3: le transposon, 4: liaison de la Tnp(pour transposase) aux ITR
du transposon, 5: l'ADN récepteur, 6: rapprochement des ITRs pour former
le transpososome, 7: détachement du transposon de son hôte en se
liant à la transposase et la formation de l'intérmédiaire
de transposition, 8: liaison de la transposase à la séquence
cible de la molécule réceptrice, 9: intégration du
transposon dans son nouveau hôte.
1' La transposase et son mode d'action:
Les transposases sont peu conservées dans leur
structure primaire, mais contiennent très souvent un motif
conservé D,DxE/D où le résidu glutamate (E) ou aspartate
(D) est généralement séparé des deux premiers
résidus aspartate par un nombre connu d'acide aminé
fréquemment conservé pour les membres d'une même famille,
ce motif est impliqué dans la liaison de deux ions métalliques
bivalents (magnésium ou
calcium) et fait partie du site catalytique de l'enzyme, le mode
d'action de la transposase est généralement comme suit:
1' Les ions bivalents piégés par le motif
D,D-E/D, interagit avec les extrémités de l'élément
et fragilise un pont phosphodiester qui devient plus sensible à une
attaque nucléophile par une molécule d'eau qui libère des
extrémités 3'-OH .
1' Une seconde attaque se lance contre les ponts phosphodiester
de l'ADN cible y libérant des extrémités 5'-P
sortantes.
1' Selon que la transposase a clivé ou non
l'extrémité 5' du transposon, la transposition sera conservative
ou réplicative. Dans le premier cas, les deux brins du transposon se
désolidarisent simultanément du site donneur et la
synthèse d'ADN s'arrête au niveau du transposon qui n'est pas
répliqué, en revanche lorsque la transposition est
réplicative, le second brin du transposon n'est pas clivé, la
synthèse à l'extrémité 3'-OH de la cible se
poursuit à travers le transposon et le duplique, en engendrant un
co-intégrat (Shao et Tu, 2001; Merlin et Toussaint, 1999).
I.2.2. Les transposons eucaryotes:
Après la découverte des éléments
de contrôle par Barbara McClintock, plusieurs transposons eucaryotes ont
été identifiés chez divers organismes comme la drosophile,
les levures, les nématodes et divers vertébrés y compris
l'homme. Comme le cas des séquences d'insertion bactériennes, les
transposons eucaryotes contiennent des ITRs qui flanquent la région
codante pour la transposase qui catalyse la transposition d'une manière
réplicative ou conservative. Ils sont divisés en plusieurs
familles individualisées par des similarités de séquence
ainsi que dans la séquence du site cible d'insertion (In Lodish et
al., 2004). Ils sont également divisés en
éléments autonomes pour leur transposition et nonautonomes qui se
déplacent via une transposase d'un élément autonome qui
appartient à la même famille, les familles les plus connues
sont:
1. Hat (h:
hobo de Drosophila melanogaster, a:
Ac du maïs, t: Tam3 du
antirrhnium majus )
2. Le groupe des éléments
PiggyBac.
3. Le groupe des éléments
P.
4. Le groupe des
MITEs/PIF/Harbinger
5. Le groupe EN/Spm
6. L'élément helitron
7. Le groupe Mutator (Mu)
8. Le groupe de la super famille
IS630/Tc1/maT/mariner
9. La famille Polintons(Mavericks)
10. La famille des éléments
Merlins
11. La Super famille des Transib
| 


