I.2.2.8. Le groupe des éléments de la super
famille IS630/Tc1/maT/mariner :
La taille des éléments de cette super famille
peut atteindre des milliers de paires de bases, les ITRs sont de 19 à
69pb, la transposase est codée par un gène
généralement sans intron, leur site d'insertion est le
dinucléotide TA (.Halaimia.Toumi et al., 2004). Les
éléments de la famille IS630 qui existent chez les
bactéries code une transposase qui contient une triade catalytique de
type DDXE (X indique une distance variable). Les TLEs (Tc1
Like-Elements) identifiés dans les champignons, les
vertébrés et les invertébrés portent le motif D,
D34E. Les MLEs (mariner like-elements) présente la triade D,
D34D à l'exception de Soymar1 qui est identifié dans le
soja ainsi que dans le riz et les nématodes et qui est
caractérisé par le motif D, D39D (Shao et Tu, 2001). La famille
des maTs est une famille intermédiaire entre les MLEs
et TLEs (<< ma >> pour mariner et
<< T >> pour Tc1) et qui a été
définie par Claudianos et al. (2002) possède le motif
D,D37D (In Halaimia Toumi, 2006). D'après les analyses des domaines
catalytiques un autre élément appelé pogo
décrit chez Drosophila melanogaster et contient le motif D,D30D
peut être classé parmi les membres de la famille
IS630/Tc1/maT/mariner (Shao et Tu, 2001).
(a)
Tc1
(D,D34E)
maT
(D,D37D)
ITmD41D
(D,D41D)
mariner
(D,D34D)
Plantes
(D,D39D)
Bactéries/ Champignons
(D,DxE/D) (x: distance
variable)
(b)
41
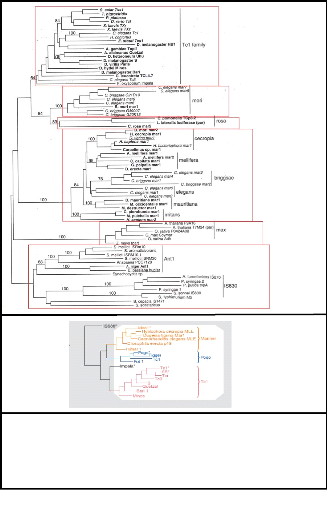
Figure 4.3 : Les différentes familles de
la superfamille
IS630/Tc1/ITmD41D/maT/mariner/Pogo/Ant1.
(a) : Positionnement des familles
IS630/Tc1/ITmD41D/maT/mariner/Ant1
(c) : Positionnement de la famille Pogo par rapport
à mariner et Tc1. (in Halaimia Toumi, 2006)
I.2.2.9. La famille Polintons(Mavericks) :
Les éléments de cette famille ont
été découverts plus récemment (2006), ils sont les
transposons les plus complexes identifiés jusqu'ici. Les polintons,
ou aussi les mavericks, sont des géants
éléments d'environ 10-20 kb qui peuvent coder au moins pour 10
protéines différentes dont une ADN polymérase B (POLB),
une intégrase rétrovirale (Int), une protéase
adénovirale (PR), et une ATPase. Ils ont un TSD de 6pb qui flanque des
ITRs longs contenant des terminaisons 5'-AG et TC-3' (Figure 4.5). Kapitonov et
Jurka (2006), Pritham et al. (2006) ont développé un
modèle de transposition unique à ces éléments,
selon ces auteurs la molécule d'ADN de polinton/mavericks
excisée par l'intégrase, au cours de la réplication du
génome de son hôte, sert comme une matrice pour une
synthèse extra-chromosomique du brin d'ADN complémentaire par
l'ADN polymérase B, la molécule d'ADN double brin s'insère
dans le génome sous l'action de l'intégrase. Ces
éléments sont largement répandus dans les génomes
eucaryotes comme les génomes des insectes, des poissons et aussi des
oiseaux (Kapitonov et Jurka, 2006 ; Pritham et al., 2006)
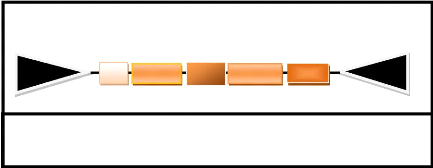
ITR Int ORFs Pro ATPase POL ITR
Figure 4.4 : Structure des
éléments de la famille Polintons/Mavericks
(D'après Tempel, 2007)
| 


