1.2.2 Analyse du profil génomique
généré par XhoI
Après l'analyse du profil de restriction de l'enzyme
XhoI (Figure 10, Annexe 3), 16 bandes au niveau du phage p
M11, 15 bandes pour le phage p T4, 14 bandes pour les phages p M3 et p M7 et
enfin 13 bandes pour le phage p A5a ont été observées.
Cependant aucune bande n'a été observée au niveau du phage
Ebrios. Le profil PFGE des deux enzymes XbaI et XhoI a montré que les
phages p T4, p A5a, p M3, p M7et p M11 ont tous une taille supérieure
à 164 Kb.
Les pulsotypes générés par XbaI
présentant un dendrogramme sur la Figure 9 ont
montré entre eux des similarités comprises entre 73 et 99 % ;
ceux générés par XhoI présentés sur la
Figure 11 ont présenté entre eux des
similarités comprises entre 40 et 96 %. Cela implique que les profils de
restrictions des phages sélectionnés sont différents entre
eux.
40

Résultats et Discussion
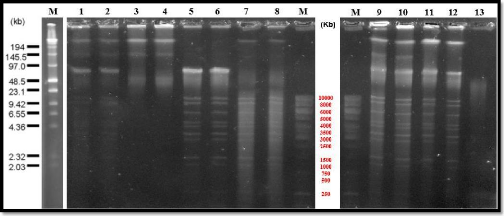
Figure 10 : Électrophorèse en
champ pulsé des génomes de bactériophages après
digestion par XhoI
M1 : marqueur de poids moléculaire
spécifique du PFGE (0.13-194 kb) et M2 : marqueur de
poids moléculaire 1 kb ; Puits 1: (p T4c ; Puits
2: (p T4c ; Puits 3: (p T4 b ; Puits
4: (p T4 b ; Puits 5: (p A5a ; Puits
6: (p A5a ; Puits 7: (p M3 ; Puits
8: (p M3 ; Puits 9: (p M7 ; Puits
10: (p M7 ; Puits 11: (p M11 ; Puits
12: (p M11 ; Puits 13: (p Ebrios.

Figure 11 : Dendrogramme
généré par le logiciel Bionumerics® avec l'enzyme
XhoI
Elle montre la distance calculée par l'indice de
similarité de Jaccard, des profils PFGE des génomes de phages
fragmentés par XhoI. Le degré de similarité (%) est
indiqué.
41

Résultats et Discussion
| 


