Chapitre III : Résultats et discussion
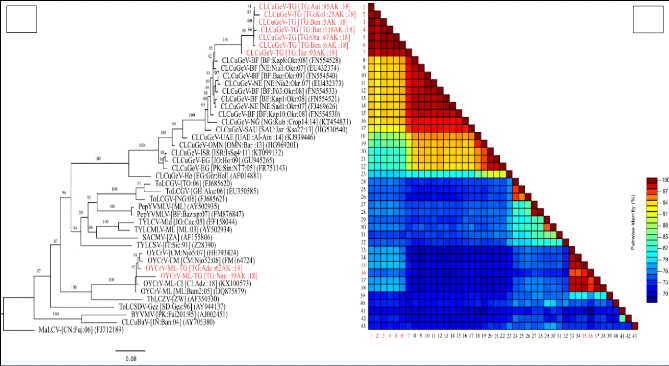
A
B
Figure16: (A) : Arbre phylogénique
généré à partir des séquences de CP des
CLCuGeV, OYCrV du Togo et d'autres Begomovirus existant dans le GenBank.
L'arbre a été construit selon la méthode dite du
«Maximum likelihood» avec 1000 répétitions de bootstrap
dont les pourcentages sont indiqués aux différents noeuds.
(B) : Matrice d'identité des 9 isolats par rapport aux
autres isolats recueillis sur GenBank.
44
Chapitre III : Résultats et discussion
II. Discussion
Le gombo (Abelmoschus esculentus (L.) Moench), de par
son importance nutritionnelle et économique constitue l'un des
légumes les plus cultivés en Afrique. Au Togo il est
cultivé durant toute l'année sur presque toute l'étendue
du territoire. Cependant, force est de constater que la production de ce
légume en Afrique de façon générale et
particulièrement au Togo est confrontée à un certain
nombre de contraintes occasionnant des pertes importantes de rendement. Parmi
ces contraintes, figure en bonne place la maladie de l'enroulement foliaire du
gombo (OLCD). Cette maladie induisant des symptômes de repliements
foliaires est fréquemment observée sur le gombo ces
dernières années au Togo tout comme dans plusieurs pays de
l'Afrique de l'Ouest. Aussi c'est un légume longtemps
délaissé par la recherche et considéré comme «
culture de femme » dans la plupart des pays dans lesquels il est
cultivé.
Dans le but d'identifier les espèces virales
associées à cette maladie et leur diversité au Togo, des
travaux de caractérisation moléculaire ont été
menés sur 118 échantillons de feuilles de gombo (symptomatiques
et asymptomatiques) collectés dans les 4 zones agro écologiques
(Foret, littoral, savane humide et savane sèche) du Togo. Au total 12
localités à grande production ont été
prospectées. Divers symptômes caractéristiques d'une
infection à Begomovirus ont été observés
sur la plupart des parcelles prospectées. Bien que l'observation des
symptômes soit utile pour l'identification de la maladie de l'enroulement
foliaire du gombo, elle n'est pas appropriée pour déterminer des
espèces virales responsables de l'OLCD. Ainsi les ADNs totaux des
échantillons ont été extraits et soumis à un
diagnostic moléculaire par PCR (Polymerase Chain Reaction)
conventionnelle à l'aide d'une amorce
dégénérée (VD 360/CD 1266) (Dellate et
al., 2005) spécifique au gène codant pour la protéine
de capside (CP) des Begomovirus. La CP a été
amplifié avec succès à partir de certains
échantillons (65/118). Ce couple d'amorces semblerait le mieux
adapté à la détection moléculaire des
Begomovirus en Afrique de l'Ouest. Les résultats obtenus ont
permis d'associer les divers symptômes observés au groupe des
Begomovirus, corroborant ainsi les travaux antérieurs portant
sur le OLCD dans certains pays de l'Afrique de l'Ouest dont le Burkina Faso
(Barro et al., 2007 ; Konaté et al., 1995 ;
Tiendrébéogo et al., 2010), la Côte d'Ivoire
(Séka et al., 2016 ), le Niger (Shih et al., 2009 ),
le Mali (Kon et al., 2009), le Ghana (Swanson et Harrison, 1993), le
Nigéria (Swanson et Harrison, 1993) et du centre comme le Cameroun (Leke
et al., 2007) et le Tchad (Swanson et Harrison, 1993). Afin de
déterminer les espèces de Begomovirus responsables, un
séquençage s'avérait nécessaire. À cet
effet, les
45
| 


