Chapitre I : synthèse bibliographique
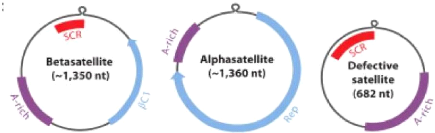
Figure6: Représentation des ADN
â satellites et á satellites codant respectivement pour les
protéines ßC1 et Rep, et des molécules défectives
sans ORFs. A-rich (Adenine-rich region); CR (Common Region); IR (Intergenic
Region); SCR (Satellite Conserved Region) (Zhou, 2013).
3.3. Réplication des Begomovirus
Les Begomovirus utilisent deux mécanismes pour
la réplication de leur génome. Une fois dans le cytoplasme
cellulaire de la plante hôte, l'ADN simple brin viral,
décapsidé, rejoint le noyau pour être converti en forme
réplicative ADN double brin (ADNdb) par la polymérase cellulaire
de l'hôte. L'ADN double brin est capable de se répliquer par un
mécanisme de cercle roulant (RCR), de manière analogue aux
plasmides bactériens et phages à ADN sb circulaire, et par un
mécanisme de réplication dépendante de la recombinaison
(RDR) (Hanley-Bowdoin et al., 1999; Jeske et al., 2001;
Preiss et Jeske, 2003 ; figure 6). La protéine Rep seule est suffisante
à l'initiation de la réplication RCR en introduisant une
ouverture au sein du motif conservé de la tige boucle (TAATATT?AC)
contenant le site d'initiation (?) de la RCR (Hanley-Bowdoinet al.,
2000). Ainsi, elle dérégule le cycle cellulaire et fournit les
facteurs cellulaires compatibles avec la réplication de l'ADN. Une
dernière étape permet le transport des génomes filles
d'ADNsb vers les cellules adjacentes via les plasmodesmes, ou leur
encapsidation pour former de nouvelles particules virales (Gutierrez, 1999).
| 


