Chapitre 3
Les résultats sur les génotypes
Résumé : Il est à noter
que les résultats de ce travail seront illustrés dans ce rapport
sur huit génotypes, qui ont été choisis parmi les 120
génotypes à traiter. Les 112 génotypes restants seront
traités dans un autre document. Ceci est du au fait que pour chaque
génotype, il faudrait 4 à 5 figures (soit au moins deux pages par
génotype pour rendre les figures visibles), ce qui prendrait plus de 200
pages dans le rapport. Donc pour illustrer la qualité du travail et
diminuer le volume du rapport, nous nous bornerons à présenter
seulement les résultats pour ces 8 génotypes. Rappelons aussi
qu'on applique le même traitement sur les 120 génotypes. Trois
documents seront préparés pour contenir les résultats sur
les 120 génotypes :
-Le rapport (document actuel), contiendra les 8
génotypes choisis.
-Un grand document, contiendra les résultats finaux sur
toute la population (les 112 Génotypes restants + aussi
les 8 génotypes choisis).
-Un fichier des valeurs des paramètres des 120
génotypes.
Comme dit à la sectin1.3.3, on va
ajuster selon deux procédures, la première consiste à
ajuster indépendamment sur les deux conditions (témoin et
stress). Cette procédure aboutit donc à deux vecteurs de
paramètres différents pour les deux conditions et la
deuxième procédure consiste à ajuster les
paramètres simultanément pour les deux conditions. On aboutit
alors à une combinaison de paramètres communes aux deux
conditions. Dans les deux premières sections, on traitera ces deux
procédures d'ajustement, puis en troisième section on fera une
analyse sur la variabilité des paramètres ajustés et
à la dernière section on présentera les résultats
finaux.
Il est à noter aussi que dans toute procédure
d'ajustement, l'heure de récolte prise en compte au niveau de
modèle de croissance du fruit est 10H qui correspond
à l'heure moyenne des prélèvements de fruits pour les
valeurs observées (sur par exemple la Fi-gure3.1, on
voit bien que la teneur en matière sèche pour un génotype
dans une condition donnée commence à croitre à partir de
l'heure 10H) et les huit paramètres seront ajustés sur les bornes
suivantes :
> phi_max E [2.0e - 03, 3.0e - 01] et
Y_param E [0.1, 20.0]
> Lp E [2.0e - 04, 1.0] et tstar E
[1.0, 1500.0]
> tau_a E [100.0, 400.0] et el E
[0.0, 0.30]
> pi_f0 E [0.0, 30.0] et nu_m E [1.0e
- 03, 0.050]

31
FIGURE 3.1 La teneur en matière sèche
prédite par le modèle au cours de 24H.
3.1 Les résultats d'ajustement
indépendant
Dans cette section, on illustre les résultats selon la
première procédure d'ajustement sur les 8 génotypes
choisis représentatifs de la population (120 génotypes). Pour
avoir ces résultats, l'algorithme NSGA-II a été
appelé sur les valeurs de la Table3.1 (revoir la
sectin2.5) par génotype. Les paramètres cprob et
mprob sur cette table, représentent respectivement la probabilité
d'appliquer les opérateurs de croisement et de mutation sur l'ensemble
des individus de la population dans l'espace de recherche. L'opérateur
de croisement a pour but d'enrichir la diversité de la population en
manipulant la structure des individus. L'opérateur de mutation a pour
but de garantir l'exploration de l'espace de recherche, c'est-à- dire le
fait que tout point de l'espace de recherche peut être atteint en un
nombre fini de mutations.
Sur l'ensemble des figures (Figure3.2, ...,
Figure3.9) de ces résultats, l'axe X représente
l'age des fruits (jours) et l'axe Y représente soit
MF(observée-point ou prédite-ligne) soit MS(observée-point
ou prédite-ligne). Et comme indiqué sur les graphiques chaque
courbe représente le meilleur compromis choisi par l'un des trois
critères de choix :
-dist min : Critère de sélection par la distance
minimale.
-dist max : Critère de sélection par la distance
maximale
-seuil :Critère de sélection par seuil
Il apparaît aussi que certains compromis sont identiques
ou très proches ne permettant pas de les distinguer sur les courbes.
32
|
Les paramètres de NSGA-II
|
Les valeurs des paramètres de NSGA-II
|
|
idim
|
8
|
|
odim
|
2
|
|
generations
|
50
|
|
popsize
|
100
|
|
cprob
|
0.7
|
|
cdist
|
5
|
|
mprob
|
0.1
|
|
mdist
|
10
|
TABLE 3.1 Tableau des valeurs de paramètres de NSGA-II
pour analyser sur 8 paramètres.
|
Critère de sélection
|
Le RRMSE de MF
|
Le RRMSE de MS
|
|
Distance minimale
|
12.54
|
12.80
|
|
Seuil
|
12.54
|
12.80
|
|
Distance maximale
|
28.86
|
12.25
|
TABLE 3.2 Les valeurs des RRMSE(s) trouvées salon les
critères de sélection pour le génotype SSD173 en condition
témoin.
Sur l'ensemble des génotypes, on voit bien que chaque
compromis trouvé par chacun des critères de sélection
simule correctement les données observées pour le génotype
désigné. Pour choisir un seul compromis parmi ces trois
trouvés par génotype, on choisit celui ayant des valeurs
minimales à la fois de RRMSE de MF et puis de RRMSE de MS (revoir la
sectin2.5). Par exemple sur la Table3.2, on
choisit pour le génotype SSD173 en condition témoin le compromis
trouvé par les critères de sélection par distance minimale
et seuil (même compromis). Nous allons donc voir dans ce qui suit qu'un
seul critère de sélection sera adapté. Donc d'une
manière globale, le modèle simule correctement l'évolution
au cours temps des deux variables MF et MS sur l'ensemble des génotype.
Nous allons maintenant voir les résultats selon la deuxième
procédure (section suivante).
Remarque 3.1. La courbe
médiane est la courbe passant par les médianes
des clusters et tracer pour voir la qualité d'ajustement pour le
meilleurs compromis choisi au final par génotype.
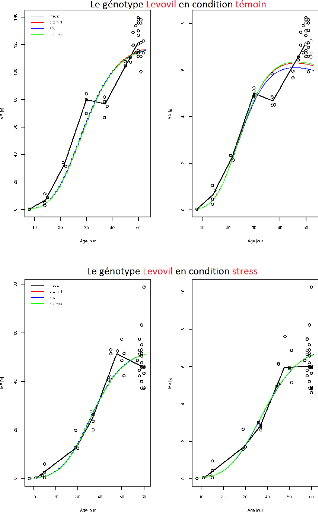
FIGURE 3.2 L'ajustement indépendant des paramètres
du génotype Levovil.
33
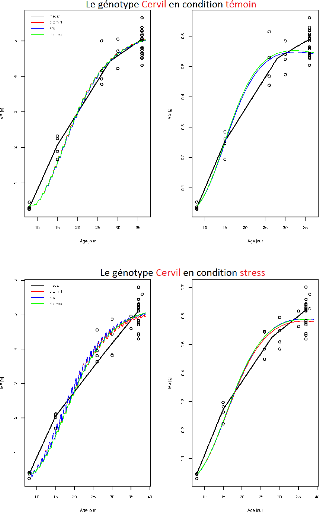
FIGURE 3.3 L'ajustement indépendant des paramètres
du génotype Cervil.
34

FIGURE 3.4 L'ajustement indépendant des paramètres
du génotype CervXLev.
35

FIGURE 3.5 L'ajustement indépendant des
paramètres du génotype SSD106.
36
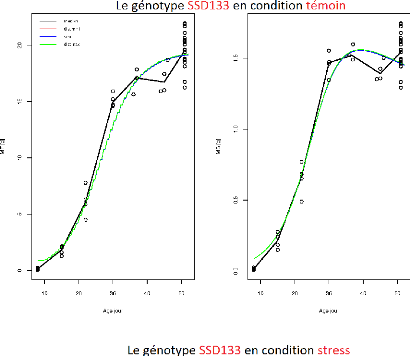
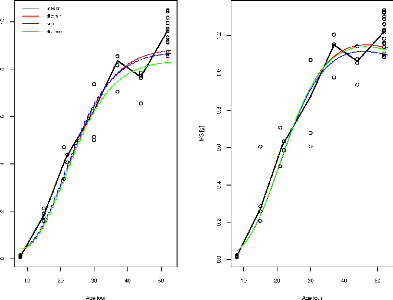
FIGURE 3.6 L'ajustement indépendant des paramètres
du génotype SSD133.
37
Le génotype SSD45 en condition témoin
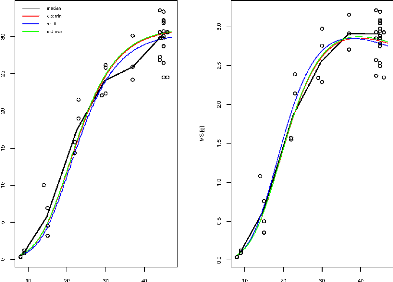
Age jour Age jour

Le génotype SSD45 en condition stress
N(V
FIGURE 3.7 L'ajustement indépendant des paramètres
du génotype SSD45.
Age jour Age jour
38

FIGURE 3.8 L'ajustement indépendant des paramètres
du génotype SSD18.
39
FIGURE 3.9 L'ajustement indépendant des paramètres
du génotype SSD173.
40

41
| 


