3.2 Les résultats d'ajustement
parallèle
Dans la section précédente, pour chaque
génotype, 8 paramètres ont été ajustés
indépendamment suivant les conditions donnant donc 8 valeurs de
paramètres en conditions témoin et 8 en conditions stress, soit
16 valeurs par génotype. Avec la procédure d'ajustement
parallèle, nous allons ajuster tous les génotypes avec un seul
jeu de paramètre, quelques soient les conditions (témoin ou
stress). Avec toujours les mêmes huit génotypes, on illustre les
résultats selon cette procédure. Ces résultats ont
été obtenus après avoir appelé l'algorithme
NSGA-II sur encore la même Table3.1 par
génotype. Sur l'ensemble des figures (Figure3.10, ...,
Figure3.13) de ces résultats deux génotypes par
figures, les informations indiquées sont :
> Le nom de génotype.
> Les.val.of.param : Les valeurs des paramètres
trouvées pour ce génotype.
> sum.of.RRMSE : le somme des valeurs des RRMSE(s) de
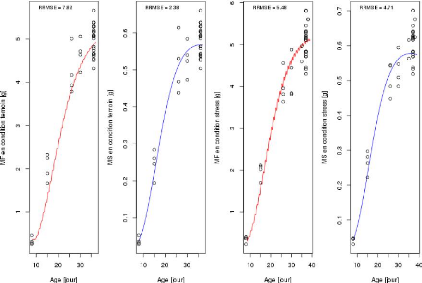
MF(respectivement, MS) en condition témoin (respectivement, stress).
La valeur de RRMSE est relative avec la qualité
d'ajustement de la courbe associée, c'est-à-dire que si cette
valeur est petite, alors la courbe va beaucoup mieux ajuster les données
observées sinon elle sous-ajuste. D'une manière
générale, on trouve que le modèle simule
l'évolution au cours temps des deux variables MF et MS sur l'ensemble
des génotype selon cette procédure, ce qui nous permet d'aller
faire une analyse sur la variabilité des paramètres (section
suivante).
Vu que cette procédure est celle choisie pour cette
analyse et éventuellement les résultats finaux, il a fallu faire
un seul choix sur les critères de sélections et comme dit
à la sectin2.5.1.4, c'est le critère de seuil
qui est le plus performant par rapport aux critères de sélection
par distance suivi du critère de la distance minimale. En effet le
critère de sélection par la distance minimale est forte pour deux
critères quand on minimise les RRMSE(s) seulement mais vu que cette
procédure a ajouté la somme pondérée
(sectin2.4), ce critère se détériore
parfois en amenant à choisir un compromis très bonne pour une des
fonctions objectif mais beaucoup moins bonne pour la seconde fonction objectif.
Ainsi que avec la minimisation de quatre critères avant découvrir
la Remarque2.2 de NSGA-II. Donc dans tout ce qui suit, seule
la procédure d'ajustement parallèle couplée au
critère de sélection par seuil sera utilisée.
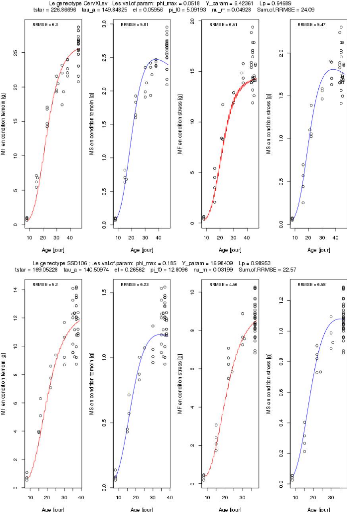
Le gneotype Cervil ; Les.val.of.param: phimax = 0.1609 Y_param
= 10.64766 Lp = 0.81023
tstar = 29.31592 tau a = 1 E0.66633 el = 0.28917 pi f0 =
6.87033 nu_m = 0.04017 Sum.of.RRMSE = 20.39
40
I I I I 1
10 20 30
Age Dour]
Age Dour]
10 20 30
Age []our]
10 20 30 40
Age []our]
MS en condition stress [g]
a
MF en condition temoin [g]
N
O
o
MS en condition temoin [g]
C)
nd tion stress [g]
m LL
Cd
O
ô
ô
o
N
ô
RRMSE -4.71
o
O
6 a
a 0
a

Age [Our]
Age [Our]
Age []our]
Age []our]
Le genotype Levovil ; Les.val.of.param: phi_max = 0.06724 Y_
cram = 4.30963 Lp = 0.42637
tatar = 218.47987 tau a = 277.57125 el = 0.14175 pi 0= 7.79146
nu_m = 0.03971 Sum.of.RRMSE = 55.36
1 1 I I I
10 30 50
1 1
10 30 50
MFen condition temoin [g]
ME en condition stress [g]
RRMSE- 10137
0
0
RRMSE - 1933
O
N
O
o -
10
a
MS en condition stress [g]
o a
O N
10 20 30 40 50
10 20 30 40 50
42
FIGURE 3.10 L'ajustement parallèle des paramètres
des génotypes Cervil et Levovil.
43
FIGURE 3.11 L'ajustement parallèle des paramètres
des génotypes CervXLev et SSD106.
MS en co nd do n te mo in [g]
ME en condition stress [g]
MS en condition stress [g]
MFen condition temoin [g]


Le wmotype SSD133 Les.val.of.param: phimax = 0.07864 Y_ram =
5.82745 Lp = 0.99707
tstar = 401.77526 tau a = 186.68099 el = 0.14069 pi f0 = 6.60399
nu_m = 0.01725 Sum.of.RRMSE = 35.87

Age [Our]
Age [Our]
Age [pur]
Age [pur]
MF en condition temoin [g]
10 20 30 40 50
MS en condition temoin [g]
10 20 30 40 50
MF en cond fion stress [g]
MS en condition stress [g]
N
N
O
10 20 30 40 50
10 20 30 40 50
Le gneotype SSO45: Les.val.of.param: phi_max = 0.04307 Y_ram =
10.83638 Lp = 0.71487
tstar = 203.84713 tau a = 178.54754 el = 0.06858 pi f0=
10.93544 nu_m = 0.04633 Sum.of.RRMSE = 31.67

LP N
N
I 1 I
10 20 30 40
Age [pur]
I I I
10 20 30 40
Aga [pur]
I
10 20 30 40
Age [Our]
I 1 I
10 20 30 40
Age [pur]
44
FIGURE 3.12 L'ajustement parallèle des paramètres
des génotypes SSD133 et SSD45.

50
I 1 I
10 20 30 40
RRMSE- 12.1 00
0
,
b b°
o
4 00
O
O
Le geneotype 55018 ; Les.val.of.param: phi_max = 0.15587
Y_param = 3.71011 Lp = 0.9E031
tstar = 35.27268 tau a = 217.59222 el = 0.27496 pi f0 = 5.19321
nu_m = 0.04434 Sum.of.RRMSE = 38.21
o -
Age []our]
I I I
10 20 30 40 50
Age []our]
10 20 30 40
Age []our]
10 20 30 40
Age []our]
MF-i7 condition stress [g]
MS en condition stress [g]
o
o
In
o
rI
In
o
MS en condition temoin [g]
o
o
N
MF en coud tion temoin [g]
RRMSE-222
4
0
o
a
00
00
O
°
O
b
Le geneotype 550173: Les.val.of.param: phi_rnax = 0.04503 Y_ram
= 4.74944 Lp = 0.35584
tstar = 231.03342 tau a = 207.55825 el = 0.16539 pi f0 =
6.33521 nu_m = 0.03257 Sum.of.RRMSE = 51.19

I I
10 20 30 40
Age []our]
MS en condition stress [g]
-r
MFen condition temoin [g]
ms en .:o nd tio n te mo in
o
I 1 I
10 20 30 40
I I I
10 20 30 40
10 20 30 40
Age [Our]
Age [pur]
Age [pur]
RRMSE-1255
a
a °
°
45
FIGURE 3.13 L'ajustement parallèle des paramètres
des génotypes SSD18 et SSD173.
46
| 


