3.8.3. Performance du CA-CFAR-BI-2D(F1F2)
Nous procédons toujours par les trois étapes :
a) On fixe PfaR F1F2 =10-6.
b) T F1F2 est calculé après
résolution de l'équation (3.22).
c) On remplace T F1F2 dans l'expression (3.21) et on
calcule la probabilité de détection PdRF1F2 pour
différentes valeurs de s.
Pd en fonction du rapport SNR avec une Pfa=1e-6
|
Pd
|
1 0.9 0.8 0.7 0.6 0.5 0.4 0.3 0.2 0.1 0
|
?
Pd=0.93 / SNR=17

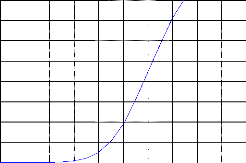
FNR=50(dB)
CACFAR-BI-2D-F1F2 / e0=.05
0 2 4 6 8 10 12 14 16 18 20
SNR (dB)
Figure 3.27 : la probabilité de détection du
CA-CFAR-BI-2D(F1F2)
en fonction du SNR pour r=50dB, e0 = 0,05,
M=4, L=6 et N=16.

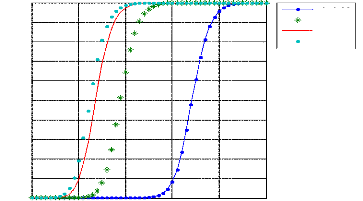
Figure 3.28 : la probabilité de détection du
CA-CFAR-BI2D(F1F2) en fonction du SNR pour ????0 = 0,05,
r=17..50dB,
Pd en fonction du rapport SNR avec une Pfa=1e-6
0 10 20 30 40 50
Pd
0.9
0.8
0.7
0.6
0.5
0.4
0.3
0.2
0.1
0
1
CACFAR-BI-2D-F1F2 / e0=0,02...0,05
M=4 L=6 N=16
FNR=50(dB)
e0=0,02 e0=0,03 e0=0,04 e0=0,05
SNR (dB)

Figure 3.30 : la probabilité de détection du
CA-CFAR-BI-2D(F1F2) en
fonction du SNR pour ????0 = 0,05, r=17..50dB, M=4, L=6 et
N=32.
Pd en fonction du rapport SNR avec une Pfa=1e-6


e0=0,02 e0=0,03 e0=0,04 e0=0,05
1
0.9
0.8

0.7
0.6
0.4
CACFAR-BI-2D-F1F2
0.3
FNR=50(dB)
0.2
M=4 L=6 N=32
0.1
0
Pd
0.5
/ e0=0,02...0,05
0 5 10 15 20
SNR (dB)

Figure 3.32 : la probabilité de détection du
CA-CFAR-BI-2D(F1F2) en
fonction du SNR pour ????0 = 0,05, r=17..50dB, M=4, L=6 et
N=48.
Pd en fonction du rapport SNR avec une Pfa=1e-6

e0=0,02 e0=0,03 e0=0,04 e0=0,05
1
0.9
0.8

0.7
0.6
0.4
CACFAR-BI-2D-F1F2
Pd
0.5
/ e0=0,02...0,05
0.3
FNR=50(dB)
0.2
M=4 L=6 N=48
0.1
0
0 5 10 15 20
SNR (dB)
Sur les figures 3.27, 3.28 et 3.29 où le nombre des
cellules de référence N=16, ont retient que le CA-CFAR-BI-2D-F1F2
est un schéma efficace, néanmoins ont remarque une nette
diminution de la probabilité de détection pour des valeurs e0
<0,04. Ceci est dû à une mauvaise estimation du signal
parasite. On peut améliorer cette estimation en augmentant le nombre des
cellules de références(N).
Sur les figures 3.30, et 3.31 où le nombre des cellules
de référence N=32, l'estimation du signal parasite a
été améliorer et la probabilité de détection
aussi. Ont remarque que la ???????? pour les différentes valeurs de e0
commence à converger vers un seul graphe.
Pour N=48 ont remarque sur les figures 3.32 et 3.33 que le
graphe de la probabilité de détection est pratiquement le
même pour les différentes valeurs de e0 et de FNR.
Pour N=48 et ???????????? = 10-6 la probabilité
de détection avoisine 0,98 pour toute les valeurs de e0 et r
(e0=0,02...0,05 et r=17dB...50dB).
| 


