|

REPUBLIQUE DE COTE D'IVOIRE
Union-Discipline-Travail
Ministère de l'Enseignement
supérieur
et de la Recherche Scientifique
Laboratoire de
Pharmacodynamie-biochimique
Année Universitaire
2017-2018
MEMOIRE
Présenté pour l'obtention du
Diplôme de Master
de
Biotechnologies-Biosécurité-Bioressources
de
l'Université Félix HOUPHOUET- BOIGNY
Spécialité : Biologie Fonctionnelle &
Moléculaire
Numéro d'ordre
Par
SYLLA Aboubacar
CARACTERISATION GENOMIQUE DES
BACTERIOPHAGES ISOLES EN
CÔTE D'IVOIRE
Composition du Jury
Soutenu publiquement
M. AHONZO Niamke L. S...Professeur Titulaire...UFHB
Président du Jury M. DJAMAN A. Joseph....Professeur Titulaire...UFHB
Superviseur
Madame KAKOU N. Solange...Maître de Recherches....
IPCI Directeur Scientifique
M. BIDIE Alain D. P....Maître de
Conférences....UFHB Examinateur
Dédicaces
i

Je dédie ce travail à :
Ma
mère,
Autant de phrases aussi expressions soient-elles ne sauraient
montrer le degré d'amour et
d'affection que j'éprouve pour
toi. Tu m'as comblé avec ta tendresse et d'affection tout le long
de
mon parcours. Tu n'as cessé de me soutenir et de m'encourager durant
toutes les années de
mes études, tu as toujours
été présent à mes côtés pour me
consoler quand il fallait. En ce jours
mémorable, pour moi ainsi que
pour toi, reçoit ce travail en signe de ma vive reconnaissance et
mon
profond estime. Puisse le DIEU TOUT PUISSANT te donne santé, bonheur et
longue vie
afin que je puisse te comble à mon tour.
Mon
épouse,
Ton encouragement et ton soutien étaient la
bouffée d'oxygène qui me ressourçait dans les
moments
pénibles, de solidarité et de souffrance. Merci d'être
toujours à mes côtés, par ta
présence, par ton
amour dévoué et ta tendresse, par te donner du goût et du
sens à notre famille.
En témoignage de mon amour, de mon
admiration et de ma grande affection, je te prie de trouver
dans ce travail
l'expression de mon estime et mon sincère attachement. Je prie DIEU le
TOUT
PUISSANT qu'il te donne bonheur et prospérité.
ii

Remerciements
Je remercie tout d'abord, le Bon DIEU Tout Puissant, de
m'avoir donné la force d'accomplir ce travail.
Je remercie Pr DOSSO M., Directrice de
l'Institut Pasteur de Côte d'ivoire qui a servi de cadre à
la réalisation dudit projet.
Un immense merci à Pr DJAMAN Allico
Joseph, Professeur Titulaire à l'Université Félix
Houphouët Boigny, Directeur du laboratoire de Pharmacodynamie-Biochimique
et Chef de Département de Biochimie à l'Institut Pasteur de
Côte d'Ivoire, qui est le directeur de ce mémoire, et qui s'y est
impliquée de manière considérable. Merci d'avoir fait plus
que ton travail de directeur.
Je tiens à exprimer mes remerciements et ma
reconnaissance à Dr KAKOU NGAZOA E. Solange,
codirectrice de ce mémoire, responsable de la Plateforme de
Biologie Moléculaire à l'Institut Pasteur de Côte d'Ivoire,
pour tout le temps qu'elle a consacré à la réalisation de
ce projet, pour ses encouragements, son support, ses conseils, sa patience, sa
gentillesse, et son esprit responsable, critique et
rigoureux.et son aide précieuse
tout au long de ce mémoire.
Mes vifs remerciements à tout le personnel de la
Plateforme de Biologie Moléculaire de l'Institut Pasteur de Côte
d'Ivoire, particulièrement à Monsieur Yavo Albert Konan,
Madame Zogba née Sangaré Flany et Madame
Mambé née Ani Perpétu pour tout leur soutien et
tous les bons conseils.
Je ne peux oublier Mlle ADDABLAH Ameyo
Yayra Audrey dont l'ardeur au travail et sa sympathie ont
créé des conditions tout à fait propices pour une bonne
productivité scientifique, mais aussi pour l'intérêt
qu'elle a accordé à ce travail.
Je remercie Pr Sylvain Moineau de l'Université de
Laval, sans lequel le Projet Phage n'aurait pas abouti. Merci pour les souches
de références.
Merci aux enseignants et chercheurs du laboratoire de
Pharmacodynamie-Biochimique de l'Université Félix Houphouët
Boigny.
Je tiens à remercier Messieurs et Mesdames, membres de
jury d'avoir acceptés de juger ce travail de mémoire de Master 2.
Je vous remercie vivement d'avoir accepté d'examiner ce travail et de
faire partie de ce jury.
Enfin, je tiens à exprimer toute notre gratitude à
tous ceux qui nous ont aidé de près ou de loin à
l'élaboration de ce travail.

iii
TABLE DES MATIÈRES
i
ii
iii
vi
vii
viii
1
3
3
3
3
6
6
6
7
7
9
9
9
9
10
11
11
12
12
12
12
13
13
16
Dédicaces
Remerciements ..
TABLE DES MATIÈRES ..
LISTE DES ABRÉVIATIONS
LISTE DES FIGURES
LISTE DES TABLEAUX
Introduction ..
Revue bibliographique
1. Bactériophages ..
1.1. Définition
1.2. Historique
1.3. Classification des bactériophages
1.4. Biologie des bactériophages
1.4.1. Cycle lytique « phages virulents »
1.4.2. Cycle lysogénique « phages
tempérés » ..
1.4.3. Cycle chronique « phages filamenteux »
2. Applications des bactériophages ..
2.1. Phagothérapie ..
2.1.1. Pharmacologie de la phagothérapie
2.1.2. Intérêts de la phagothérapie
2.1.3. Contraintes de la phagothérapie
..
2.2. Essor du génie génétique et de la
Biotechnologie ..
2.3. Agroalimentaire
2.4. Traitement des eaux usées ..
2.5. En médecine humaine et vétérinaire
..
3. Rôle des phages en Biologie moléculaire
.
3.1. Rôle des bactériophages dans le transfert des
gènes (phage lysogénique)
3.2. Les phage display
3.3. Les protéines des phages
3.4. La génomique des phages dans la nature
16 18 18 18 21 21 21 21 21 21 21 21 25 25
25
25
25
25
25
26
26
26
26
26
27 29 29 31 31
31
32
34

iv
3.5. La métagénomique des Bactériophages
..
4. Caractérisation génomique
4.1. Cas du bactériophage T4
4.2. Méthodes de génotypage
Matériel et Méthodes
1. Matériel ..
1.1 Cadre d'étude
1.2. Matériel d'étude
1.2.1. Matériel biologique .
1.2.2. Matériel technique ..
1.2.2.1. Equipements et consommables .
1.2.2.2. Réactifs et tampons
2. Méthodes
2.1. Concentration des bactériophages
2.1.1. Principe .
2.1.2. Protocole
2.2. Extraction de l'ADN des phages .
2.2.1. Principe .
2.2.2. Protocole
2.3. Dosage fluorimétrique
2.3.1. Principe
2.3.2. Protocole
2.4. Digestion enzymatique
2.4.1. Principe
2.4.2. Protocole
2.5. Electrophorèse sur gel d'agarose
2.6. Electrophorèse en champ pulsé
2.6.1. Encapsidation des phages dans l'agarose
2.6.2. Lyse virale des phages dans les plugs d'agarose
2.6.3. Digestion enzymatique du génome
2.6.4. Électrophorèse sur gel d'agarose
2.7. Analyse des profils de restriction
34
34
34
35
35
35 37 37 39 41
43
44

v
2.7.1. Evaluation de la similarité des profils
2.7.2. Analyse phylogénétique
2.7.3. Attribution de pulsotype et de cluster
Résultats et Discussion
1. Résultats
1.1. Analyse du profil génomique des phages par
les endonucléases sur gel d'agarose
1.2. Analyse du profil génomique par la méthode en
champ pulsé .
1.2.1. Analyse du profil génomique
générée par XbaI
1.2.2. Analyse du profil génomique
générée par XhoI
2. Discussion ..
Conclusion Et Perspectives ..
Références bibliographiques

vi
LISTE DES ABRÉVIATIONS
ADN : Acide
désoxyribonucléique
AFLP : Amplified Fragment Length
Polymorphism
ARN : Acide ribonucléique
ARNm : Acide ribonucléique messager
(ARN messager)
ARNt : Acide ribonucléique transfert
(ARN de transfert)
bp : paire de base
BSA : Bovine serum albumin
dsDNA : double-stranded DNA (ADN double
brin)
dsRNA : double-stranded RNA (ARN double
brin)
EDTA : Éthylène Diamine
tétra-acétique acide
ICTV : International Committee on the
Taxonomy of Viruses
IPCI : Institut Pasteur de Côte
d'ivoire
kbp : kilobase pair
NaCl : chlorure de Sodium
NGS : Next Generation Sequencing
OMS : Organisation Mondiale de la
santé
PCR: polymerase Chain reaction
PEG: polyEthylène Glycol
PFGE: Pulse Field Gel Electrophoresis
RADP: Random Amplified Length Polymorphism
RFLP: Restriction Fragment Length
Polymorphism
Rpm : Rotation par minute
SDS : Sodium Dodecyl Sulfate
TAE : Tris-Acetate-EDTA
TBE : Tris-Borate-EDTA
ufp/ml : Unité formant plage de
lyse
RFLP : Restriction Fragment Length
Polymorphism
LISTE DES FIGURES

vii
Figure 1 : Schéma illustrant la
morphologie et les différentes protéines structurales (Effantin,
2005): . 5
Figure 2 : Schéma des cycles
d'infection phagique (lytique et lysogénique) (García et al.,
2010). 8
Figure 3 : Processus d'infection du phage T4
(Kanamaru et al., 2002). 15
Figure 4 : Arbre phylogénétique
des Caudovirales (Roux et al., 2012). 17
Figure. 5 : Carte génétique du
génome du bactériophage T4 (Miller et al., 2003). 19
Figure 6 : Schéma descriptif de
l'électrophorèse en champ pulsé. 30
Figure 7: Profil des phages après
digestion par des endonucléases. 36
Figure 8 : Électrophorèse en
champ pulsé de bactériophage digéré par l'enzyme de
restriction XbaI 38
Figure 9 : Dendrogramme
généré par le logiciel Bionumerics® avec l'enzyme
XbaI. 38
Figure 10: Électrophorèse en
champ pulsé des génomes de bactériophages après
digestion par XhoI.40
Figure 11 : Dendrogramme
généré par le logiciel Bionumerics® avec l'enzyme
XhoI. 40

viii
LISTE DES TABLEAUX
Tableau I : Différents gènes
fonctionnels en fonction des couleurs (Miller et al., 2003). 20
Tableau II: Matériel biologique 22
Tableau III : Appareils et consommables 22
Tableau IV : Réactifs et Tampons du
PFGE 23
Tableau V : Réactifs et Tampons 24
Tableau VI : Les caractéristiques des
endonucléases utilisées 24
Tableau VII : Mélange
réactionnel de la digestion enzymatique 28
Tableau VIII : Mélange
réactionnel de la pré-digestion enzyamtique du PFGE 33
Tableau IX: Mélange réactionnel
de la digestion enzyamtique du PFGE 33
Tableau X : Détermination du profil
génomique des phages. 36
Introduction

1
Introduction
Les bactériophages ou phages sont des virus qui
infectent les bactéries. Ces parasites intracellulaires obligatoires et
absolus ont été découverts par Frederick W. Twort et Felix
d'Hérelle en 1915 et 1917 respectivement. Le terme bactériophage
a été inventé par d'Hérelle qui signifie
étymologiquement « mangeur de bactéries
».
On les trouve partout ou l'on trouve des bactéries
(sol, mer, désert et eaux d'égouts) et ils sont visibles
uniquement en microscope électronique. Ils sont omniprésents dans
la biosphère et constituent l'entité biologique la plus nombreuse
de la planète (Williamson et al,. 2005 ; Dublanchet et
Patey, 2011). Les bactériophages constituent un modèle
biologique fascinant en écologie et en évolution.
(Ceyssens et al., 2011) L'évolution des
phages, entraînée par la résistance des hôtes
bactériens, peut affecter plusieurs gènes, et ceci est
reflété par la grande diversité génomique à
l'intérieur des espèces de phages (Brussow et al.,
2004). Les virus constituent un immense réservoir de
diversité génétique fréquemment
révélé par la découverte de nouveaux gènes
en particulier dans les génomes de phages nouvellement
séquencés (Suttle, 2005 ; Essoh et al.,
2013). Les données métagénomiques de
l'environnement humain montrent que la plus grande partie de la
diversité virale reste non caractérisée (Breitbart
et al., 2003).
L'utilisation de l'antibiothérapie a fait surgir au
cours des dernières décennies le phénomène qu'est
la résistance des bactéries aux antibiotiques ( BMR :
Bactéries Multi-Résistantes). L'origine de cette
multi-résistance est due à la pression sélective
exercée par l'utilisation abusive des antibiotiques ainsi qu'à
l'accumulation de mécanismes de défense par la bactérie
(résistance naturelle, mutation de gènes, transfert
génétique, etc.) (Vallot , 2015).
Selon un rapport de l'OMS, 60% des infections nosocomiales
sont causées par des bactéries résistantes aux
antibiotiques (Rosner et al., 2004). Face à
l'évolution inexorable des infections nosocomiales à
bactéries multi-résistantes (Carminati et al.,
2016), des infections entériques (Lappe et
al., 2009), et des infestions respiratoires (Hsu et
al., 2013), l'absence de nouvelles molécules antibiotiques
efficaces, ainsi que la montée en gravité des maladies
émergentes, l'une des alternatives pour faire face à ce
problème est un retour à la phagothérapie
(Carminati et al., 2016).
La phagothérapie est l'utilisation de
bactériophages lytiques afin de traiter un grand nombre de maladies
infectieuses d'origine bactérienne, en particulier les infections
multi-résistantes, peu effet secondaire (Dublanchet, 2009).
Cette thérapie des infections humaines est pratiquée en
Europe de l'Est en particulier en Géorgie et en Pologne
(Ackermann, 2004).

2
Introduction
L'utilisation des phages comme thérapie
nécessite plusieurs étapes de caractérisation biologique
et génomique . Après l'isolement et la purification des phages,
le matériel génétique est extrait afin de réaliser
le typage moléculaire par l'utilisation des enzymes de restrictions dans
le but de vérifier le type de matériel génétique,
la technique d'électrophorèse en champs pulsé (PFGE en
anglais) pour voir le profil génomique, la PCR et le séquencage
pour étudier les gènes par comparaison avec d'autres
séquences d'ADN , le séquencage du génome complet par la
technique la technique de Next Generation Sequencing (NGS), etc.
En Côte d'Ivoire, la phagothérapie n'est pas
utilisée comme thérapie et reste méconnue par le corps
médical professionnel (Ehui et al., 2017).
Plusieurs études récentes ont mis en évidence
l'isolement de phages lytiques à potentiel thérapeutique en
Côte d'Ivoire (Essoh et al., 2015 ; Ngazoa-Kakou et
al., 2017) mais aucune application n'a été
explorée. Vue l'émergence des bactéries et
bactéries multi-resistantes dans l'environnement (Baguy, 2014),
la sélection et la caractérisation de phages candidats
potentiels en faveur de la phagothérapie et la mise en place des
biocollections de phages lytiques en Cote d'Ivoire devient une
nécessité et une urgence scientifique.
Aujourd'hui, grâce à la biologie
moléculaire et au séquençage du génome viral, on
peut rapidement éliminer les phages contenant des séquences
génomiques potentiellement dangereuses (gènes de toxines,
gènes de virulence, gène de résistance aux antibiotiques
etc.) (Gorski et al., 2009).
Aussi, ce travail se propose comme objectif général
de :
Déterminer le profil génomique de quelques
phages isolées en Côte d'Ivoire, et comme objectifs
spécifiques de :
? Déterminer le type de matériel
génétique après digestion enzymatique suivie d'une
électrophorèse,
? Estimer la taille du génome par analyse du profil
génomique via la technique de l'électrophorèse en champ
pulsé (PFGE).
.
Revue bibliographique

3
Revue bibliographique
1. Bactériophages
1.1. Définition
Les bactériophages, communément appelés
phages sont des parasites obligatoires n'ayant aucune activité
biochimique à l'extérieur d'une cellule et n'infectent que les
bactéries (Ackermann et DuBow, 1987 ; Kutter et Sulakvelidze,
2005). Ils sont généralement spécifiques à
une espèce bactérienne voire à juste certains individus de
cette espèce. Mais, plusieurs bactériophages peuvent être
spécifiques d'une même bactérie (Sulakvelidze,
2011).
Bien que parasites obligatoires, constitués d'un acide
nucléique (ADN ou ARN) et de protéines, comme tous les virus, ce
ne sont pas des êtres vivants au sens strict, mais des entités
biologiques (Drouji, 2009).
1.2.Historique
L'histoire des phages a 100 ans. Aujourd'hui, il est admis que
la découverte du bactériophage appartient conjointement à
deux microbiologistes :
Frederick William Twort :
bactériologiste anglais qui a la propriété de la
description du principe lytique sur des colonies de Micrococcus, en
1915, il est donc indubitable qu'il avait fait une
interprétation imprécise et, surtout, n'avait pas poursuivi ses
recherches et encore moins envisagé une utilisation
thérapeutique. Il n'est donc pas contestable que le mérite de la
phagothérapie appartient à Félix d'Hérelle
(D'Hérelle, 1926).
Félix d'Hérelle : biologiste
canadien d'origine française, qui a présenté très
tôt le rapport entre un phénomène observé au
laboratoire et le phénomène de la guérison clinique en
1917. Pour lui, l'apparition de plages claires,
observée dans les boîtes de Pétri sur lesquelles
cultivaient les bactéries responsables de dysenterie bacillaire,
semblait annoncer la guérison. En effet, dès 1918,
d'Hérelle après avoir constaté que « la
pathogénie et la pathologie de la
dysenterie bacillaire sont dominées par deux facteurs agissant en sens
contraire : le bacille dysentérique, agent pathogène et le
microbe filtrant bactériophage, agent d'immunité », affirma
que c'est « logique de proposer comme traitement de la dysenterie
bacillaire l'administration, dès l'apparition des premiers
symptômes, de cultures actives du microbe bactériophage »
(D'Hérelle, 1919).
Par ailleurs, l'étude des phages a contribué
à la naissance de la biologie moléculaire et fourni des
indications fondamentales sur le mode de réplication et la
morphogenèse des virus. Les phages ont également contribué
à la compréhension de certaines épidémies et des
maladies infectieuses. Ils sont par ailleurs responsables de fermentations
défectueuses dans l'industrie laitière, et sont utilisés
dans le

4
Revue bibliographique
diagnostic (Singh et Arutyunov, 2012)
et dans le biocontrôle. Actuellement, beaucoup de recherches
sont orientées sur la génomique des phages, l'évolution,
l'écologie et la découverte de nouveaux phages à des fins
thérapeutiques.
A ce jour, plusieurs milliers (environ 6000) de phages ont
été décrits mais on estime que seulement 10% d'entre eux
ont été découverts (Ackermann, 2009).
La tête du bactériophage a
généralement une structure icosaédrique qui est
constituée par un noyau très compact d'acide nucléique,
entouré d'un enduit protéinique ou capside. La capside est
uniquement constituée de protéines dont l'ensemble forme une
enveloppe qui protège le génome viral (ADN ou ARN, en simple ou
en double brin) (Figure 1).
La capside est amorcée par un
connecteur qui est un complexe multiprotéique situé à l'un
des sommets de la capside, le connecteur est à la fois en interaction
avec elle, mais aussi avec l'ADN et les éventuelles protéines
internes ainsi qu'avec la queue du phage (Lurz et al, 2001).
Le connecteur est l'élément central de la machinerie
servant à l'incorporation de l'ADN viral dans la capside lors de la
réplication des phages dans la bactérie. Et c'est aussi en
passant par le connecteur puis la queue que l'ADN viral est injecté dans
le cytoplasme de la bactérie (Ponchon et al, 2005)
(Figure 1).
La queue est un complexe
multiprotéique assemblé indépendamment de la capside et du
connecteur. L'ancrage de la queue sur la capside au niveau du connecteur
termine le cycle d'assemblage des bactériophages. La queue des phages
est impliquée dans la reconnaissance et l'attachement aux
bactéries hôtes, dans le transpercement des membranes
bactériennes ainsi que dans le transfert de l'ADN viral de la capside
dans le cytoplasme (Ackermann, 2003).

5
Revue bibliographique

Figure 1 : Schéma illustrant la
morphologie et les différentes protéines structurales
(Effantin, 2005) :
(A) le phage T4 (Myoviridae, queue
contractile), (B) le phage T7 (Podoviridae, queue courte) et
(C) le phage SPP1 (Siphoviridae, queue non contractile),
(D) Image prise en coloration négative du phage X
(Siphoviridae, queue non contractile) illustrant les différentes parties
le constituant notamment sa queue

6
Revue bibliographique
1.3. Classification des bactériophages
Un système de classification de phages a été
élaboré par le Comité international de taxonomie
des
virus (ICTV) au cours des
dernières décennies.
La structure sur laquelle est basée la classification
des bactériophages est extrêmement variée. Les
critères de classification sont (Dublanchet, 2009) (Figure 2)
:La nature de l'acide nucléique : généralement
ADN double brin, parfois ARN simple brin (Inal , 2003) ; la
forme de la capside (icosaédrique ou tubulaire) et la présence ou
non d'une enveloppe (nommée péplos).
Approximativement 90% de tous les phages infectent et tuent
les bactéries, mais ne conviennent pas tous à la
phagothérapie (Gill et Hyman, 2010). De ce fait,
l'intérêt est porté sur les phages lytiques
représentés par 3 familles de l'ordre des Caudovirales : les
Myoviridae, les Siphoviridae
et les Podoviridae (Ackermann,
2011).
1.4. Biologie des bactériophages
La capacité de reproduction des phages à l'aide
d'une bactérie nécessite l'insertion du matériel
génétique de ce dernier au sein de la
bactérie et du détournement de la machinerie bactérienne
pour son métabolisme. Cette dernière propriété fait
des phages des particules virales car ne possédant pas d'autonomie de
reproduction. (Weinbauer, 2004 ; Drulis-Kawa et al.,
2012).
On discerne trois types de bactériophages : les phages
dits « virulents » ou « lytiques »,
représentant près de 90 % des bactériophages, ceux dits
« tempérés » ou « endogènes » ou
« lysogénique » représentant environ
10 % et ceux dits « phages filamenteux » ou
cycle chronique, bien plus minoritaires (moins d'1 %). Parmi
tous les bactériophages, les plus étudiés sont le phage
T4, appartenant aux phages virulents, et le phage X, appartenant aux phages
tempérés (Inal, 2003).
1.4.1. Cycle lytique « phages virulents »
Les phages lytiques, comme leur nom l'indique,
détruisent la bactérie. Ils détournent la
machinerie bactérienne à leur profit pour se
reproduire et se multiplier. Au terme du processus appelé cycle lytique,
la bactérie éclate et plusieurs dizaines de nouveaux phages
identiques à l'original- sont libérés dans le milieu et
donc disponibles pour s'attaquer à d'autres bactéries de la
même espèce. Véritables « tueurs professionnels
», les phages lytiques sont les prédateurs naturels des
bactéries. Ce sont précisément ces phages lytiques qui
sont utilisés à des fins thérapeutiques (depuis
D'Hérelle) pour lutter contre les infections
bactériennes (phagothérapie) (Dublanchet, 2009 ; Ravat
et al., 2015).
Le cycle lytique comporte différentes phases
(Figure 2) :
Arrimage, c'est-à-dire fixation du phage sur la
bactérie grâce à des récepteurs spécifiques
au phage et à l'espèce bactérienne à laquelle il
s'attaque.

7

8

9

10

11
Revue bibliographique
Perforation de la paroi et de la membrane bactériennes
à l'aide d'enzymes contenus dans le phage Injection de l'ADN du phage
dans le cytoplasme bactérien, le plus souvent à l'aide des
molécules contractiles du fourreau, qui se comporte ainsi comme une
seringue
Production avec fragmentation de l'ADN bactérien et
utilisation de celui-ci pour synthétiser les éléments
constitutifs des futurs phages.
Maturation et assemblage des différents
éléments produits et éclatement de la bactérie et
libération des phages dans le milieu. Cette opération conduit
à la mort bactérienne et à la production de 50 à
100 clones du phage original pour chaque cycle lytique.
1.4.2. Cycle lysogénique « phages
tempérés »
Des bactériophages, appartenant tous à l'ordre
des Caudovirales, sont appelés des phages
tempérés. Ces phages peuvent établir avec les
bactéries des rapports de longue durée, éventuellement
réversibles, qualifiés de lysogénie (Anas,
2011).
Le cycle lysogénique, aussi nommé «
lysogénisation » (Dublanchet et Patey, 2011), a
lieu lorsqu'un phage tempéré pénètre dans une
cellule bactérienne. Le génome du phage s'insère dans
celui de la bactérie et devient dès lors partie intégrante
de celle-ci (on appelle le génome viral intégré «
prophage »). La réplication de ce matériel
génétique a ainsi lieu en même temps que celui de la
bactérie. Celle-ci, dont le chromosome bactérien a
été envahi, transmet ensuite lors de sa division ce nouveau
patrimoine génétique à sa descendance (figure
2) (Dublanchet, 2009). Cet état «
silencieux » demeure jusqu'à ce qu'à un moment donné,
le cycle lysogénique s'active en cycle lytique et que le génome
du phage s'excise du chromosome bactérien. Ce changement d'état
est relativement rare : un cas pour 100 000 phages tempérés
environ (Dublanchet, 2009), mais sa fréquence est
augmentée lors d'un stress, induit par exemple par des rayons
ultra-violets, des rayons X ou des substances chimiques comme des oxydants.
1.4.3. Cycle chronique « phages filamenteux
»
La durée d'un cycle de réplication typique chez
les phages filamenteux varie entre 10 et 15 minutes. Lors de ce cycle, le phage
se fixe sur la membrane de la bactérie à partir du pilus sexuel
bactérien et injecte son ADN dans la bactérie. L'ADN viral
introduit dans la cellule bactérienne est converti de simple brin
à double brin puis répliqué et transcrit en ARNm ; les
ribosomes bactériens traduisent ensuite les ARNm en protéines de
la capside virale, qui s'insèrent ensuite dans la membrane
bactérienne, et s'assemblent entre elles en entourant un nouveau brin
d'ADN viral, tout en traversant la membrane bactérienne jusqu'à
être totalement excrétées sous forme de nouveaux virions.
Cette invasion ne détruit généralement pas les
bactéries et n'est donc pas intéressante en ce qui concerne la
phagothérapie (Ackermann, 2003 ; Clokie et al.,
2011).
Revue bibliographique

Figure 2 : Schéma des cycles d'infection
phagique (lytique et lysogénique) (García et al.,
2010)
Lors d'un cycle lytique, un phage virulent va
pénétrer au sein de la cellule bactérienne,
détruire le génome bactérien, en détourner la
machinerie pour produire des éléments constitutifs de nouveaux
phages et enfin lyser la bactérie pour libérer ces nouveaux
virions.
Lors du cycle d'un phage tempéré, le
génome phagique va s'intégrer au génome bactérien
et prendre le nom de « prophage ». Ce prophage peut être
transmis par transfert bactérien vertical (cellule mère à
cellule fille) ou horizontal (entre deux bactéries sans lien de
parenté). Parfois le cycle lysogénique s'active en cycle lytique
notamment à l'occasion d'un stress.
Revue bibliographique
2. Applications des bactériophages
2.1.Phagothérapie
Les phages sont des virus et comme tous virus se reproduisent
aux dépens de cellules vivantes. Un phage ne peut utiliser que les
constituants d'une bactérie, celle-ci appelée
bactérie-hôte. La spécificité d'hôte
caractérise un phage par la reconnaissance d'un groupe restreint de
bactéries (gamme d'hôtes) appartenant à la même
espèce, voire de quelques souches d'une seule espèce, rarement
plusieurs espèces voisines (Dublanchet, 2014).
En 1896, Ernest Hanbury Hankin,
démontrait que les eaux du Gange et de la rivière de Yamuna
contiennent des principes biologiques qui empêchent la croissance de
cultures de bactéries responsables du choléra. Selon lui ces
substances peuvent passer à travers les microfiltres (Hankin,
1896). C'est plus tard en 1915 que les bactériophages ont
été décrits pour la première fois par Frederick
William Twort (bactériologiste) lors de ses travaux sur les virus «
ultra-microscopiques » (Twort, 1925 : Dublanchet et Fruciano,
2008).
2.1.1. Pharmacologie de la phagothérapie
La pharmacologie des phages et ceux des antibiotiques
diffèrent sur trois points principaux :
Les phages ont une grande taille en comparaison aux
molécules d'antibiotiques
La variation d'efficacité des traitements dans des cas
de maladies infectieuses d'origine bactérienne en fonction de la
concentration de phages
La variation de la virulence des phages
La densité joue également un rôle
important dans l'efficacité de la phagothérapie étant
donné qu'elle est tributaire de la diffusion des phages dans
l'organisme. En raison de leur taille, et donc d'une densité
élevée, les phages mettent beaucoup plus de temps à
diffuser au sein de l'organisme hôte. Des modèles
théoriques ont clairement démontré l'importance de la dose
initiale ainsi que du temps d'inoculation sur l'efficacité d'infection
d'une population bactérienne par des phages (Payne et Jansen,
2001).
2.1.2. Intérêts de la
phagothérapie
Un avantage très important concerne la
résistance des bactéries vis-à-vis des
bactériophages. En effet, cette résistance existera toujours mais
à la différence des antibiotiques, les bactériophages sont
des organismes « vivants ». Certes, on peut contester l'appellation
« vivant » concernant ces virus, mais ce ne sont pas de banales
molécules inertes. Les bactériophages évoluent, au
même titre que les bactéries. Les phages changent avec le temps
par des mutations et ils auront toujours la capacité d'infecter les
bactéries, même si ces dernières développent de
nouvelles résistances. Donc les bactéries multi-
Revue bibliographique
résistantes aux antibiotiques continuent à
être attaquées et tuées par les bactériophages
virulents. Il s'agit de processus aussi ancien que la vie sur terre en
constante évolution (Dublanchet et Patey, 2011).
Un argument qui peut peser face aux antibiotiques, est la
diversité des bactériophages. A quelques rares exceptions
près, il existe au moins un bactériophage qui peut infecter une
bactérie (Chambon, 2006). Il ne reste plus qu'à
isoler le ou les phages pour s'attaquer à une infection donnée,
chose très aisée face aux années de recherches qu'implique
la découverte d'une nouvelle famille d'antibiotiques.
L'avantage de l'étroite spécificité des
bactériophages pour leur cible réside dans le fait qu'ils ne se
multiplient qu'au sein de l'infection. Cette particularité les oppose
aux antibiotiques, responsables du déséquilibre des flores,
elles-mêmes à l'origine de troubles digestifs, mycoses et
infections opportunistes secondaires. Il est, en outre, nécessaire
d'administrer les antibiotiques régulièrement pour palier leur
élimination (destruction et/ou excrétion) continue par
l'organisme (Dublanchet et Patey, 2011).
2.1.3. Contraintes de la phagothérapie
Le principal inconvénient de la phagothérapie
est le besoin de déterminer rapidement l'étiologie de la
bactérie responsable de l'infection avec certitude. Il n'existe pas de
phagothérapie probabiliste comme est le cas en antibiothérapie.
La spécificité des relations entre les phages et les
bactéries est un principal avantage mais également une limite
quant à l'application de la phagothérapie. Un échantillon
clinique doit être isolé, cultivé puis identifier selon des
procédures microbiologiques standards avant l'administration des phages.
Or, ce processus peut s'étendre jusqu'à 5 jours, comme dans le
cas de la fièvre typhoïde. Ce problème pourrait trouver
solution en l'utilisation de cocktails de phages (Loc-carrillo et
Abedon, 2011).
Nécessité d'utiliser un bactériophage
lytique et non un bactériophage tempéré car en plus
d'être inefficace il peut être à l'origine de transfert de
gènes pathogènes vers les bactéries. Cependant,
grâce à la biologie moléculaire et au
séquençage du génome viral, on peut rapidement
éliminer les phages contenant des séquences génomiques
potentiellement dangereuses (gènes de toxines, gènes de
virulence, gène de résistance aux antibiotiques etc.).
(Gorski et al., 2009)
Et pour finir, il est noté également le
développement de résistance chez la bactérie cible
résultant d'une mutation, d'une sélection ou de l'acquisition
d'un nouveau matériel génétique par un phage
tempéré. Il existe au moins 4 mécanismes de
résistance que peut déployer une bactérie face à un
phage spécifique : la perte ou l'absence du récepteur, sa
modification structurale, ou par un mécanisme dans lequel le
récepteur est caché au phage, contrant ainsi l'adhésion du
phage à la bactérie. La perte du récepteur se produit
lorsque la modification de la composition de la surface de la cellule
bactérienne
Revue bibliographique
advient, tel qu'il a été démontré
pour Bordetella spp. (Liu et al., 2002).
Fort heureusement, la fréquence de résistance obtenue
in vivo lors de la phagothérapie est basse (Kutter
et al., 2010) comparée aux observations faites in
vitro. De plus, l'isolement de nouveaux phages actifs à partir de
l'environnement offre de nouvelles possibilités de traitement
2.2 Essor du génie génétique et de la
biotechnologie
Les bactériophages, décrits en 1915 par F. Twort
et en 1917 par F. d'Hérelle, ont conduit à la découverte
de nombreux concepts dans la biologie et la virologie et ont permis l'essor de
la biologie moléculaire et de la génétique entre les
années 30 et les années 60.
Hershey et Chase ont pu démontrer, grâce au
bactériophage T2, que l'ADN était le support de l'information
génétique (Hershey et Chase, 1952). Aujourd'hui,
les bactériophages sont toujours utilisés en biologie
moléculaire, comme par exemple dans les techniques de clonage ou de
phage display.
Les bactériophages ont une importance majeure pour
l'évolution. Ils ont été décrits comme des agents
de transfert latéral de gènes (Canchaya et al.,
2003). Le transfert des gènes crée un avantage de
sélection pour la bactérie hôte, exemple des gènes
de résistance aux antibiotiques. Toutefois, le même processus peut
être utilisé au profit de la thérapeutique par transfert de
gènes rendant la bactérie plus sensible à certains
antibiotiques (Lu et Collins, 2009). Une autre approche
consiste à inverser la résistance à l'antibiotique en
injectant des gènes spécifiques qui confèrent une
sensibilité accrue. Ceci a été récemment
démontré par la possibilité de rendre des bactéries
résistantes à la streptomycine et à l'acide nalidixique
sensibles (Edgar et al., 2012).
2.3. Agroalimentaire
Selon les estimations des "Centers for Disease Control and
Prevention" 9,4 millions de cas de maladies d'origine alimentaire, presque 56
000 hospitalisations et plus de 1 350 décès sont
rencontrées chaque année aux États-Unis seulement
(Scallan et al, 2011). Cependant, les produits
(LISTEX P100TM et ListShieldTM) ciblent Listeria
monocytogenes, contaminant potentiel de viande, de volaille, de produits
laitiers et de légumes crus et pousse même dans les aliments
réfrigérés en provocant après ingestion une
infection grave appelée listériose. D'autres produits ciblant
Salmonella sp. (SalmoFreshTM et SALMONELEXTM) et
E. coli O157 :H7 (EcoShieldTM) sont en instance
d'autorisation. (García et al, 2010 ; Zhang et al,
2012).
L'utilisation des bactériophages semblent être
une alternative biologique aux substances chimiques utilisées
actuellement en agriculture (pesticides, antibiotiques). Concernant les
cultures, sont notamment commercialisés deux produits ciblant
Xanthomonas sp. Ou P. syringae, deux pathogènes de la
tomate

12
Revue bibliographique
entre autres (AgriPhageTM). Concernant l'élevage,
plusieurs études ont montré une efficacité de
l'utilisation des bactériophages dans la lutte contre les infections
à Campylobacter jejuni dans les élevages de volailles
(Loc-Carrillo et al, 2005 ; Wagenaar et al., 2005 ; Carvalho
et al, 2010;).
2.4. Traitement des eaux usées
Au cours du traitement des eaux usées par les boues
activées ces dernières s'installent dans les réservoirs,
et le surnageant est évacuée pour davantage de purification. Mais
ce processus est déjoué par des microorganismes filamenteux comme
Sphaerotilus natans, qui poussent de longs tentacules qui suspend les
boues et diminuer la décantation. (Choi et al.,
2011). Des phages isolés à partir d'eaux usées
ont été testés contre le chlore : le traitement standard
par le chlore, a retiré 40 % de biofilms de Pseudomona
aeroginosa. Les phages appliqués seuls ont tué 89 %. Alors
que les phages suivis de chlore ont éliminé 97 % des biofilms
(Zhang et Hu, 2013).
2.5. En médecine humaine et
vétérinaire
L'étude des phages a des implications importantes en
médecine et en génétique, surtout pour la
compréhension des infections virales, des anomalies
génétiques et de la résistance des bactéries aux
antibiotiques. Les phages sont aussi utilisés dans le traitement
vétérinaire de différentes maladies animales, y compris
les infections oculaires d'origine bactérienne (Kutateladze et
Adamia, 2008).
3. Rôles des phages en Biologie
moléculaire
3.1.Rôle des bactériophages dans le transfert
des gènes (phage lysogénique)
Les prophages inactifs, sont parfois intégrés au
génome bactérien lorsqu'ils ont subi des modifications
génétiques irréversibles. Certains sont actifs et peuvent
s'exciser du génome bactérien, se reproduire et infecter de
nouveaux hôtes. Lors de son intégration dans le génome de
la bactérie, le phage tempéré dirige la synthèse
d'un répresseur qui bloque l'expression de certains de ces propres
gènes ainsi que ceux d'autres phages lysogènes très
proches de lui. Ceci est un bénéfice non négligeable pour
la bactérie d'autant que certains prophages aident en plus la
bactérie en la protégeant contre l'infection par certains phages
lytiques sans rapport avec eux (Berdjeb et Jacquet, 2009).
Avec le temps, de nombreux résultats de recherche
montrent que non seulement les phages contribuent à l'évolution
des bactéries par le biais de la transduction mais, aussi ceux-ci
contribuent directement à la toxicité des bactéries.
L'acquisition de nouveaux gènes par l'intermédiaire d'un phage
lysogène peut, dans certains cas, modifier sensiblement la
pathogénie de la bactérie (Lebaron et Nicolas
2003). Ainsi, il a été montré que certaines
toxines contribuant significativement à la nocivité d'une

13
Revue bibliographique
bactérie sont codées par des gènes de
bactériophages. Par exemple, la toxine du choléra est contenue
dans le génome du bactériophage filamenteux CTXÖ qui infecte
les bactéries Vibrio Cholerae (Davis et Waldor, 2003).
Par ailleurs, certains gènes portés par des phages
peuvent modifier profondément n'importe quelle étape du processus
infectieux d'une bactérie comme l'adhésion, la colonisation,
l'invasion, la résistance aux défenses immunitaires, la
sensibilité aux antibiotiques et la transmissibilité entre
humains.
3.2. Les phages display
Les phages sont également utilisés pour la
recherche antivirale. La méthode de phage-display permet un criblage
rapide de molécules en utilisant leur affinité pour une cible.
Elle consiste à fusionner le peptide d'intérêt à une
protéine de surface du bactériophage. Cette méthode est
efficace pour isoler et/ou améliorer de nombreuses molécules
notamment, les inhibiteurs d'infection. Les phages recombinants sont
sélectionnés pour leur capacité de liaison à une
cible, telle qu'un anticorps, une enzyme, un récepteur purifié,
un acide nucléique ou tout autre molécule de nature non
protéique (Castel et al., 2009). Le phage par
excellence est le phage M13. Toutefois les phages fd, f1, T4 et T7 sont
également proposés (Souriau et al.,
1998).
3.3. Les protéines des phages
La diversité des phages dans la nature reste toujours
mal connue car beaucoup de bactéries de l'environnement (et de ce fait
leurs phages) ne sont pas cultivables. Pour contourner cette difficulté,
des analyses de séquences génétiques ont été
faite directement sur de l'ADN extrait de l'environnement et amplifié
sélectivement par PCR (polymerase chain reaction), sans passer
par l'étape de mise en culture (Filee et al.,
2006.). La partie centrale de la séquence du gène
codant pour la protéine principale (g23) de la tête du phage T4
(la capside) peut servir de bon substitut phylogénique à
l'ensemble du génome (Filee et al., 2005).
L'assemblage de la queue du phage T4, dont au moins les
produits de 22 gènes sont impliqués dans l'assemblage de la queue
du phage T4. La voie d'assemblage de la queue est strictement basée sur
les interactions des protéines par ordre séquentiel
(Kanamaru et al., 2002).
Les baseplates, remarquablement ont une structure
multi-protéique complexe qui sert d'unité de contrôle de
l'infection par le T4, sont assemblées en premier. Le baseplate est
composé d'environ 150 unités d'au moins les produits de 16
gènes différents sont oligomériques. Le baseplate est
assemblé à partir de six portions identiques. Le gp11 (le STF
connecting protein), gp10, gp7, gp8, gp6, gp53, et gp25 sont combinées
pour construire un coin. Le noyau central est constitué par gp5, gp27
(King, 1968) et gp29. L'assemblage de baseplate est
complété par l'adjonction de gp9, le STF-connexion
protéines,

14
Revue bibliographique
STFs, gp48, et gp54. Les deux dernières
protéines sont nécessaires pour initier la polymérisation
du tube de la queue. Le tube de queue est terminé avec gp3
(King, 1968 ; Vianelli et al., 2000). Le tube de la
queue sert de modèle pour l'assemblage de gp18 qui forme la gaine
contractile de la queue (Figure 3). La longueur du tube de la
queue est probablement déterminée par la gp29, qui participe
également à l'assemblage des baseplates (Kikuchi et King,
1975). La longueur de la gaine de la queue est
déterminée par le tube. Le tube de la queue et la gaine ont une
symétrie hélicoïdale avec une hauteur de 40.6A° et des
sous-unités répétées successivement tous les
17.7A° (Vadim et Mesyanzhinov, 2004).
L'assemblage de la queue est complété par une
gp15 hexamère qui lie le dernier anneau de la gaine de la queue
(Zhao et al., 2003). La queue s'associe à la
tête après compactage de l'ADN. L'assemblage de la queue et de la
gaine représente une structure métastable supramoléculaire
qui subit des changements de conformation après liaison des phages
à des récepteurs de la cellule hôte. Au cours de la
contraction irréversible, la longueur de la gaine diminue de 980
à 360A°, et son diamètre extérieur augmente de 210
à 270A° (Figure 3).
Le phage T4 contient trois types de protéines fibreuses
: des fibres longues (LTFs), des fibres courtes
(STFs), et moustaches. Chaque LTF se compose
par une partie proximale codée par le gène 34 et une partie
distale codée par le gène 37. Les deux parties sont
reliées par gp35 et gp36 qui forment une région charnière.
Les protéines qui forment la LTF sont homotrimeres, mais pour gp35 est
assemblé comme un monomère. Les fibres courtes
(STFs) codées par gp12 qui interagissent avec le
lipopolysacharide de la bactérie lors de l'infection (Coombs et
Arisaka, 1994).

15
Revue bibliographique

Figure 3 : Processus d'infection du phage T4
(Kanamaru et al., 2002)
(A) Phage fixant la plaque de base à la surface de la
cellule. (B) Queue contraction provoquant la perforation de la
membrane cellulaire externe par l'aiguille gp5. (C) La
dissociation gp5C à partir du tube de queue, activant ainsi les trois
domaines de lysozyme. (D) Les domaines du lysozyme
créant une ouverture dans la couche de peptidoglycane. (E)
L'association gp27 avec un récepteur sur la membrane interne et
initier la libération d'ADN dans le cytoplasme. Pour simplifier, les LTF
ne sont pas montrés en B à travers
E.

16
Revue bibliographique
3.4. La génomique des phages dans la nature
Il n'y a pas de marqueur universel pour les phages de la
même manière que le gène de l'ARNr 16S peut être
utilisé pour remplacer de manière fiable l'affinité
phylogénétique de toutes les bactéries. En effet, il n'y a
pas de gènes convenablement conservés dans tous les phages, ou
même par exemple présents dans les Caudovirales (Paul
et al., 2002 ; Clokie et al., 2011). Les chercheurs
ciblent généralement les gènes qui codent les
protéines structurales en tant que marqueurs phylogéniques. Un
gène qui a été largement utilisé est le gène
qui code pour la protéine portale qui est située au sommet du col
du phage et à travers laquelle passe l'ADN en descendant la gaine de la
queue (Zhong et al., 2002 ; Marston et Sallee, 2003).
Des amorces ont été développé à
partir de ce gène pour étudier les séquences dans les
phages de type T4 qui sont connus pour infecter un large éventail
d'hôtes bactériens (Filee et
al.,2005).
3.5. La métagénomique des
bactériophages
Les virus de l'environnement sont à la fois très
nombreux, très diversifiés et largement méconnus.
Au-delà de leur pouvoir pathogène, on leur reconnaît
aujourd'hui une influence plus large sur des aspects fondamentaux de
l'écologie de notre planète, comme les cycles
biogéochimiques, la régulation des communautés de
micro-organismes ou encore l'évolution des organismes vivants et de
leurs génomes (Willner et al., 2009).
Les approches de métagénomique virale,
consistant en un séquençage aléatoire massif des acides
nucléiques encapsidés, ont permis durant cette dernière
décennie de mieux connaître la composition des communautés
virales naturelles ainsi que la diversité génétique des
virus (Roux et al., 2013). Les
métagénomes viraux permettent de comparer les communautés
virales et ainsi de mieux comprendre la répartition des populations
virales dans la biosphère. Parmi les séquences de viromes
affiliées aux virus, la majorité est similaire à des
génomes de bactériophages, appartenant principalement au groupe
des Caudovirales (groupe de phages bactériens à structure
tête-queue).
L'existence de gènes conservés au sein des
différents groupes viraux rend en effet possible de mener des analyses
phylogénétiques pour ces groupes. Le gène codant pour la
grande sous unité de la terminase (TerL) est
le marqueur le plus utilisé dans le cas des Caudovirales.
Une partie de ces séquences de viromes sont
affiliées au groupe des phages de type T4, groupe de
référence incluant le phage T4 infectant la bactérie
Escherichia coli et utilisé comme modèle depuis
plusieurs décennies. Deux sous-groupes sont ainsi retrouvés dans
les viromes lacustres : le groupe des cyanophages de type T4 et le groupe des
Far-T4 (Comeau et Krisch, 2008), (Figure 4) pour lequel le
phage « Rhodothermus phage RM378 » est le génome de
référence le plus proche.

17
Revue bibliographique

Figure 4 : Arbre phylogénétique
des Caudovirales (Roux et al., 2012)
A) Arbre phylogénétique
basé sur l'alignement des gènes codant pour la grande
sous-unité de la terminase, gène commun à l'ensemble des
Caudovirales (bactériophages à queue). Cet arbre permet
d'associer des séquences de références (en noir) et des
séquences issues des viromes (en rouge et bleu) ; B)
zoom sur le sous-arbre comprenant la famille des phages T4. Les trois
sous-groupes connus (near-T4, cyano-T4, Far-T4) sont indiqués sur
l'arbre.

18
Revue bibliographique
4. Caractérisation génomique
4.1.Cas du bactériophage T4
Le bactériophage T4 a environ 300 gènes
probables emballés dans son génome dont la taille est de 168,903
pb. Il a un total de 289 gènes codant pour des protéines, 8
gènes d'ARNt et au moins 2 d'autres gènes qui codent de petits
ARN stables de fonction inconnue (Miller et al., 2003).
Son génome est entièrement séquencé et la
fonction de la majorité de ses ORFs est connue (Figure 5).
Les différents processus biologiques impliqués dans la
survie du phage T4 : la transcription, la traduction, le métabolisme
nucléotidique, la réplication de l'ADN, la recombinaison, la
réparation, l'encapsidation, les virions protéiques, les
protéines chaperons, la lyse de la cellule hôte, les interactions
avec l'hôte, les endonucléases et des fonctions inconnues
(Tableau I) (Miller et al., 2003).
Les études structurales des protéines du phage
T4 ont commencé avec la cristallisation et la détermination de la
structure tridimensionnelle de gpe (lysozyme). Le phage T4 lysozyme est un
excellent exemple de l'analyse structurale et de remplacement des acides
aminés ciblés utilisés de manière à
démêler les propriétés catalytiques d'une enzyme et
la conformation de la protéine (Bardy et al., 2016)
(Annexe 1).
4.2.Méthodes de génotypage
Dans le but de comprendre le mode de dissémination des
infections dans les communautés et les hôpitaux, mais
également d'appréhender les changements évolutifs qui ont
donné lieu à des avantages sélectifs, la distinction
précise entre différents isolats d'une espèce est
indispensable. Ceci peut être fait grâce aux techniques
moléculaires de typage ou génotypage.
Plusieurs techniques de génotypage sont disponibles et
des études menant à leur comparaison ont été
réalisées permettant d'identifier les plus informatives pour
évaluer la diversité des souches cliniques et environnementales
(Wommack et al., 1999).
Il s'agit de méthodes reposant sur l'analyse du
polymorphisme de longueur des fragments d'ADN générés par
digestion enzymatique. Parmi ces techniques figure le PFGE (Pulse Field Gel
Electrophoresis) d'où électrophorèse en champ
pulsé (ECP) est fondé sur l'analyse de fragments d'ADN de
très grande taille issus de la digestion enzymatique
(macro-restriction). Il demeure la méthode de choix pour plusieurs
bactéries mais aussi des bactériophages (Clokie et
al., 2011).
Les méthodes basées sur l'analyse des produits
d'amplification. Ces techniques possèdent d'une grande
sensibilité et spécificité permettant d'analyser une
faible quantité d'ADN. On a le RADP (Random Amplified Length
Polymorphism) et le AFLP (Amplified Fragment Length Polymorphism)
(Steward, G.F., 2001).

19
Revue bibliographique

Figure. 5 : Carte génétique du
génome du bactériophage T4 (Miller et al.,
2003)
Les ORFs caractérisés et hypothétiques
sont montrés sur carte. Les gènes schématisés par
des flèches portant la mème couleur appartiennent à une
mème catégorie fonctionnelle de gènes
caractérisés. Les flèches blanches correspondent à
des gènes dont la fonction est inconnue. Le schéma en couleur est
expliqué dans le Tableau I

20
Revue bibliographique
Tableau I : Différents gènes
fonctionnels en fonction des couleurs (Miller et al.,
2003).
|
Catégories des gènes
fonctionnels du
phage T4
|
Gènes
|
|
Transcription (Rouge)
|
asiA, dsbA, goF, modA,
motA, motB, mrh, rpbA, srd,
srh, 33, 55 (alc, alt,
45)
|
|
Traduction (Marron)
|
cef, dmd, modB, regA,
regB, vs, rnlA, rnlB (modA),
tRNAR, tRNAI, tRNAT, tRNAS, tRNAP, tRNAG, tRNAL, tRNAE; rnaC,
rnaD
|
|
Métabolisme de Nucléotides
(Orange)
|
cd, denA, denB, frd,
nrdA,B,C,D,G,H,
nudE, pseT, td, tk, 1,
42, 56
|
|
La réplication de l'ADN, la
recombinaison, la
réparation,
l'emballage (jaune)
|
dda, denV, dexA, repEA,
repEB, rnh, uvsW, uvsX, uvsY,
16, 17, 30, 32,
39, 41, 43, 44, 45, 46,
47, 49, 59, 52, 60,
61/58, 62
|
|
Virions protéiques (bleu)
|
Head: soc, hoc, inh, ipI,
ipII, ipIII, 2, 4, 20,
23, 24, 67, 68,
(22, 21) , Neck: 13, 14
,Tail: 3, 5, 6, 7, 8,
9, 10, 11, 12, 15, 18,
19, 25, 26, 27, 28, 29,
48, 53, 54 ;Tail fiber: wac, 34,
35, 36, 37 (rnlA)
|
|
Chaperons (bleu points)
|
21, 22, 31, 38, 40,
51, 57A, rnlA
|
|
Lyse (Vert)
|
e, rI, rIII, sp, t
(rIIA, rIIB)
|
|
Interactions avec hôte (violet)
|
ac, arn, _-gt, _-gt,
dam, imm, pin, rIIA, rIIB,
stp, (gol, pseT, rnlA)
|
|
Arrêt d'hôte (rose)
|
alc, alt, gol, ndd
(denA, denB, modA, modB)
|
|
Endonuclease (peche)
|
I-TevI-III, mobA-mobE, segA-segG
|
|
Inconnue (blanc ou ~)
|
Inconnu
|
Matériel et Méthodes

21
Matériel et Méthodes
1. Matériel
1.1.Cadre d'étude
La présente étude a été
réalisée à la plateforme de biologie moléculaire de
l'Institut Pasteur de Côte d'Ivoire de Juin 2018 à Octobre 2018 (5
mois).
1.2. Matériel d'étude
1.2.1. Matériel biologique
Le matériel biologie est constitué de 5
bactériophages provenant de différents sites d'isolement. Il
s'est agi de 2 phages isolés dans les eaux lagunaires (lagune
Ebrié) et de 3 phages isolés des micromammifères. Le phage
T4 a été utilisée comme phage contrôle de cette
étude (Tableau II).
1.2.2. Matériel technique, équipement et
consommables
La réalisation de cette étude a
nécessité l'utilisation de plusieurs appareils et consommables
(Tableau III) qui ont permis de réaliser l'extraction
et la purification du matériel génétique ainsi que
l'électrophorèse en champ pulsé (PFGE).
1.2.3. Réactifs et tampons
Divers réactifs et tampons ont été
nécessaires pour l'étude moléculaire (Tableau IV
et V). De plus, l'eau pure utilisée était de
qualité biologie moléculaire, elle a permis de réduire les
variations du pH du milieu réactionnel. Par ailleurs, les tampons et
réactifs mentionnés dans le tableau IV ont permis la
caractérisation génomique (PFGE), tandis que ceux du tableau V
ont été utilisés pour la concentration des phages,
l'extraction du matériel génétique et la digestion
enzymatique à l'aide des enzymes de restriction (XbaI, XmnI, XhoI) dont
les caractéristiques sont contenus dans le Tableau
VI.

22
Matériel et Méthodes
Tableau II: Matériel biologique
|
Phages
|
Origine
|
Année
d'isolement
|
Réference
|
|
p Ebrios
|
Lagune Ebrié
(Cote d'Ivoire)
|
2015
|
Ngazoa-Kakou et al., 2017 ;
Kakou et
al, 2018.
|
|
p A5a
|
Abobodoumé/ Lagune
Ebrié
(Cote d'Ivoire)
|
2017
|
-
N.P
|
|
p M3
|
Décharge Akouedo
(Cote d'Ivoire)
|
2017
|
N.P
|
|
p M7
|
Gbetitapea
(Cote d'Ivoire)
|
2017
|
N.P
|
|
p M11
|
Soko (Cote d'Ivoire)
|
2017
|
N.P
|
|
p T4
|
Univerité Laval
(Canada)
|
1944
|
Szermer-Olearnik et Boratyñski,
2015.
|
N P : Non Publié
Tableau III : Appareils et consommables
|
Désignations
|
Rôles
|
|
V' Cônes stériles
(ART®)
|
|
|
V' Tubes Falcon 15 et 50ml
(WVR®)
|
|
|
V' Alcool 70%, Eau distillée
|
|
|
V' Tube eppendorf 1,5 et 2 mL
|
|
|
V' Béchers 500-1000 mL
|
|
|
Consommables
|
|
Extraction et
|
|
V' Bistouris, Embouts
|
purification
|
|
V' Moules (Bio-Rad Laboratories)
|
|
|
V' Réfrigérateur 4°C, Incubateur
à 50°C
|
|
|
V' Bain marie à 54°C,Microcentrifugeuse
|
|
|
V' Spatule stérile, Scalpel
stérile
|
|
|
V' Générateur avec systèmes PFGE
(CHEF-DR III
|
Caractéristion
|
|
Équipements
|
System) (Bio-Rad)
V' Micropipettes et des cones.
|
génomique,
PFGE
|
|
V' Balance électronique de précision
(BEL)
|
|
|
V' Gel Doc Imager (Bio-Rad)
|
|

23
Matériel et Méthodes
Tableau IV : Réactifs et tampons du
PFGE
|
Désignations
|
Réactifs
|
Quantité/L
|
|
1.0 M Tris, pH 8.0
|
· Tris [Tris hydroxymethyl aminomethane]
· Ajuster le pH à 8.0
· H2O
|
60.57g
350 ml
500 ml
|
0.5 M EDTA, pH 8.0
|
· EDTA
· Ajuster le pH à 8.0
· H2O
|
93.05 g
500 ml
|
Tris-EDTA (TE) buffer, 1X, (10 mM
Tris and 1 mM EDTA, pH
8.0).
|
· 1M Tris, pH 8.0
· 0.5 M EDTA, pH 8
· H2O
|
1 ml
0,2 ml
100 ml
|
Tris-Borate-EDTA (TBE), Buffer, 5X
(0.45 M Tris borate et
0.01 M EDTA).
|
· Tris base
· Acide borique
· 0.5 M EDTA, pH 8.0
· H2O
|
54 g 27,5 g 20 ml 1 L
|
Tris-Borate-EDTA (TBE), Buffer,
0.5X (45 mM Tris borate et
1 mM
EDTA)
|
· 5X TBE
· H2O
|
200 ml
1 L
|
Phage suspension (PS) Buffer (0.1 M
Tris et 0.1 M EDTA, pH
8.0).
|
· 1.0 M Tris, pH 8.0
· 0.5 M EDTA, pH 8.0
· H2O
|
10ml
20ml
100 ml
|
Plug agarose (1.2% SeaKem Gold
Agarose, 1X TE Buffer).
|
· SeaKem Gold Agarose
· 1X TE Buffer
· Chauffer jusqu'à dissolution puis maintenir
à 50 ?C
|
1.2 g
100 ml
|
Phage lysis (PL) buffer (50mM Tris,
50mM EDTA, and 1% (w/v)
SDS).
|
· 1.0M Tris, pH 8.0
· 0.5M EDTA, pH 8.0
· SDS
· H2O
|
5 ml
10ml
1 g
85ml
|
Protéinase K solution, 20 mg/ml.
|
· Protéinase K
· Sterile nuclease-free water
|
20mg
1ml
|
PFGE agarose (1% SeaKem Gold
Agarose, 0.5X TBE).
|
· SeaKem Gold Agarose
· 0.5X TBE Buffer
· H2O
· Chauffer jusqu'à dissolution et refroidir à
50 ?C
|
1,2 g
120 ml
1 L
|
PBS 1X
|
· Un comprimé
|
500 ml
|
· PFGE low range DNA Marker in
http://www.neb.com/; Catalogue
|
|
agarose plugs (New England Biolabs; Ipswich, MA, No. N0350S),
approximately 0.13-194 kb
|
|
|

24
Matériel et Méthodes
Tableau V : Réactifs et Tampons
|
Désignations
|
Fournisseurs
|
Roles
|
|
Bench Top 1 Kb DNA Ladder GeneRuler 1 Kb DNA Ladder Blue 6X
loading dye
Sybr® Safe DNA Gel Strain Ultra TM Agarose
TAE Buffer 10X
|
Promega
Thermo Scientific
Thermo
Scientific
Invitrogèn
Invitrogèn
Promega
|
Réalisation de
l'électrophorèse
standard
|
|
Polyethylene Glycol (PEG) 8000 Phosphate Buffered Saline (PBS)
NaCl (Chlorure de sodium) Alcool Isopropanol 100% Alcool Iso-amylique
Ethanol95%
Acetate de Sodium
Phénol : Chloroforme : Alcool Isoamylique (25 : 24 : 1)
Protéinase K (20 mg/ml) RNase A 4 (mg/ml)
DNase I
|
Promega
Invitrogèn
Promega
Riedl-de Haen
AMRESCO
AMRESCO
Promega
Promega
Roche
|
Concentration des phages et
l'extraction du
matériel
génétique
|
|
XbaI
XmnI
XhoI
Bovine Serum Albumin (BSA)
|
Promega
Promega
Roche
Promega
|
Digestion enzymatique
|
Tableau VI : Les caractéristiques des
endonucléases utilisées
|
Endonucléase
(Source)
|
Sites de restriction
|
Température
|
Temps
d'incubation
|
Origine
|
|
XbaI
(Promega)
|
5'...TCTAG " A...3' 3'...A.?.GATCT...5'
|
37 ° C
|
4 Heure
|
Xanthomonas
badrii
|
|
XmnI
(Promega)
|
5'...GAANN " NNTTC...3' 3'...CTTNN ?. NNAAG...5'
|
37 ° C
|
4 Heure
|
Xanthomonas
campestris pv.
manihotis
|
|
XhoI
(Roche)
|
5'...C " TCGA...3' 3'...G AGCT?.C...5'
|
37 ° C
|
4 Heure
|
Xanthomonas
holcicola
|

25
Matériel et Méthodes
2. Méthodes
2.1. Concentration des bactériophages
2.1.1. Principe
La concentration des phages consiste à concentrer les
virus par l'intermédiaire de deux solutions:
le Polyéthylèneglycol (PEG 8000 à 40 %)
et le NaCl (5 M). Ce mélange va permettre au PEG de former un complexe
avec les cellules phagiques (Phage-PEG) et ce complexe est
précipité par centrifugation après incubation sur glace.
Le PEG est éliminé par le chloroforme pendant que les phages
demeurent intacts. Ensuite les phages sont détruits par du phénol
tandis que l'élimination des protéines phagiques a
été possible par l'ajout du mélange phénol /
chloroforme.
2.1.2. Protocole
Les phages ont été concentrés selon la
méthode de Kumari et al., 2009 avec
quelques
modifications. Ainsi, 3 mL de phages purifiés
(1010-1011 PFU/ mL) sont ajoutés à 400
ìL de NaCl 5 M, et 800 ìL de PEG 8000 à 40 % et
conservés à 4 O C pendant une nuit. Les précipités
recueillis après centrifugation (10 000 rpm / min pendant 10 minutes, 4
O C) sont remis en suspension dans 500 ìL de PBS 1X (pH 7,2). Le
mélange obtenu est traité avec un égal volume de
chloroforme-alcool-isoamylique (24: 1) puis centrifugé à 9000 rpm
/ min pendant 2 minutes ; ce qui permet d'éliminer le PEG 8000 et les
débris cellulaires de la suspension
bactérienne.
2.2. Extraction de l'ADN des phages
2.2.1. Principe
L'extraction de l'ADN est une technique qui permet d'isoler l'ADN
à partir d'une cellule en
quantité et en qualité suffisante pour permettre
son analyse. Elle comporte plusieurs étapes à savoir la
libération de l'ADN et des différents constituants cellulaires
suivie d'une lyse par digestion enzymatique avec la Protéinase K; la
déproteinisation des proteines membranaires par le
Phénol-Chloroforme- Alcool Isoamylique (25:24:1) et la purification
sélective de l'ADN par précipitation avec l'Alcool-Isopropanol
qui aboutit à la formation d'un culot (précipité).
2.2.2. Protocole
Des particules de phages préalablement purifiées
(10-10-1011 PFU / mL) sont traitées avec 20
ìL
de DNase I (1mg / mL) et 15 ìL de RNase A (4 mg / mL)
à 37 O C pendant 30 minutes. Dix microlitres de protéinase K (20
mg / mL) et 50 ìL de SDS (10 %) sont ajoutés à la solution
obtenue, puis le tout est incubée à 56 O C pendant 30 minutes.
Après l'incubation, un volume de 500 ìl de
Phénol-Chloroforme-Alcool Isoamylique (25 : 24 : 1) sont
additionnés au mélange qui est ensuite centrifugé à
10 000 rpm

26

27
Matériel et Méthodes
pendant 2 minutes pour séparer les phases. Cette
opération d'ajout de phénol-chloroforme-alcool isoamylique suivi
de la centrifugation est répétée trois fois. La phase
aqueuse (phase supérieure) est transférée dans un nouveau
micro-tube de 1,5 mL puis 500 ìL d'isopropanol (100 % glacial) et 50
ìL d'acétate de sodium (3 M) y sont ajoutés. Après
une incubation de 10 min sur la glace (pour permettre la précipitation),
le mélange est centrifugé à 14 000 rpm pendant 15 min
à 4 ° C. Le culot d'ADN est lavé deux fois avec 200
ìL d'éthanol à 70% et séché à l'air
ou à 65° C dans le Thermoblock pendant 15 min. L'acide
nucléique est mis en suspension dans 50 ìL de TE (Tris-HCl 10 mM,
pH 7,0, EDTA 1,0 mM, pH 7,0) selon la procédure standard
(Sambrook et al., 1989 ; Sambrook et Russell
2001).
2.3.Dosage fluorimétrique
2.3.1. Principe
Le système Qubit(R) 3.0 utilise la technologie des
sondes fluorophores, qui contiennent des colorants qui ne fluorescent que
liées aux ADN, ARN et protéines. Cette spécificité
permet une quantification précise des acides nucléiques et non de
contaminants. Le système Qubit est sensible et peut être
utilisé même pour de faibles concentrations (10
ìg/ìL).
2.3.2. Protocole
Pour la réalisation du dosage, deux tubes d'analyse ont
été utilisés pour les standards (les étalons) et un
tube pour chaque échantillon. La solution de travail Qubit TM est
préparée en diluant le réactif Qubit TM 1 : 200 dans le
tampon Qubit TM. Un volume de 190 ìL du mélange (réactif
Qubit TM + tampon Qubit TM) est réparti dans les tubes à raison
de 10 ìL pour les solutions d'étalon et 195 ìL pour les
échantillons à doser plus 5 ìL d'extrait d'ADN. Ensuite,
la solution est mélangée au vortex pour une
homogénéisation suivie d'une incubation de 2 minutes à la
température ambiante. Après l'incubation, le test de dosage est
réalisé dans le fluoromètre Qubit® 3.0.
2.4. Digestion
enzymatique
2.4.1. Principe
Les enzymes de restriction sont des endonucléases
capables de reconnaître spécifiquement une courte séquence,
de 4 à 10 pb et de cliver les liaisons phosphodiesters entre 2
nucléotides à l'intérieur de l'ADN double brin au site
reconnu. Ils permettent de fragmenter l'ADN en segments de taille
réduite, ou de le couper à tel ou tel site désiré.
Certains enzymes coupent le site en son milieu et produisent deux fragments
dont les extrémités sont franches. Cependant, la plupart
réalisent une coupure dissymétrique :
Matériel et Méthodes
on parle dans ce cas d'extrémités cohésives
(chaque fragment possède une chaîne qui dépasse l'autre de
quelques bases).
2.4.2. Protocole
L'ADN de bactériophage est mis à digérer par
chaque endonucléase (XbaI, XmnI) et incubé à 37 °C
pendant 4 heures en présence d'un tampon RE 10X buffer et de bovin serun
albumin (BSA) fourni par le fabricant (Tableau VII).
Après incubation, les produits obtenus ont
été séparés sur gel d'agarose à 0,8% dans du
tampon TAE 1X (Promega) contenant 3 ìL de Sybr Safe (10000X)
(Invitrogen) à 100 volt pendant 2 heures.

28
Matériel et Méthodes
Tableau VII : Mélange réactionnel
de la digestion enzymatique
|
Réactifs
|
Quantité
|
|
H2O
|
12,3 jiL
|
|
RE 10X Buffer &
|
2 jiL
|
|
Acetylated BSA, 10 jig! jiL
|
0,2 jiL
|
|
Enzyme de restriction 10U! jiL
|
0,5 jiL
|
|
ADN phage
|
5 jiL
|
|
Température d'incubation
|
37 ° C
|
|
Temps d'incubation
|
4 heures
|
&RE 10X Buffer contient à 1X : pH 7,5
à 37°C ; Tris-HcL 6 mM ; MgcL2 6 mM et NaCL 6 mM.

29
Matériel et Méthodes
2.5. Electrophorèse sur gel d'agarose
Avant la migration, un gel d'agarose à 0,8% est
préparé dans le tampon TAE 1X puis coloré avec 3 ìL
de Sybr Safe (10000X). Un volume de 20 ìL d'extrait d'ADN sont
mélangés à 4 ìL de tampon de charge (colorant Blue
6X). Le mélange obtenu est déposé dans un puit du gel et
soumis à une migration à 100 Volt pendant 2 heures jusqu'à
ce que le front du colorant soit proche du fond du gel. La
révélation s'est faite sous UV sur Gel DOCTM EZ
(BioRad) pour visualiser la présence de bandes d'ADN.
2.6. Electrophorèse en champ pulsé
L'électrophorèse en champ pulsé s'est
déroulée en 4 étapes.
Une étape d'encapsulation des phages dans l'agarose,
une étape de lyse des phages et de libération des génomes,
une étape de digestion enzymatique par les endonucléases et une
étape de migration sur gel d'agarose suivi de l'analyse et de
l'interpretation des résultats (Figure 6).

30
Matériel et Méthodes

Figure 6 : Schéma descriptif de
l'électrophorèse en champ pulsé
1 :Encapsidation des phages dans l'agarose ;
2 : Lyse virale et caqqure des protéines de la capside
avec la protéinase K ; 3 : Digestion enzymatique de
l'ADN avec les endonucléases ; 4 : Migration
électrophorétique et analyse et interpretation des
résultats.

31

32
Matériel et Méthodes
2.6.1. Encapsidation des phages dans l'agarose
Une solution d'agarose est préparée par
mélange de 0,1 g d'agarose (concentration finale à 1% ; Pulsed
Field Certified agarose, Bio-Rad, France) et 10 mL de tampon TBE porté
à 100 °C. Après dissolution complète de l'agarose, la
solution est placée dans un bain à 55 °C pendant 5
minutes.
Parallèlement les phages purifiés sont
placés dans le tampon Phage Suspension (100 mM Tris, 100 mM EDTA, pH
8,0), puis un volume total de 0,5 -1 mL de suspension de phages purifiés
est maintenu dans un bain marie ou bloc chauffant à 50 - 54 °C et
un volume de 400 uL cette suspension de phage est tranférée dans
des tubes eppendorf de 2 mL. Après homogénéisation, 400
ìL de la préparation d'agarose (1% d'agarose) conservée
à 55 °C sont ajoutés et mélangés doucement
avec la suspension phagique maintenue à 50 - 54°C par aspiration et
refoulement avec une pipette sans qu'il n'y ait de bulles d'air. Ce
mélange est ensuite immédiatement réparti dans les moules
à plugs, à raison de 100 ìL par plug. Ceux-ci sont
laissés à température ambiante pour se solidifier ou
à + 4 °C pendant 10 à 15 min (Figure 6).
2.6.2. Lyse virale des phages dans les plugs
d'agarose
Une préparation de 5 mL de tampon de lyse [(Phage Lysis
: 50 mM Tris, 50 mM EDTA, pH 8,0), et SDS à 1 %] additionné de 25
ìL de protéinase K à 20 mg/mL est mis un tube de 5mL. Les
tubes sont ouverts délicatement, les plugs d'agarose sont
rétirés des moules et transférés dans les tubes
correspondants. Ces tubes sont alors placés dans un bain-marie à
54 °C pendant 2 heures. Le tampon de lyse est soigneusement
éliminé des tubes à l'aide d'une pipette par aspiration.
Les plugs sont lavés avec 5 mL de tampon TE (10 mM Tris, 1 mM EDTA, pH
8,0) stérile qui est chauffé à 54 °C pendant 15 min
dans un bain d'eau sous faible agitation. Les tubes contenant les plugs sont
rétirés du bain-marie, et le tampon est aspiré
soigneusement en veillant à ce que les plugs ne soient pas
cassés. Cette opération de lavage avec le tampon TE est
répétee trois fois et deux fois avec de l'eau. Cependant le
tampon est changé après chaque lavage. Les plugs sont alors
stockés à +4 °C jusqu'à ce qu'ils soient prêts
à être soumis à digestion par les enzymes de restriction
(Figure 6).
2.6.3. Digestion enzymatique du génome
Les plugs d'agarose sont soumis à une
pré-digestion selon le mélange réactionnel
(Tableau VIII) avant de réaliser une digestion
enymatique proprement dite selon le mélange réactinnel
(Tableau IX) dans un bain marie à 37 °C pendant 2
heures. Les plugs de phages sont rétirés du tampon TE avec un
petit spatule plate désinfecté à l'éthanol,
coupés à l'aide d'un bistouri ou une lame de scalpel, le long de
l'axe des bouchons en faisant des tranches d'environ 2 mm de la longueur prise.
Les portions restantes
Matériel et Méthodes
des bouchons sont stockés à 4°C dans le
tampon TE. Les plugs découpés sont placés dans un volume
de 200 uL de tampon de restriction dilué à 1X de concentration
pour une pré-digestion (Tableau VIII), puis
incubés à 37°C pendant 10 à 15 min à la
température ambiante. Après l'incubation, le tampon est
aspiré à l'aide d'une micropipette et 200 uL de master mix de
digestion (Tableau IX) est ajouté à chaque tube
puis incubé à la température appropriée de l'enzyme
XbaI ou XhoI pendant 2 heures. Après l'incubation, le tampon de
restriction est aspiré puis les plugs découpés sont mis en
suspension dans 200 uL de TBE 0,5X pendant 5 min (Figure
6).
2.6.4. Électrophorèse sur gel
d'agarose
Avant la migration, un gel d'agarose de 1% est
préparé dans un tampon de TBE 0,5X puis conservé au bain
mari à à 55 °C. Ensuite, la cuve
d'électrophorèse en champ pulsé CHEF-DRIII system (Bio-Rad
Laboratoires) est remplie avec 2,2 litres de tampon 0,5X et l'automate est
laissé allumer jusqu'à ce que la température de la cuve de
migration passe à 14°C. Parallèlement les plugs
découpés sont déposés le long du peigne. On laisse
refroidir entre 45-50 °C et le gel préparé est
conservé à 55 °C avant de le couler dans la direction de la
longueur de l'axe. Après que le gel soit solidifié avec les plugs
sur le peigne, on rétire le peigne puis on le dépose dans la cuve
électrophorèse. L'électrophorèse est
réalisée à 14°C avec un voltage à 6 V/cm,
pendant 6 heures avec un temps de pulsation de 2,0-12,0 sec, et un angle de
120° (des temps d'impulsion peuvent être nécessaires pour
optimiser la séparation des bandes) (Figure 6). Une
fois la migration terminée, le gel est immergé dans une solution
de sybr safe 10000X (40 uL dans 400 mL d'eau déionisée) pendant
30 min sous une faible agitation. Le gel est ensuite décoloré
dans 500 mL d 'eau désionisée pendant 20 min et à chaque
10 min l'eau désionisée est changée. Le gel est
examiné à l'aide d'un automate appélé GelDoc
(BioRad Laboratoires) couplé à un ordinateur puis capturé,
imprimé et enregistré l'image du gel coloré avec un
système de documentation de gel.

33
Matériel et Méthodes
Tableau VIII : Mélange réactionnel
de la pré-digestion enzyamtique du PFGE
|
Réactifs
|
Volume de réactifs par plug
(bouchon) en
ìL
|
Volume de réactifs pour 10
plugs (bouchon) en
ìL
|
|
H20
|
180
|
1800
|
|
Buffer 10X Enzyme
|
20
|
200
|
|
Volume Total
|
200
|
2000
|
Tableau IX: Mélange réactionnel de
la digestion enzyamtique du PFGE
|
Réactifs
|
Volume de réactifs par plug
(bouchon) en
ìL
|
Volume de réactifs pour 10
plugs (bouchon) en
ìL
|
|
H20
|
173
|
1730
|
|
Buffer 10X Enzyme
|
20
|
200
|
|
BSA 10 ìg / ìL
|
2
|
20
|
|
XbaI ou XhoI 10 U/ ìL
|
5
|
50
|
|
Volume Total
|
200
|
2000
|
Matériel et Méthodes
2.7. Analyse des profils de restriction
Les analyses de profils PFGE ont été
réalisées grâce au logiciel Bionumerics® (Applied
Maths). Les images des gels d'électrophorèse ont
été normalisées après que les bandes de restriction
du marqueur de poids moléculaire aient été
identifiées.
2.7.1. Evaluation de la similarité des
profils
La dissemblance entre 2 génotypes est quantifiée
par le calcul du coefficient de Jaccard. Il permet d'estimer le degré de
dissemblance entre deux profils de restriction par le calcul du pourcentage de
bandes communes sur le nombre total de bandes (Jaccard, 1908).
Le cofficient de similarité de Jaccard entre deux profils de restriction
i et j est calculé comme suit :
|
nij est le nombre de bandes communes entre les deux profils i et
j, ni est le nombre de bandes du profil i,
nj est le nombre total de bandes du profil j.
|
|
clij= nij
nij+ni+nj
|
Le coefficient de Jaccard varie de 0 à 100%, un
coefficient égal à 100% signifiant que les profils de restriction
sont identiques. Le coefficient de Jaccard entre 2 profils est ajusté
grâce à des paramètres d'optmisation et de
tolérance. Lorsque les profils i et j sont comparés, deux bandes
sont considérées comme identiques si la position d'une bande du
profil i est comprise dans une fenêtre autours de la bande du profil j.
La taille de cette fenêtre correspond à la tolérance. Le
critère d'optimisation correspond ici au même principe en prennant
en compte le positionnement des profils entiers les uns par rapport aux autres.
La tolérance et l'optimisation sont chacun fixées à 2%.
2.7.2. Analyse phylogénétique
Les liens de parentés entre les isolats sont
établis par construction de dendrogrammes. Ceux-ci sont construits par
classification hiérarchique selon la méthode Unweight Pair Group
Method with Arithmetic mean (UPGMA) qui fait l'hypothèse d'une
évolution indépendante des différentes lignées
à une vitesse constante, avec 1000 répétitions.
2.7.3. Attribution de pulsotype et de cluster

34
Il s'agit d'identifier les différents profils de
restriction grâce au seuil de tolérance défini
précédemment. Dans cette étude, deux profils ayant au
maximum 2% de différence, seront considérés comme
identiques et appartenant au même pulsotype..
Résultats et Discussion
35

Résultats et Discussion
1. Résultats
1.1. Analyse du profil génomique des phages par les
endonucléases sur gel d'agarose
L'analyse génomique par la digestion enzymatique montre
que les phages p T4, p Ebrios, p M3 et p M7 sont des phages à ADN double
brin car on a une apparition des bandes sur le gel d'agarose (Figure
7A, Annexe 4).
Les phages p Ebrios et p A5a provenant des eaux lagunaires sont
des phages à ADN, et montrent un profil après digestion aux
enzymes XbaI et XmnI (Figure 7, Annexe 4).
Les phages p M3, p M7 et p M11 issus de mammifères
montrent des profils différents. Ainsi, p M3 et p M7 sont des phages
à ADN, tandis que p M11 n'a revelé aucune bande après
digestion (Annexe 4). Le phage p T4 a été
positif pour la digestion aux enzymes de restrictions (Figure 7 et
Annexe 4 ; Tableau XI).
Les concentrations d'ADN obtenus après l'extraction, la
purification ont varié de 8,41 à 445 ng/uL, avec une moyenne de
276,18 ng/uL (Tableau XI).
36

Résultats et Discussion

Figure 7: Profil des phages après
digestion par des endonucléases
M : marqueur de poids moléculaire
GeneRuler 1 kb DNA Ladder;
(A) : Profil de digestion par XbaI :
Puits 1 : (p T4c ; Puits 2 : (p T4 b ; Puits 3 : (p A5a ;
Puits 4 : (p M3 ; Puits 5 : (p M7 ; Puits 6 : (p M11.
(B) : Profil de digestion par XmnI :
Puits1: (p Ebrios ; Puits 2 : (p A5a ; Puits 3 : (p M3 ; Puits
4 : (p M7 ; Puits 5 : (p M11.
Tableau X : Détermination du profil
génomique des phages
|
Désignation du
Phage
|
Concentration des
Acides
Nucléiques
(ng/uL)
|
Digestion enzymatique
(XbaI/XmnI)
|
Type Génome
(ADN/ARN)
|
|
(p T4c
|
46,90
|
+/+
|
dsDNA
|
|
(p T4b
|
8,41
|
+/+
|
dsDNA
|
|
(p Ebrios
|
265
|
+/+
|
dsDNA
|
|
(p A5a
|
369
|
+/+
|
dsDNA
|
|
(p M3
|
445
|
+/+
|
dsDNA
|
|
(p M7
|
356
|
+/+
|
dsDNA
|
|
(p M11
|
443
|
-/-
|
ND
|
+/+ : Profil de restriction positif d'une
digestion enzymatique par XbaI et XmnI, -/- : Profil de
restriction négatif de la digestion enzymatique par XbaI et XmnI.
dsDNA : ADN double brin,
ssDNA : ADN simple brin,
ssRNA : ARN simple brin, ND : Non
Déterminé
37

Résultats et Discussion
1.2.Analyse du profil génomique par la
méthode en champ pulsé
1.2.1. Analyse du profil génomique
généré par XbaI
Après la digestion des phages avec l'enzyme XbaI, les
résultats ont montré une diversité entre les 5 phages de
cette étude (Figure 8, Annexe 2), 10 bandes au niveau
du phage ö M7, 9 bandes pour les phages ö T4c et ö M11 ont
été observées. Par contre avec les phages ö Ebrios et
ö A5a, aucune bande n'a été observée.
Une analyse par le logiciel BioNumérics
(Applied Maths vesion 7.6 de 2017) a permis l'obtention de dendogramme
(Figure 9).
38

Résultats et Discussion

Figure 8 : Électrophorèse en champ
pulsé de bactériophage digéré par l'enzyme de
restriction XbaI
M1 : marqueur de poids moléculaire
spécifique du PFGE (0.13-194 kb) ; M : marqueur de
poids moléculaire GeneRuler 1 kb DNA Ladder;
Puits 1 : p T4b ; Puits 2 : p
T4c ; Puits 3 : p A5a ; Puits 4 : p A5a ;
Puits 5 : p Ebrios ; Puits 6 : p M3 ; Puits
7 : p M3 ; Puits 8 : p M7, Puits 9
: p M7 ; Puits 10: p M11 ; Puits 11:
p M11.

Figure 9 : Dendrogramme
généré par le logiciel Bionumerics® avec l'enzyme
XbaI
Elle montre la distance calculée par l'indice de
similarité de Jaccard, des profils PFGE des phages isolés par
l'enzyme XbaI. Le degré de similarité (%) est indiqué.
39

Résultats et Discussion
1.2.2 Analyse du profil génomique
généré par XhoI
Après l'analyse du profil de restriction de l'enzyme
XhoI (Figure 10, Annexe 3), 16 bandes au niveau du phage p
M11, 15 bandes pour le phage p T4, 14 bandes pour les phages p M3 et p M7 et
enfin 13 bandes pour le phage p A5a ont été observées.
Cependant aucune bande n'a été observée au niveau du phage
Ebrios. Le profil PFGE des deux enzymes XbaI et XhoI a montré que les
phages p T4, p A5a, p M3, p M7et p M11 ont tous une taille supérieure
à 164 Kb.
Les pulsotypes générés par XbaI
présentant un dendrogramme sur la Figure 9 ont
montré entre eux des similarités comprises entre 73 et 99 % ;
ceux générés par XhoI présentés sur la
Figure 11 ont présenté entre eux des
similarités comprises entre 40 et 96 %. Cela implique que les profils de
restrictions des phages sélectionnés sont différents entre
eux.
40

Résultats et Discussion
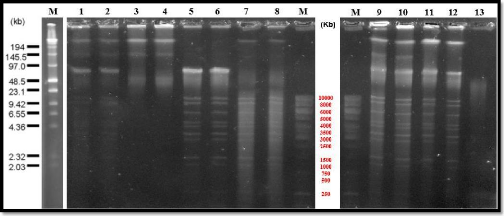
Figure 10 : Électrophorèse en
champ pulsé des génomes de bactériophages après
digestion par XhoI
M1 : marqueur de poids moléculaire
spécifique du PFGE (0.13-194 kb) et M2 : marqueur de
poids moléculaire 1 kb ; Puits 1: (p T4c ; Puits
2: (p T4c ; Puits 3: (p T4 b ; Puits
4: (p T4 b ; Puits 5: (p A5a ; Puits
6: (p A5a ; Puits 7: (p M3 ; Puits
8: (p M3 ; Puits 9: (p M7 ; Puits
10: (p M7 ; Puits 11: (p M11 ; Puits
12: (p M11 ; Puits 13: (p Ebrios.

Figure 11 : Dendrogramme
généré par le logiciel Bionumerics® avec l'enzyme
XhoI
Elle montre la distance calculée par l'indice de
similarité de Jaccard, des profils PFGE des génomes de phages
fragmentés par XhoI. Le degré de similarité (%) est
indiqué.
41

Résultats et Discussion
1. Discussion
? L'analyse génomique des phages par les enzymes
de restriction
La digestion enzymation a permis de mettre en evidence les
différences entre les phages étudiés. Ainsi, une absence
de bande d'ADN se justifirait soit par l'incapacité de digérer
l'ADN des phages par les enzymes XmnI et XbaI qui pourrait être due
à l'absence de sites cibles pour l'enzyme de restriction testée,
soit par une perte de fragment de petites tailles lors de
l'électrophorèse, soit par une faible concentration d'ADN
où encore lors de l'extraction de l'ADN génomique. Cette
hypothèse a été rapportée par certains auteurs qui
pourrait être probablement due à une dégradation de l'ADN
durant le processus (Wong et al., 2004) ou à
sa méthylation (Xydas et al., 1996).
En effet, l'analyse du profil de restriction des phages ö
T4 et ö Ebrios a montré que leurs génomes sont
supérieurs à 10000 Kbp. Le matériel génétque
de ces phages est du type ADN double brin. Ces résultats confirment des
travaux antérieurs pour les phages ö T4 et ö Ebrios avec des
génomes respectifs de 168903 bp et 39 752 pb (Szermer-Olearnik
et Boratyñski, 2015 ; Ngazoa-Kakou et al., 2018). Par
contre les phages ö A5a, öM3, öM7 montrent des profils dont le
génome est également supérieur à 10000 Kbp.
Plusieurs études ont montré la caractérisation
moléculaire grace aux enzymes de restriction des phages à
Pseudomonas aeruginosa (Kumari et al., 2009
, Essoh et al., 2015), Salmonella
enterica (Lappe et al., 2009), et klebsiella
pneumoniae (Hsu et al., 2013).
Enfin, la non-reconnaissance des sites de restriction de
l'enzyme démontre la limite de la méthode pour les phages
à ADN double brin.
? L'analyse génomique en champ
pulsé
L'électrophorèse en champ pulsé est une
électrophorèse sur gel d'agarose utilisant des champs
électriques alternés. Les changements de direction des
molécules en fonction de leur structure tridimensionnelle durant la
migration font varier leur distance totale de migration (Lai et
al., 1989). L'électrophorèse en champ pulsé
sur gel d'agarose a connu un important essor parmi les techniques
d'étude des génomes (Safa et al.,
2005). Cette technique utilise des enzymes de restrictions pour
générer des profils typiques à chaque souche
bactérienne où phagique. L'utilisation des enzymes
supplémentaires dans le PFGE permet d'offrir une meilleure
discrimination possible des modèles les plus faciles à
interpréter d'où l'intérêt pour nous d'utiliser le
XbaI et le XhoI.
Les résultats obtenus dans cette étude sont
similaires aux travaux antérieurs de Clokie et Kropinski,
2009, qui a utilisé les gros coliphages purifiés dans
l'optimisation de l'électrophorèse en champ pulsé sur gel
d'agarose. En effet, les résultats obtenus par le PFGE ont
été analysés par le logiciel Bionumerics®, il nous a
permis d'estimer la taille du génome des phages ö A5a, ö M3,
ö M7, ö M11 et
42

Résultats et Discussion
ö T4 qui est supérieure à 164 Kb. Le phage
Ebrios n'a aucune bande concernant les deux enzymes de restrictions
utilisées. Cela s'expliquerait par une absence totale de
molécules d'ADN, ce qui signifierait que les molécules d'ADN ont
été dégradées ou détruites où soit
par une insuffisance de molécules d'ADN due à la méthode
de concentration et de purification des phages utilisant le PEG et le NaCl.
Cette absence de bande pourrait être due
également par une incapacité de l'endonucléase a
dégradé la molécule d'ADN où encore une absence de
sites de restriction. Cette notion d'absence de bande a été
rapportée par certains auteurs qui pourrait être probablement
dû à une dégradation de l'ADN durant le processus
(Wong et al., 2004) ou à sa méthylation
(Xydas et al., 1996).
En effet, des différences entre les phages ont
été obtenues par les profils de restriction PFGE par rapport
à la méthode de digestion enzymatique. Le PFGE a un pouvoir
discriminant plus élevé que la digestion enzymatique. De plus, la
PFGE peut s'avérer longue à mettre en oeuvre et souffre d'un
manque de reproductibilité inter- (voire intra-) laboratoires. Ainsi, le
pouvoir discriminant du PFGE a été soutenu par (Hsu
et al., 2013 ; Czajkowski et al., 2015 ; Krasowska et al.,
2015 ; Wang et al., 2016) caractérisant le
génome de nouveaux phages lytiques comme candidats potentiels pour la
phagothérapie et le biocontrôle des aliments en industrie
agroalimentaire.
D'autres travaux antérieurs ont étudié
les différents facteurs influençant les résultats de
l'électrophorèse en champ pulsé, incluant la
préparation des plugs d'agarose, la lyse des cellules bactérienne
ou phagique, la digestion enzymatique (Mulvey et al., 2001 ;
Murchan et al., 2003 ; Zhang et al., 2007), ainsi
que les paramètres électrophorétiques à savoir le
temps total de migration, influençant la distribution des fragments de
restriction dans le gel et donc influence le pouvoir discriminatoire de la
technique
Conclusion et Perspectives

43
Conclusion et Perspectives
Ce travail a permis de caractériser 5 phages à
savoir ö Ebrios, ö A5a, ö M3, ö M7, ö M11 par la
digestion enzymatique et l'électrophorèse en champ pulsé,
de produire des élements de connaissance sur le type de matériel
génétque de ses phages ainsi que la taille du génome par
des profils de restriction. L'analyse de ses profils obtenus a permis de mettre
en evidence des différences entre les phages. Cela a permis de
démontrer la diversité génétique entre les phages
étudiés.
Il en résort que le PFGE s'est avéré
être une méthode plus sensible que la digestion enzymatique pour
la caractérisation des phages au vue des résultats
rapportés. Il a révelé plus de bandes discriminantes que
la digestion enzymatique. Le PFGE et la digestion enzymatique sont deux
méthodes dépendantes. La digestion enzymatique des phages est
simple et rapide et pourrait être excellente dans la mise en oeuvre en
première intention de la discrimination des contraintes.
En définitive, les profils de restriction du PFGE et de
la digestion enzymatique permettent de comparer plusieurs échantillons ;
cependant, ils ne donnent malheureusement pas accès à
l'information génétique en tant que telle.
En perspectives, il serait intéressant de
poursuivre ce travail de recherche afin de réaliser l'étude des
gènes d'intéret par PCR suivie de séquençage
à haut débit (NGS) afin de fournir de précieuses
informations sur la diversité de ces gènes.
Ce type d'information pourra à long terme permettre de
réaliser la métagénomique afin d'avoir une meilleure
compréhension sur la diversité génétique ainsi que
la composition de ses phages.
Le monde étant actuellement confronté aux
infections nosocomiales difficiles à traiter, les bactériophages
représentent une alternative intéressante et un potentiel
médicinal naturel offrant une panoplie d'applications relativement peu
coûteuses, permettant de répondre en toute sécurité
à la lutte antibactérienne.
Il serait important de caractériser des phages comme
candidats potentiels à la phagothérapie.
.
Références bibliographiques
44

45

Références bibliographiques
Ackermann H. W. & DuBow M. S., 1987.
Viruses of Prokaryotes: General properties of bacteriophages. CRC
Press, p. 1, 18, 29-32, 46, 51-53.
Ackermann H. W., 2003. Bacteriophage
observations and evolution. Research in Microbiology ; 154
(4):245-51.
Ackermann H. W., 2004. Les
bactériophages entrent dans le 3e millénaire. Virologie
; 8 : p 409-12. Ackermann H. W., 2011. Bacteriophage
taxonomy. Microbiology Australia; 32 (2): p. 90-94.
Anas E., 2011. Phagothérapie
Passé Et Avenir (Nouveaux faits et procédures pour une
réhabilitation). Sante Publique - Profession Médicale ;
Mémoire de Thèse Numéro P0852011. Université
Mohammed V Faculté De Médecine Et De Pharmacie, Rabat, Maroc, 107
p.
Baguy O. M., Guessennd K. N., Coulibaly N. D., Saraka
N. D., Coulibaly K. J., Koffi-Nevry R., Ouattara G. D., Gbonon V., Tiekoura K.
B. & Dosso M., 2014. First Report of Qnr Genes in Multidrugs
Resistant (ESBL) Enterobacteria Isolated from Different Ecosystems in Abidjan,
Ivory Coast. International Journal of Biological Sciences and Applications.
Vol. 1, No. 4, pp. 170-175.
Bardy P., Pantucek R., Benesýk M. & Doskar
J., 2016. Genetically modified bacteriophages in applied
microbiology. Journal of Applied Microbiology ; 121: 618-633.
Berdjeb L. & Jacquet S., 2009. La
viriosphère : quelle place dans le fonctionnement et l'évolution
des écosystèmes aquatiques (partie 2) ? Virologie, 13 :
189-99.
Boulanger P., 2009. Purification of
bacteriophages and SDS-PAGE analysis of phage structural proteins from ghost
particles. In : Clokie, M.R.J. and Kropinski, A.M. (eds.). Bacteriophages:
Methods and Protocols, Volume 2: Molecular and Applied Aspects. United
Kingdom: Humana Press, p. 227 - 238.
Brussow H., Canchaya C. & Hardt W. D.,
2004. Phages and the evolution of bacterial pathogens: from genomic
rearrangements to lysogenic conversion. Microbiology Molecular Biology
Review 68: 560-602.
Canchaya C., Fournous G., Chibani-Chennoufi S.,
Dillmann M. L. & Brussow H., 2003. Phage as agents of lateral gene
transfer. Current Opinion in Microbiology, 6 : 417-424.
Carminati A., Kerkour N., Landry C., Lopes B.L. &
Meyer F., 2016. Phagothérapie : une alternative à
l'antibiothérapie ? Revue Médicale Suisse, 12 :
1676-1677.
Carvalho C. M., Gannon B. W., Halfhide D. E., Santos
S. B., Hayes C. M., Roe J. M. & Azeredo J. 2010. "The in vivo
efficacy of two administration routes of a phage cocktail to reduce numbers of
Campylobacter coli and Campylobacter jejuni in chickens".
BMC microbiology ; 10 : 232p.
Castel G., Heyd B. & Tordo N., 2009. Le
phage display : une nouvelle arme pour la recherche antivirale. Virologie
;13 (2) :93-102.
Références bibliographiques
Ceyssens P. J., Glonti T., Kropinski N. M., Lavigne
R., Chanishvili N. & Kulakov L., 2011. Phenotypic and genotypic
variations within a single bacteriophage species. Virologie Jorunal ;
8: 134.
Chambon P., 2006. La filière
géorgienne des virus qui soignent. Science & Vie ;
n°1060 : 117-106. Choi J., MeherKotay S. & Goel R., 2011.
Bacteriophage-based biocontrol of biological sludge bulking in waste
water. Bioengineered Bugs; 2(4): 214-217.
Clokie M. R. J. & Kropinski A. M., 2009.
Bacteriophages: Methods and Protocols, Volume 2: Molecular and Applied
Aspects, Methods in Molecular Biology vol. 502: Humana Press, a
part of Springer, 379 p.
Clokie M. R. J., Andrew D., Millard, Andrey V.,
Letarov & Shaun H., 2011. Phage in nature. Landes Bioscience
Review Bacteriophage ;1(1) :31-45.
Comeau A. M. & Krisch H. M., 2008. The
capsid of the T4 phage superfamily: the evolution, diversity, and structure of
some of the most prevalent proteins in the biosphere. Molecular Biology and
Evolution; 25: 1321-32.
Conway J. F., Duda R. L., Cheng N., Hendrix R. W.
& Steven A. C., 1995. Proteolytic and conformational control of
virus capsid maturation: the bacteriophage HK97 system. Journal of
Molecular Biology; 253 (1) :86-99.
Coombs D. H. & Arisaka F., 1994. T4 tail
structure and function. In Molecular Biology of Bacteriophage T4. (J. D.
Karam, ed.). American Society for Microbiology, Washington, DC pp
259-281.
Czajkowski R., Ozymko Z., De Jager V., Siwinska J.,
Smolarska A., Ossowicki A., Narajczyk M. & Lojkowska E., 2015.
Genomic, Proteomic and Morphological Characterization of Two Novel Broad Host
Lytic Bacteriophages ÖPD10.3 and ÖPD23.1 Infecting Pectinolytic
Pectobacterium spp. and Dickeya spp. PLoS ONE; 10 (3):
e0119812. doi :10.1371/journal. Pone.0119812.
D'Hérelle F., 1919. Sur le rôle
du microbe bactériophage dans la typhose aviaire. CR de
l'Académie des Sciences, Paris ; 169 : 932-3 p.
D'Hérelle F., 1926. Le
bactériophage et son comportement. Edition Masson et Cie ;
Paris :160-181p. D'Hérelle, F., 1917. Sur
un microbe invisible antagoniste des bacilles dysentériques. Comptes
Rendus de l'Académie des Sciences ; Paris 165 : 373-375.
Davis B. M. & Waldor M. K., 2003.
Filamentous phages linked to virulence of Vibrio cholerae. Current
opinion Microbiology; 6(1):35-42.
Diemer G. S. & Stedman K. M., 2012. A
novel virus genome discovered in an extreme environment suggests recombination
between unrelated groups of RNA and DNA viruses. Biology Direct ; 7 :
13.
46

Références bibliographiques
Drouji S., 2009. Development of
bacteriophages: features and applications. Sante Publique-Profession
Médicale ; Mémoire de Thèse Numéro P0692009.
Université Mohammed V Faculté de Médecine et de Pharmacie
Rabat, Maroc, 117p.
Drulis-Kawa Z., Majkowska-Skrobek G., Maciejewska B.,
Delattre A. S. & Lavigne R., 2012. Learning from
bacteriophages-advantages and limitations of phage and phage-encoded protein
applications. Current Protein and Peptide Science ;13 (8) :699-722.
Dublanchet A. & Fruciano E., 2008.
Brève histoire de la phagothérapie. Médecine
et maladies infectieuses ;38 (8) :415-420p.
Dublanchet A. & Patey O., 2011. La
phagothérapie : passé et avenir (faits nouveaux et
procédure (s) pour une réhabilitation). Immunology- analyse
& Biologie Spécialisée ;26 : 165-175.
Dublanchet A., 2009. Des virus pour combattre
les infections : la phagothérapie, renouveau d'un traitement au secours
des antibiotiques. Edition Favre : 247 p.
Dublanchet A., 2014. Qu'est-ce que la
phagothérapie ? Hegel Vol. 4 N° 4 : 354-370p.
Edgar R., Friedman N., Molshanski-Mor S. & Qimron
U., 2012. Reversing bacterial resistance to antibiotics by
phage-mediated delivery of dominant sensitive genes. Applied and
Environmental Microbiology; 78 (3): 744-751.
Effantin G., 2005. Études par
Cryo-microscopie électronique de la structure de virus : application aux
bactériophages Gifsy-2 de Salmonella Typhimurium et T5 d`
E.Coli. Thèse de Doctorat Biophysique [physics.bio-ph].
Université Joseph-Fourier- Grenoble, France ; 190p.
Ehui P. J., Sekongo K. W. & Kouassi F. K., 2017.
L'adhesion Du Personnel Soignant Ivoirien A La Phagotherapie.
International Journal Of Law, Education, Social And Sports Studies
Vol.4.Issue.4 :10p.
Essoh C., Blouin Y., Loukou G., Cablanmian A., Lathro
S. & Kutter E., 2013. The Susceptibility of Pseudomonas
aeruginosa Strains from Cystic Fibrosis Patients to Bacteriophages.
PLoSOne 8: e60575.
Filee J., André M. C., Curtis A. S. & Henry
M. K., 2006. Les bactériophages de type T4 : des composants
prépondérants de la « matière noire » de la
biosphère. Médecine-Science (Paris); 22(2): p.
111-112.
Filee J., Tetart F., Suttle C. A. & Krisch H. M.,
2005. Marine T4-type bacteriophages, a ubiquitous component of the
dark matter of the biosphere. Proceedings of the National Academy of
Sciences of United States of America; 102:12471-6.
García P., Martínez B., Rodríguez
L. & Rodríguez A., 2010. Synergy between the phage
endolysin LysH5 and nisin to kill Staphylcoccus aureus in pasteurized
milk. International Journal of Food Microbiology; Issue 3;151-155.
47

48

Références bibliographiques
García P., Rodríguez L.,
Rodríguez A. & Martínez B., 2010. Food
biopreservation: promising strategies using bacteriocins, bacteriophages and
endolysins. Trends in Food Science & Technology; 21 (8): p.
373-382.
Gill J., & Hyman P., 2010. Phage choice,
isolation, and preparation for phage therapy. Current Pharmaceutical
Biotechnology; 11, (1): 2-14.
Gorski A., Miedzybrodzki R., Borysowski J.,
Weber-Dabrowska B., Lobocka M., Fortuna W., Letkiewicz S., Zimecki M. &
Filby G., 2009. Bacteriophage therapy for the treatment of infections.
Current Opinion in Investigational Drugs; 10 (8): 766-74.
Hankin E. H., 1896. L'action
bactéricide des eaux de la Jumna et du Gange sur le vibrion du
choléra, Annales de l'Institut Pasteur ;10 : 511-523p.
Hershey A. D. & Chase M., 1952.
"Independent functions of viral protein and nucleic acid in growth of
bacteriophage. The journal of general physiology; 36 (1):
39-56.
Hsu C., Lin T., Pan Y., Hsieh P. & Wang J.,
2013. Isolation of a Bacteriophage Specific for a New Capsular Type of
Klebsiella pneumoniae and Characterization of Its Polysaccharide
Depolymerase. PLOS ONE ; 8 (8) : e70092.
Inal j., 2003. Phage therapy: a reappraisal
of bacteriophages as antibiotics. Archivum immunologiae et therapiae
experimentalis-english edition; 51 (4): 237-44.
Jaccard P., 1908. Nouvelles recherches sur la
distribution florale. Bulletin de la Société Vaudoise des
Sciences Naturelles ;44: 223-270.
Kanamaru S., Leiman P. G., Kostyuc henko V. A.,
Chipman P. R., Mesyanzhinov V. V., Arisaka F. & Rossmann M. G., 2002.
Structure of the cell puncturing device of bacteriophage T4.
Nature; 415: p. 553-557.
Kikuchi Y. & King J., 1975. Genetic
control of bacteriophage T4 baseplate morphogenesis. III. Formation of the
central plug and overall assembly pathway; Journal of Molecular Biology
;99 (4):716- 695.
King J., 1968. Assembly of the tail of
bacteriophage T4. Journal of Molecular Biology; 32(2):231-262.
Krasowska A., Biegalska A., Augustyniak D., £ooe M., Richert M.
& £ukaszewicz M., 2015.
Isolation and Characterization of Phages Infecting
Bacillus subtilis. BioMed Research
International :10 p.
http://dx.doi.org/10.1155/2015/179597.
Kumari S., Harjai K. & Chhibber S., 2009.
Characterization of Pseudomonas aeruginosa PAO Specific
Bacteriophages Isolated from Sewage Samples. American Journal of Biomedical
Sciences; 1(2): 91-102.
Kutateladze M. & Adamia R., 2008. Phage
therapy experience at the Eliava Institute. Médecine et Maladies
Infectieuses ; 38(8):426-30.
Références bibliographiques
Kutter E., De Vos D., Gvasalia G., Alavidze Z.,
Gogokhia L., Kuhl S. & Abedon S. T., 2010. Phage therapy in
clinical practice: treatment of human infections. Current Pharmaceutical
Biotechnology; 11, (1), 69-86.
Kutter E., Raya R. & Carlson K., 2005.
Molecular mechanisms of phage infection. Bacteriophages: Biology and
applications. Boca Raton, USA, CRC Press: 163-220
p.
Lai E., Birren B. W., Clark S. M., Simon M. I. &
Hood L., 1989. Pulsed-Field Gel Electrophoresis techniques for
separating 1 to 50 Kilobase DNA fragments. Analytical
Biochemistry;7: 34-42.
Lappe N. De., Doran G., O'Connor J., O'Hare C. &
Cormican M., 2009. Characterization of bacteriophages used in the
Salmonella enterica serovar Enteritidis phage typing scheme.
Journal of Medical Microbiology; 58:86-93p.
Lebaron P. & Nicolas J. C., 2003. Le
virioplancton : des virus essentiels au bon fonctionnement de l`océan et
au maintien de la biodiversité. Virologie; 7(2) :125-139.
Liu M., Deora R., Doulatov S. R., Gingery M.,
Eiserling F. A., Preston A., Maskell D. J., Simons R. W., Cotter P. A.,
Parkhill J. & Miller J. F., 2002. Reverse transcriptase-mediated
tropism switching in Bordetella bacteriophage. Science; 295 (5562):
2091-2094.
Loc-carrillo C. & Abedon S., 2011. Pros
and Cons of phage therapy. Bacteriophage 1: 111-14.
Lu T. K. & Collins J. J., 2009.
Engineered bacteriophage targeting gene networks as adjuvants for
antibiotic therapy. Proceedings of the National Academy of Sciences of the
United States; 106 (12): 4629-4634.
Lurz R., Orlova E. V, Gunther D., Dube P., Droge A.,
Weise F., Van Heel M. & Tavares P., 2001. Structural organization
of the head-to-tail interface of a bacterial virus. Journal of Molecular
Biology; 310 (5):1027-1037.
Marston M. F. & Sallee J. L., 2003.
Genetic diversity and temporal variation in the cyanophage community
infecting marine Synechococcus species in Rhode Island's coastal waters.
Applied Environmental Microbiology; 69: 4639-47.
Miller E. S., Kutter E., Mosig G., Arisaka F.,
Kunisawa T. & Ruger W., 2003. Bacteriophage T4 Genome.
Microbiology and molecular biology reviews; Vol. 67, No. 1: p.
86-156.
Mulvey M. R., Chui L., Ismail J., Louie L., Murphy C.,
Chang N. & Alfa M., 2001. Development of Canadian standardized
protocol for subtyping methicillin-resistant Staphylococcus aureus
using Pulsed-Field Gel Electrophresis. Journal of Clinical
Microbiology; 39: 3481-3485.
Murchan S., Kaufmann M. E., Deplano A., Ryck R.,
Struelens M., Zinn C. E., Fussing V., Salmenlinna S., Vuopio-Varkila J., Solh
N. E., Cuny C., Witte W., Tassios P. T., Legakis N., Leeuwen W., Belkum A.,
Vindel A., Lacoucha I., Garaizar J., Haeggman S., Olsso-Liljequist B., Ransjo
U., Coombes G. & Cookson B., 2003. Harmonization of Pulsed-Field
Gel Electrophoresis Protocols for Epidemiological Typing of Strains of
Methicilin-resistant
49

50

Références bibliographiques
Staphylococcus aureus: a single approach developed by
consensus in 10 european laboratories and its application for tracing the
spread of related strains. Journal of Clinical Microbiology; 41:
1574-1585.
Ngazoa-Kakou E. S., kouya D., Koudou A., Abole A.,
Guessend K. N., Sylla A., Sina K. M., Coulibaly N. D., Kouassi K. S., Meite S.,
Aoussi S. & Dosso M., 2017. New identification of phages in water
and in fishes from lagoon Ebrié and their application for Multidrug
strains in Abidjan, West Africa. World Journal of Pharmaceutical Research,
Vol 6, Issue 9: 84-89.
Ngazoa-Kakou S., Philippe C., Tremblay D. M., Loignon
S., Koudou A., Abole A., Ngolo Coulibaly D., Kouassi K. S., Kouamé S.
M., Aoussi S., Dosso M. & Moineau S., 2018. Complet
séquence du génome d'Ebrios, un nouveau T7virus isolé de
la lagune Ebrié à Abidjan, Côte d'Ivoire. Génome
Announcement 6: e00280-18.
Paul J. H., Sullivan M. B., Segall A. M. & Rohwer
F., 2002. Marine phage genomics. Comparative
Biochemistry And Physiology - Part B: Biochemistry &
Molecular Biology; 133:463-76. Payne R. J. H. & Jansen V. A.
A., 2001. Understanding bacteriophage therapy as a
density-dependent
kinetic process. Journal of Theoretical Biology, 208,
(1), 37-48p.
Ponchon L., Mangenot S., Boulanger P. & Letellier
L., 2005. Encapsidation and transfer of phage DNA into host cells:
From in vivo to single particles studies. Biochimica et Biophysica Acta
;1724: 255-261.
Ravat F., Jault P. & Gabard J., 2015.
Bactériophages Et Phagothérapie : utilisation de virus naturels
pour traiter les infections bactériennes : Using Natural Viruses To
Treat Bacterial Infections. Annals of Burns and Fire Disasters, vol. XXVIII
(1): 20p.
Rosner A. J., Becker D. L., Wong A. H., Miller E.
& Conly J.M., 2004. "The costs and consequences of
methicillin-resistant Staphylococcus aureus infection treatments in Canada.
Canadian Journal of Infection Diseases and Medical Microbiology,
15(4): 213-220.
Roux S., Debroas D. & Enault F., 2013.
Application des approches métagénomiques à
l'étude de la diversité virale environnementale. Virologie
, 17 (4) : 229-42.
Roux S., Enault F. & Robin A., 2012.
Assessing the diversity and specificity of two freshwater viral
communities through metagenomics. PLoSONE ; 7 : e336-41.
Safa A., Bhuiyan N. A., Alam M., Sack D. A. & Nair
G. B., 2005. Genomic relatedness of the new Matlab variants of
Vibrio cholerae O1 to the classical and El Tor biotypes as determined
by pulsed-field gel electrophoresis. Journal of Clinical Microbiology;
43: 1401-1404. Sambrook J. & Russell D.W., 2001.
Purification of nucleic acids by extraction with phenol: chloroform. In:
Molecular Cloning. 3rd ed. New York: Cold Spring Harbor Laboratory
Press: 379p.
Références bibliographiques
Sambrook J., Fritsch E. F. & Maniatis T., 1989.
Molecular cloning: a laboratory manual, 2nd ed. Cold Spring Habor
laboratory Press, Cold Spring Harbor, N.Y., 1626 p.
Scallan E., M-Hoekstra R., J-Angulo F., V-Tauxe R.,
Widdowson M. A., L-Roy S., L-Jones J. & M-Griffin P., 2011.
Foodborne illness acquired in the United States-major pathogens. Emerging
Infectious Diseases, 17(1):7-17.
Singh A. & Arutyunov D., (2012).
"Bacteriophage based probes for pathogen detection. The Analyst
;137(15) : 3405-3421p.
Souriau C., Hua T., Lefranc M. & Weill M.,
1998. Présentation à la surface de phages filamenteux :
Les multiples applications du phage display. Médecine /
sciences; 14: 300-9.
Steward G. F., 2001. Fingerprinting viral
assemblages by pulsed field gel electrophoresis (PFGE). In
Methods in Microbiology, ed. J.H. Paul, San Diego:
Academic: Vol. 30, p. 666. Sulakvelidze A., 2011.
Bacteriophage: A new journal for the most ubiquitous organisms on
Earth.
Bacteriophage; 1 (1): 1-2p.
Suttle C.A., 2005. Viruses in the sea.
Nature ; 437: 356-361.
Szermer-Olearnik B. & Boratyñski J., 2015.
Removal of Endotoxins from Bacteriophage Preparations by Extraction
with Organic Solvents. PLoS ONE ; 10 (3): e0122672.
Twort F. W., 1915. An investigation on the
nature of ultra-microscopic viruses. Lancet 2: 1241-1243.
Twort F. W., 1925. The Discovery of the «bacteriophage,
The Lancet, 205, (5303) 845p.
Vadim V. M., 2004. Bacteriophage T4
structure, Assembly, and initiation infection studied in three dimensions.
Elsevier; 0065-3527.
Vallot J., 2015. Intérêt des
bactériophages dans les infections à Pseudomonas aeruginosa
résistant aux antibiotiques, chez les patients atteints de la
mucoviscidose. Thèse de Doctorat d'Etat en Pharmacie de
l'université de Nantes, UFR Sciences Pharmaceutiques et Biologiques,
France, 94p.
Wagenaar J. A., Van Bergen M. A. P., Mueller M. A.,
Wassenaar T. M. & Carlton R. M., 2005. "Phage therapy reduces
Campylobacter jejuni colonization in broilers". Veterinary
microbiology; 109 (3-4): 275-283.
Wang J. B., Lin N. T., Tseng Y. H. & Weng S. F.,
2016. Genomic Characterization of the Novel
Aeromonas hydrophila Phage Ahp1 Suggests the Derivation
of a New Subgroup from
phiKMV-Like Family. PLoS ONE; 11(9): e0162060.
doi:10.1371/ journal. pone.0162060. Weinbauer M. G., 2004.
Ecology of prokaryotic viruses. Federation of European
Microbiological
societies Reviews, 28, (2), 127-181.
Williamson K. E., Radosevich M. & Wommack K. E.,
2005. Abundance and diversity of viruses in six Delaware soils.
Applied Environmental Microbiology; 71: 3119-3125.
51

Références bibliographiques
Willner D., Thurber R. V. & Rohwer F., 2009.
Metagenomic signatures of 86 microbial and viral metagenomes.
Environmental Microbiology.; 11(7) :1752-66.
Wommack K. E., et al., 1999.
Population dynamics of chesapeake bay virioplankton: total-community
analysis by pulsed-field gel electrophoresis. Applied Environmental
Microbiology; 65 (1): 231-240.
Wong H. C., Chen S. Y., Chen M. Y., Oliver J. D., Hor
L. I. & Tsai W. C., 2004. Pulsed-Field Gel Electrophoresis
Analysis of Vibrio vulnificus Strains Isolated from Taiwan and the
United States. Applied & Environmental Microbiology;
70: 5153-5158.
Xydas S., Lange C.S., Phil D. & Shih D. Y., 1996:
Effect of methylation on the electrophoretic mobility of chromosomal
DNA in pulsed-field agarose gels. Applied and theoretical electrophoresis:
the official journal of the International Electrophoresis Society;
6:43-47.
Zhang H., Bao H., Billington C., Hudson JA. & Wang
R., 2012. Isolation and lytic activity of the Listeria bacteriophage
endolysin LysZ5 against Listeria monocytogenesin soya milk. Food
microbiology; 31(1): 133-136.
Zhang J., Diao B., Zhang N., Cui Z., Zhang L., Xu J.
& Kan B., 2007). Comparison of different electrophoretic
parameters of Pulsed-Field Gel Electrophoresis for Vibrio cholerae
subtyping. Journal of Microbiology Methods, 71:15-22.
Zhang Y. & Hu Z., 2013. Combined
treatment of Pseudomonas aeruginosa biofilms with bacteriophages and
chlorine. Biotechnology and Bioengineering; 110 (1):286-295.
Zhao L., Kanamaru S., Chaidirek C. & Arisaka F.,
2003. P15 and P3, the tail completion proteins of bacteriophage T4,
both form hexameric rings. Journal of Bacteriology. 185: p.
1700-1693.
Zhong Y., Chen F., Wilhelm S. W., Poorvin L. &
Hodson R. E., 2002. Phylogenetic diversity of marine cyanophage
isolates and natural virus communities as revealed by sequences of viral capsid
assembly protein gene g20. Applied Environmental Microbiology; 68
:1576-84.
Annexes
Annexes
Annexe 1 : Les Protéines Phage T4 dans la base
de données (Miller et al., 2003).
|
Protéines
|
Description
|
Banque de données
|
|
AsiA
|
Anti- regulatory protein
|
1JR5, 1KA3
|
|
f3-gt
|
f3 Glycosyltransférase
|
1QKJ
|
|
DenV
|
Pyrimidine-dimer excisionase
|
2END, 1VAS
|
|
gpe
|
Lysozyme
|
1LYD
|
|
I-TevI
|
Intron-homing endonuclease
|
1I3J
|
|
MotA
|
Transcription regulatory factor
|
1BJA, 1I1S
|
|
NrdC
|
Glutaredoxin, thioredoxin
|
1ABA, 1DE1
|
|
NrdD
|
Anaerobic NTP reductase, large chain
|
1H77
|
|
RegA
|
Translation regulatory protein
|
1REG
|
|
Rnh
|
RNase H
|
1TFR
|
|
TS
|
Thymidylate synthase
|
1TIS
|
|
Wac
|
Fibritin deletions E and M
|
1AAO, 1AVY
|
|
gp1
|
dNMP kinase
|
1DEK
|
|
gp5/27
|
Tail-associated lysozyme
|
1K28
|
|
gp9
|
Long-tail fiber connector
|
1QEX
|
|
gp11
|
Baseplate-short-fiber connector
|
1EL6
|
|
gp12
|
Short tail fiber
|
1H6W
|
|
gp31
|
Cochaperone
|
1G31
|
|
gp32
|
ssDNA-binding protein
|
1GPC
|
|
gp42
|
dCMP-hydroxymethylase
|
1B5D
|
|
gp43
|
T4 DNA polymerase fragment, RB69,
DNA polymerase
|
1NOY,
1WAF
|
|
gp45
|
Processivity clamp
|
1CZD
|
|
gp49
|
EndoVII
|
1E7D
|
|
gp59
|
Helicase assembly protein
|
1C1K
|
|
nrdD intron
|
Group IA intron RNA/ribozyme
|
1SUN
|
Annexes
Annexe 2 : Electrophorèse en champ
pulsé (PFGE) de bactériophage digéré par l'enzyme
de restriction XbaI.

Puits 1 : marqueur (PFGE low range DNA
approximately 0.13-194 kb), Puits 2 : p T4b Puits 3 :
p T4c, Puits 4 : p A5a, Puits 5 : p
A5a, Puits 6 : p Ebrios, Puits 7 : p Ebrios,
Puits 8 : marqueur (PFGE low range DNA Marker in agarose
plugs), Puits 9 : p M3, Puits 10 : p M3,
Puits 11 : p M7, Puits 12 : p M7, Puits
13 : p M11, Puits 14 : p M11, Puits
15 : marqueur (PFGE low range DNA Marker in agarose plugs).
Annexe 3: Electrophorèse en champ
pulsé (PFGE) de bactériophage digéré par l'enzyme
de restriction XhoI.
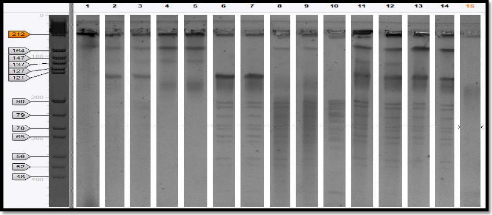
Puits 1 : marqueur (PFGE low range DNA Marker
in agarose plugs), Puits 2 : p T4c, Puits 3 :
p T4c, Puits 4 : p T4b, Puits 5 : p
T4b, Puits 6 : p A5a, Puits 7 : p A5a, Puits
8 : p M3, Puits 9 : p M3, Puits 10 :
marqueur de poids moléculaire GeneRuler 1 kb DNA Ladder, Puits
11 : p M7, Puits 12 : p M7, Puits 13
: p M11, Puits 14 : p M11, Puits 15 :
p Ebrios.
Annexes
Annexe 4 : Profil de digestion des phages par les
enzymes de restrictions XbaI et XmnI

M :Marqueur de poids moléculaire 1 kb;
Puits 1: p M7 ; Puits 2 : p
M3 ; Puits 3 : p M11 ; Puits 4 : p Ebrios ;
Puits 5 : p T4 ; Puits 6 : p A5a ; Puits
7 : p M7 ; Puits 8 : p M3 ; Puits 9
: p M11 ; Puits 10 : p Ebrios ; Puits 11
: p T4 ; Puits 12 : p A5a.
RÉSUMÉ
Les bactériophages ou phages sont les virus des
bactéries et ont la capacité de lyser celles-ci. Cette action
lytique fait des phages une alternative thérapeutique appelée
phagothérapie face à l'antibiothérapie. L'utilisation
abusive de l'antibiothérapie cause aujourd'hui une recrudescence
mondiale de la résistance bactérienne aux anti-infectieux
disponibles. En Côte d'Ivoire, les bactéries
multirésistantes sont rapportées dans les études cliniques
et environnementales. La phagothérapie méconnue du corps
médical professionnel et des populations. Des études
récentes ont rapporté la présence de phages à
potentiel thérapeutique mais aucune application n'est rapportée.
La mise au point de la phagothérapie nécessite une
caractérisation morphologique et génomique complète des
phages à potentiel lytique. L'objectif de déterminer le profil
génomique de bactériophages isolés en Côte d'Ivoire.
Spécifiquement d'analyser leur génome par la technique de
l'électrophorèse en champ pulsé et par la digestion
enzymatique. 5 phages dont 2 phages lagunaires de la Lagune Ebrié (p
A5a, Ebrios) et 3 phages (M3, p M7, p M11) isolés des
micromammifères ont été inclus dans cette étude.
Les enzymes XbaI, XmnI et XhoI ont été utilisés pour la
digestion des génomes. Les résultats obtenus montrent que les
phages sont des phages à ADN double brin. L'analyse en champ
pulsé montrent que les phages p A5a, p M3, p M7, p M11 ont une taille de
génome est supérieure à 164 Kpb. La diversité
génomique des phages est démontrée par les profils de
restriction de l'électrophorèse en champ pulsé et de la
digestion enzymatique. Cette étude montre que
l'électrophorèse en champ pulsé (PFGE) est une
méthode plus sensible que la digestion enzymatique pour la
caractérisation génomique. Par contre la digestion enzymatique
des phages est simple et rapide et pour déterminer le type de
matériel génétique des phages.
Mots clés : Bactériophages,
Diversité génétique, champ pulsé, Digestion
enzymatique, Cote d'Ivoire.
ABSTRACT
Bacteriophages or phages are the viruses of bacteria and have
the ability to lyse them. This lytic action makes phages a therapeutic
alternative called phagotherapy against antibiotic therapy. The misuse of
antibiotic therapy is now causing a worldwide resurgence of bacterial
resistance to available anti-infectives. In Côte d'Ivoire, multiresistant
bacteria are reported in clinical and environmental studies. The unknown
phagotherapy of the professional medical profession and populations. Recent
studies have reported the presence of phages with therapeutic potential, but no
application is reported. The development of phage therapy requires complete
morphological and genomic characterization of phages with lytic potential. The
objective of determining the genomic profile of isolated bacteriophages in
Ivory Coast. Specifically, to analyze their genome by the technique of pulsed
field electrophoresis and enzymatic digestion. 5 phages including 2 lagoon
phages of the Ebrié Lagoon (p A5a, Ebrios) and 3 phages (M3, p M7, p
M11) isolated from the micromammals were included in this study. XbaI, XhoI and
XmnI enzymes have been used for digestion of genomes. The results obtained show
that the phages are double-stranded DNA phages. Pulsed field analysis shows
that phages p A5a, p M3, p M7, p M11 have a genome size greater than 164 Kpb.
The genomic diversity of phages is demonstrated by the restriction profiles of
pulsed field electrophoresis and enzymatic digestion. This study shows that
pulsed field gel electrophoresis (PFGE) is a more sensitive method than
enzymatic digestion for genomic characterization. On the other hand, the
enzymatic digestion of the phages is simple and fast and to determine the type
of phage genetic material.
Keywords: Bacteriophage, Genetic
diversity, PFGE, Enzymatic digestion, Ivory Coast.
| 


