CHAPITRE 3. ÉTAT DE L' ART
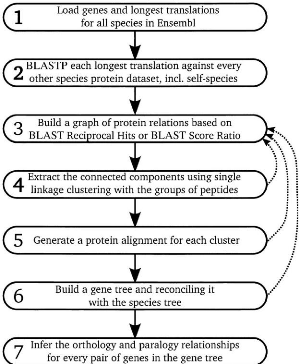
FIGURE 3.4 - Méthode de Ensembl-Compara pour la
construction des arbres de gènes
23
CHAPITRE 3. ÉTAT DE L' ART
de gènes a plus de 750 membres, les étapes 3 et
4 sont répétées en augmentant le seuil de conservation de
relation entre deux protéines.
5. Alignements multiples : Les
protéines d'une même famille de gènes sont alignées
à l'aide du programme "Multiple Sequence Comparison by Log-Expectation"
(MUSCLE) [8, 9] pour obtenir un alignement multiple.
6. Arbre de gène et réconciliation
: L'alignement multiple des protéines d'une famille de
gènes et l'arbre des espèces sont donnés en entrée
au programme de construction d'arbres "Tree Building guided by Species Tree"
(TreeBeST) 4, pour construire l'arbre des gènes de la
famille. L'arbre de gènes est alors réconcilié avec
l'arbre d'espèces afin d'étiqueter comme duplication ou
spéciation.
7. Inférence des orthologues et paralogues
: Les arbres de gènes obtenus sont finalement aplatis en des
tables d'orthologues et de paralogues décrivant les relations
d'homologie entre paires de gènes.
De manière générale, tous les arbres de
gènes disponibles dans les bases de données et dans la
littérature sont construits sur ce modèle.
3.2.2 Limites des arbres de gènes actuels
Dans cette section, nous illustrons les limites des arbres de
gènes construits par la méthode d'Ensembl Compara. Suivant cette
méthode, seule la protéine 3 du gène décrit dans la
figure 3.5 devrait être considérée pour la construction de
l'arbre des gènes de la famille à laquelle le gène
appartient. En effet, cette protéine est la plus longue. Elle
possède quatre exons sur les six que compte le gène. Cependant,
bien qu'étant la plus longue, elle ne couvre pas les exons E2 et E3 qui
ne seront jamais considérés par la suite, alors qu'ils sont
présents dans les protéines 1 et 2 qui sont produites par le
même gène.
| 


