I.2.4.3. Résistance des bactéries
entériques aux antibiotiques
On parle de résistance bactérienne lorsqu'un
antibiotique perd sa capacité à inhiber efficacement la
croissance bactérienne c'est-à-dire que les bactéries
continuent à se multiplier en présence de concentrations
thérapeutiques d'antibiotiques. Cette résistance aurait deux
origines essentielles, naturelles et acquises. La première est
programmée au niveau génomique alors que la seconde est
développée en fonction des conditions métaboliques (Julian
et Dorothy, 2010).
I.2.4.3.1. Résistance naturelle ou
intrinsèque
Une résistance intrinsèque se définit
comme une caractéristique fonctionnelle ou structurelle conférant
une certaine tolérance, (Muylaert et Mainil, 2012). La résistance
intrinsèque à un antibiotique est essentiellement due à la
présence de gènes chromosomiques ; elle est donc commune à
toutes les bactéries d'une même espèce. Elle peut
être due à des particularités structurales s'opposant
à l'action de l'antibiotique sur sa cible comme la présence d'une
membrane externe chez les bactéries à Gram négatif les
rendant naturellement résistantes aux antibiotiques de poids
moléculaire élevé comme les glycopeptides. La
résistance naturelle peut enfin être médiée par
l'expression constitutive ou induite d'une enzyme d'inactivation ou par la mise
en oeuvre d'un processus d'échappement vis-à-vis de
l'antibiotique (Diallo, 2013).
I.2.4.3.2. Résistance acquise
Ce terme est utilisé pour désigner le
résultat d'un processus permettant à des bactéries d'une
espèce originellement sensible de devenir résistante à un
ou plusieurs antibiotiques (Diallo, 2013). Une bactérie peut
acquérir une résistance aux antibiotiques par deux grands
mécanismes génétiques : l'un a pour support le
chromosome et définit une résistance chromosomique
(résultant d'une mutation), l'autre a pour support les plasmides
(liée à la synthèse de protéines additionnelles) ou
les éléments transposables ou les intégrons et
définit une résistance extra-chromosomique (Lozniewski et Rabaud,
2010).
I.2.4.3.3. Mécanismes de la résistance
bactérienne
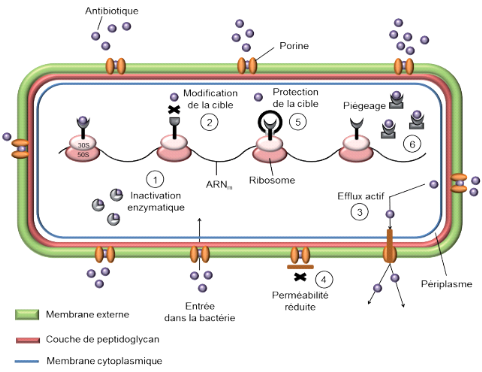
Les bactéries ont développé
différents mécanismes afin de neutraliser l'action des agents
antibactériens, la figure II nous renseigne sur ces mécanismes.
Il s'agit entre autre de:
· Inactivation enzymatique de
l'antibiotique : elle représente le principal
mécanisme de résistance aux bêta-lactames, aminoglycosides,
phénicolés, MLS (macrolides, lincosamides, streptogramines), les
tétracyclines, la fosfomycine et plus récemment pour les
fluoroquinolones. Les plus grands producteurs de â-lactamases sont les
staphylocoques, mais surtout les Gram négatifs ; les
anaérobies produisent surtout des céphalosporinases;la
chloramphénicol-acétylase confère aux Gram-positifs et
négatifs une résistance au chloramphénicol (Muylaert et
Mainil, 2012);
· Modification ou remplacement de la cible de
l'antibiotique : ce mécanisme est particulièrement
important pour des résistances aux pénicillines, aux
glycopeptides et aux molécules du groupe MLS chez les bactéries
Gram-positives, et pour les résistances aux quinolones chez les
bactéries Gram positives et Gram-négatives (Muylaert et Mainil,
2012);
· L'efflux actif : il est
médié par des protéines transmembranaires connues sous le
terme de pompes à efflux ou transporteurs actifs. C'est un
mécanisme nécessitant de l'énergie et utilisé par
les bactéries pour expulser à l'extérieur des
métabolites et des composés toxiques étrangers tels que
des antibiotiques et d'autres médicaments (Muylaert et Mainil, 2012).
Les pompes SDR (specific drug-resistance) sont utilisées par les
bactéries Gram-négatifs pour résister aux
tétracyclines principalement, tandis que les pompes MDR
(multiple-drug-resistance) sont retrouvées chez les bactéries
telles que P. aeruginosa, Escherichia coli, S. aureus (Muylaert et
Mainil, 2012);
· Perméabilité
réduite : il s'agit d'un mécanisme de
résistance cliniquement très important chez les bactéries
Gram-négatives plus précisément chez P.
aeruginosa et les Enterobacteriaceae, à causes de large spectre
d'antibiotiques qu'elles ciblent. Les structures en cause sont les porines,
canaux aqueux constitués de trois molécules de protéines
qui laissent diffuser diverses molécules de faible masse
moléculaire comme les antibiotiques. Le dysfonctionnement de l'une
d'entre elles peut entrainer une résistance à divers
antibiotiques comme les ß-lactamines, l'acide nalidixique, le
triméthoprime, la fosfomycine, la tétracycline ou encore le
chloramphénicol (Muylaert et Mainil, 2012 ; Diallo, 2013) ;
· Protection de la cible de
l'antibiotique : c'est un mode de résistance bien connu
pour la famille des tétracyclines et plus récemment décrit
pour les quinolones et les fluoroquinolones. Ainsi, on ne dénombre pas
moins de huit protéines de protection ribosomale qui confèrent
une résistance aux tétracyclines en les déplaçant
de leur site de fixation par la création d'un encombrement
stérique au niveau du ribosome (Muylaert et Mainil, 2012);
· Piégeage de l'antibiotique :
Les bactéries sont capables de piéger un antibiotique en
augmentant la production de sa cible ou en produisant une autre molécule
possédant une affinité pour ce dernier. Il en résulte une
diminution de la concentration de l'antibiotique à l'état libre
au niveau de la cible. C'est le cas de la résistance aux glycopeptides
chez certaines souches de S. aureus, et à la tobramycine chez
E. coli (Muylaert et Mainil, 2012).
| 


