III.4.1. Polymorphisme des marqueurs
utilisés
Les cinq marqueurs considérés se sont
montrés polymorphes avec des nombres d'allèles variables. La
figure 13 donne les fréquences alléliques par locus et pour
chaque espèce de glossines. Nous n'insisterons pas sur les
espèces pour lesquelles peu de glossines ont été
analysées.
Chez G. p. pallicera, 6, 10, 5, 10 et 9
allèles ont été respectivement identifiés aux loci
55.3, GPCAG, PGP13, C102 et PGP24. Au sein des populations de G. p.
pallicera, la comparaison des nombres d'allèles de
différents loci n'a montré aucune différence significative
(P= 0,47). Les loci GPCAG et C102 étaient les plus polymorphes avec
environ dix allèles distincts pour chacun des loci.
Chez G. p. palpalis, 10, 8, 16, 9 et 15
allèles ont été identifiés respectivement pour les
loci 55.3, GPCAG, PGP13, C102 et PGP24. Les loci PGP13 et PGP24 étaient
les plus polymorphes avec respectivement 16 et 15 allèles. Aucune
différence significative (P= 0,20) n'a été observée
en comparant les nombres d'allèles de différents loci au sein des
populations de G. p. palpalis.
Les valeurs de la richesse allélique entre G. p.
pallicera (4,960) et G. p. palpalis (10,785) montrent que la
variation du nombre d'allèle est non-significative entre ces deux
espèces (P= 0,33). Ces valeurs ont été calculées
parce qu'il était impossible de comparer le nombre d'allèle entre
ces deux espèces à cause de la disproportion entre le nombre
d'échantillons de G. p. pallicera et G. p.
palpalis.
39
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris

Figure 13: Nombre d'allèles en fonction des
espèces de glossines
Gnigr : G. nigrofusca ; Gpal
: G. p. pallicera ; Gcal : G. caligenea
; Gfus : G. fuscipes; Gpp :
G. p. palpalis ; Gmp : G. morsitans
La figure 14 donne le nombre total d'allèles par locus
pour l'ensemble des espèces de glossines. Le locus PGP24 a
été le plus polymorphe avec environ vingt-six allèles
distincts

Figure 14: Nombre d'allèles par locus
III.4.2. L'hétérozygotie et le
coefficient de consanguinité
L'analyse du coefficient de consanguinité (FIS)
révèle un déficit en hétérozygote non
significatif (FIS = 0,04 ; P = 0,31) pour G. p. pallicera.
Par contre, chez G. p. palpalis, le déficit en
hétérozygote est significatif (FIS = 0,145 ; P =
0,0001).
Les différentes valeurs de FIS reflètent
la différence entre les valeurs de Ho et Hs de G. p. pallicera
(tableau 5) et G. p. palpalis (tableau 6)
40
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Tableau 5: Hétérozygotie et les valeurs de
FIS en fonction des loci chez G. p. pallicera
|
Loci
|
55.3
|
GPCAG
|
PGP13
|
C102
|
PGP24
|
Moyennes
|
|
Ho
|
0,167
|
0,294
|
0,451
|
0,154
|
0,750
|
0,363
|
|
Hs
|
0,319
|
0,753
|
0,523
|
0,768
|
0,494
|
0,571
|
|
FIS
|
-0,199
|
0,519
|
-0,681
|
0,778
|
-0,535
|
0,040
|
Ho = Hétérozygotie
observée Hs = Hétérozygotie attendue
Tableau 6: Hétérozygotie et les valeurs de
FIS en fonction des loci chez G. p. palpalis
|
Loci
|
55.3
|
GPCAG
|
PGP13
|
C102
|
PGP24
|
Moyenne
|
|
Ho
|
0,917
|
0,467
|
1,000
|
0,500
|
0,923
|
0,761
|
|
Hs
|
0,886
|
0,857
|
0,955
|
0,879
|
0,952
|
0,906
|
|
FIS
|
-0,034
|
0,456
|
-0,048
|
0,431
|
0,030
|
0,163
|
Ho = Hétérozygotie
observée Hs = Hétérozygotie attendue
Les valeurs des FIS sont variables entre loci et sont
toutes différentes de zéro. Les figures 15 et 16
présentent les valeurs des FIS par locus, respectivement pour
G. p. pallicera et G. p. palpalis. La valeur moyenne de
FIS est proche de zéro pour G. p. pallicera et
éloignée de zéro pour G. p. palpalis.
41
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
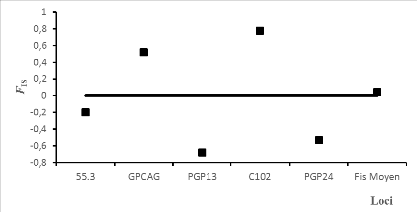
Figure 15: Valeurs de la FIS en fonction des
loci et pour les populations de G. p. pallicera
Figure 16: Les valeurs de la FIS en fonction
des loci et pour les populations de G. p. palpalis
Effet des allèles nuls sur les déficits
en hétérozygote ou les valeurs du FIS pour G. p.
palpalis.
Compte tenu du fait que la présence des allèles
nuls peut avoir un effet sur le déficit en hétérozygote,
nous avons analysé la relation entre les allèles nuls et les
FIS. La figure 17 illustre l'association entre le nombre
d'allèles nuls et les FIS pour G. p. palpalis. Cette
courbe de régression des allèles nuls (figure 17) donne la
relation entre les valeurs de FIS et les allèles
42
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
nuls. Le coefficient de corrélation (R2 =
0,00627) montre qu'il existe une corrélation négative entre les
allèles nuls et les valeurs de FIS.

Figure 17 : Courbe de régression des FIS
en fonction des allèles nuls pour G. p. palpalis
Effet des allèles nuls sur les déficits en
hétérozygote ou les valeurs du FIS pour G. p.
pallicera.
La figure 18 montre la courbe de régression des
FIS en fonction des allèles nuls. Le coefficient de
corrélation (R2 = 0,4681) montre qu'il existe une
corrélation négative entre les allèles nuls et les valeurs
de FIS.
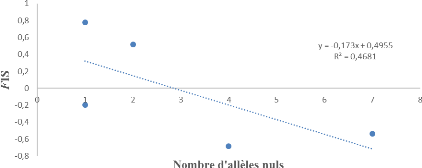
Figure 18 : Courbe de régression des FIS
en fonction des allèles nuls pour G. p. pallicera
43
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
La fréquence des allèles nuls de la sous
population G. p. palpalis a été estimée en
utilisant la deuxième méthode de Broockfield. En comparant les
allèles attendus, il ressort que les loci GPCAG et C102 ont
présenté des allèles nuls qui auraient contribué
à augmenter leurs valeurs de FIS. Tous les autres loci n'ont
pas présenté d'allèles significativement nuls (tableau
7).
Tableau 7 : Fréquence des allèles nuls
observés et attendus
|
Locus
|
Stuttering
|
Méthode de
|
PB22
|
N
|
N'
|
Blancs
|
Blancs
|
|
|
Brookfield 2
|
|
|
|
observés attendus
|
|
553
|
non
|
0
|
0
|
7
|
7
|
0
|
1,142
|
|
GPCAG
|
non
|
0,1047
|
0,0109
|
7
|
10
|
3
|
1,714
|
|
PGP13
|
non
|
0
|
0
|
7
|
7
|
0
|
1,142
|
|
C102
|
non
|
0,1538
|
0,0236
|
7
|
10
|
3
|
0,999
|
|
PGP24
|
non
|
0
|
0
|
7
|
7
|
0
|
1,428
|
PB22 : Fréquence
génotypique attendue des individus homozygotes nuls ;
Méthode de Brookfield 2 : Nous donne la
fréquence attendue des allèles nuls ; N : Nombre
d'individus génotypés ; N' : Correspond
à la somme de N et les blancs (homozygotes nuls) observés.
Déséquilibre de liaison
Pour voir s'il existait des associations entre les loci
microsatellites analysés, nous avons évalué le
déséquilibre de liaison. En effet, Le déséquilibre
de liaison est évalué sous l'hypothèse de l'absence
d'association statistique entre les loci. Globalement, les valeurs (tableau 8)
du test de déséquilibre de liaison montrent globalement une
absence d'association statistiquement significative aussi bien pour les
populations de G. p. palpalis que celles de G. p. pallicera
(tableau 8). Néanmoins, une association significative a
été observée au sein des populations de G. p.
pallicera pour les combinaisons de loci 55.3 et PGP13 (P = 0,002) au seuil
de significativité P= 0,0025 (en fait, en combinant les P-values de
plusieurs tests pour obtenir une P-value générale pour chaque
paire de locus, le logiciel recalcule le nouveau seuil qui est en
général 0,05/nombre de tests combinés). Ces loci sont donc
en déséquilibre de liaison.
44
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Tableau 8: P-values du déséquilibre de
liaison entre paires de loci
|
(Seuil de significativité : P=
0,0025)
|
|
|
|
|
Permutation
|
Gpal
|
Gpp
|
Tous
|
|
55.3 X GPCAG
|
0,125
|
1,000
|
0,152
|
|
55.3 X PGP13
|
0,002*
|
1,000
|
0,002
|
|
55.3 X C102
|
0,407
|
0,467
|
0,357
|
|
55.3 X PGP24
|
0,550
|
1,000
|
0,572
|
|
GPCAG X PGP13
|
0,035
|
1,000
|
0,035
|
|
GPCAG X C102
|
0,005
|
1,000
|
0,005
|
|
GPCAG X PGP24
|
0,225
|
1,000
|
0,255
|
|
PGP13 X C102
|
0,882
|
1,000
|
0,887
|
|
PGP13 X PGP24
|
0,115
|
1,000
|
0,115
|
|
C102 X PGP24
|
0,217
|
1,000
|
0,252
|
Gpal : G. p. pallicera; Gpp
: G. p. palpalis ; * : association statistiquement
significative
|
|



