
RtpuBLiQuE ALGtRiENNE DtMocRATiQuE ET
popuLAiRE
Ministère de l'enseignement supérieur et de
la recherche scientifique
Université de Tébessa
Faculté
de sciences : Institut de la science de la nature/vie
Département de
Biologie
Mémoire de fin d'étude pour
l'obtention du diplôme des études
supérieures
Option : Biochimie
Cas de transposon mariner
« Une approche expérimentale »
Préparé par : FERHI Rahma
Encadré par : Dr. TOUMI Nassima
Promotion : 2008/2009
Dédicace
Je dédis ce travail à mon très cher
père qui nous a quitté très tôt. Que dieu lui garde
dans ses vastes paradis. Je le dédis spécifiquement à ma
mère qui m'a beaucoup soutenu et à mes frères, mes soeurs,
mes beaux frères, mes belles soeurs, mes cousins et cousines surtout
Hasna, je vous apprécie beaucoup.
Je dédis ce travail à tout mes amis, ceux de
BirMokadem surtout Rabab et Rawia, et ceux de Tébessa surtout Fatima,
Hassina, Zina, Dalel et Meriem, Je vous respecte beaucoup, et je vous souhaite
le meilleur.
Remerciements
Je remercie l'université de Tébessa surtout le
département de Biologie qui nous a ouvert les portes pour l'obtention du
diplôme des études supérieures en Biochimie.
Un remerciement très chaleureux est dirigé vers
Docteur Halaimia-Toumi Nassima pour son encadrement, sa grande patience et sa
gentillesse, merci également pour le temps que vous avez consacré
pour la correction de ce manuscrit.
Je remercie les gens qui s'occupent de notre bibliothèque
pour leur sympathie.
Liste des figures :
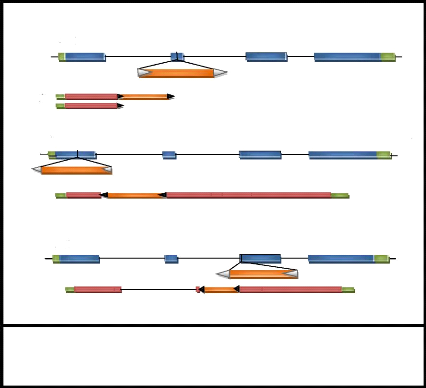
Figure 1 : Structure générale d'un
rétrotransposon à LTR.
Figure 1.1 : L'organisation génomique des
différentes familles des rétrotransposons à LTR.
Figure 1.2 : Cycle de vie d'un
rétrotransposon.
Figure 2 : Organisation structurelle des
rétroposons. Figure 2.1 : Modèle de
rétrotransposition de l'élément L1. Figure 3
: La structure d'une séquence d'insertion (IS1).
Figure 3.1 : La duplication du site cible lors
de l'insertion d'un élément transposable par la coupure
cohésive effectuée par la transposase.
Figure 3.2: La structure du transposon Tn10, les
IS sont orientées en sens inverse et forme des ITR.
Figure 3.3: la transposition
réplicative.
Figure 3.4: Le mécanisme
général de la transposition conservative. Figure
4: structure de l'élément Ac.
Figure 4.1 : La structure et l'épissage
somatique et germinale de l'élément P. Figure
4.2: Structure de l'élément Helitron du
maïs.
Figure 4.3 : Les différentes familles de
la superfamille
IS630/Tc1/ITmD41D/maT/mariner/Pogo/Ant1.
Figure 4.4 : Structure des
éléments de la famille Polintons/Mavericks.
Figure 5: Effet de l'insertion du
rétrotransposon à LTR, Tnt1, sur les transcrits du
gène nia2 codant la nitrate réductase chez Nicotiana
tabacum.
Figure 5.1 : Origine du système
immunitaire des mammifères par coaptation de l`activité
transposase d`un élément transposable.
Figure 6 : Résumé des
expériences conduisant à la découverte du transposon
mariner. Figure 6.1: La structure
générale des TLEs et des MLEs.
Figure 6.2: La structure d'un transposon
mariner, le cas du Mos1.
Figure 6.3 : Structure générale
de la transposase des MLEs (ici la transposase de Mos1).
Figure 6.4 : Structure de la transposase des TLEs
(ici la transposase de Sleeping Beauty). Figure 6.5:
Schéma du mécanisme de transposition des MLEs.
Figure 6.6: Représentation
schématique des différents complexes transposase/ITR.
Figure 6.7 : Les étapes de l'excision d'un
élément de type mariner.
Figure 6.8 : Les différentes
étapes de la transgénèse à l'aide de
mariner chez la souris. Figure 7 : Les
différentes étapes de la technique PCR.
Figure 8 : Schéma démontrant le
principe de Southern Blot.
Figure 9: Le séquençage selon la
méthode de Sanger.
Figure 10 : Structure nucléique de
l'élément Alvcmar.
Figure 11 : Localisation des
différents motifs et zones conservées sur la séquence
consensus des éléments Alvcmar. Lettres en gras : ITR,
lettres en bleu : UTR, lettres surlignes en gris : hélices á et
HTH.
Figure 12 : Expression de la transposase
fusionnée à la MBP par le plasmide pMAL.
Figure 13 : Le clonage du gène de la
transposase mariner ligué avec le plasmide pMAL dans une
bactérie.
Figure 14: Analyse de la production des
protéines sur gel SDS -PAGE.
Figure 15 : Principe de la technique de retard
sur gel.
Figure 16 : Analyse de la liaison de la
transposase Mos1.
Figure 17 : Schéma explicatif du principe
de l'analyse de spécificité.
Figure 18 : Analyse de la
spécificité de la fixation de la transposase Bytmar avec
leurs ITRs (Conditions expérimentales 30°C-2h).
Figure 19 : Schéma explicatif du test de
coupure.
Figure 20 : Analyse en test de coupure de la
transposase Alvcmar avec la construction 5T5.
Figure 21 : Test de transposition in
vivo.
Figure 22 : Les différents étapes
permettant la construction de vecteur porteur de gène da la
transposase.
Figure 23 : Les différentes étapes
de construction du vecteur pBC KC (+) porteur du pseudotransposon.
Figure 24 : Les différentes constructions
de pBC porteur de pseudo-transposon réalisées.
Figure 25 : Une représentation
schématique de la transposition inter-plasmidique obtenue avec la
transposase Mos1 au cours des tests de transposition in
vivo.
Liste des tableaux
Tableau 1: Transposons caractéristiques
d'E.coli.
Tableau 2 : Les différences
nucléiques entre les MLEs et les TLEs. Tableau
3: les différences protéiques entre les MLEs et
les TLEs. Tableau 4 : Quelques éléments
mariner potentiellement actifs.
Sommaire
Dédicace 2
Remerciements 3
Liste des figures 4
Liste des tableaux 7
Résumé 13
Introduction et historique 14
La Partie I : Les concepts de base des
éléments transposables 16
I- Classification générale des
éléments transposables: 17
I.1. Les éléments de la Classe I : Les
rétrotransposons 17
I.1.1. Les rétrotransposons à LTR : 17
I.1.1.a. Caractères généraux : 17
I.1.1.b. La classification : 19
I.1.1.b1. Les éléments autonomes : 19
I.1.1.b1.1. La super famille ty1/copia :
Pseudoviridae 19
I.1.1.b1.2. La super famille ty3/gypsy :
Métaviridae 20

I.1.1.b2. Les éléments non autonomes : 20
I.1.1.b2.1. La famille des TRIM : 20
I.1.1.b2.2. La famille des LARDs 21
I.1.1.c. Cycle de vie d'un rétrotransposon : 22
I.1.2. Les rétrotransposons sans LTR : ou
rétroposons 23
I.1.2.1. Les LINEs : 23
I.1.2.2. Les SINEs 25
I.1.2.3. Les RTEs : 26
I.2. Les éléments de la Classe II: Les
Transposons 26
I.2.1. Structure et diversité des transposons
procaryotes: 26
I.2.1.1. les séquences d'insertions: (IS)
26
I.2.1.2. les transposons composites: "Tn" 28
I.2.1.3. Les mécanismes de transposition: 30
1' La transposition réplicative : 30
1' La transposition conservative: 30
1' La transposase et son mode d'action: 32
I.2.2. Les transposons eucaryotes: 33
I.2.2.1. Le groupe des éléments hat:
34
I.2.2.2. Le groupe des éléments
PiggyBac: 35
I.2.2.3. Le groupe des éléments P:
36
I.2.2.4. Le groupe des MITEs/PIF/Harbinger: 37
I.2.2.5. Le groupe En/Spm: 38
I.2.2.6. L'élément Helitron : 39
I.2.2.7. Les éléments Mutator (Mu) 39
I.2.2.8. Le groupe des éléments de la super
famille IS630/Tc1/maT/mariner : 40
I.2.2.9. La famille Polintons(Mavericks) : 42
I.2.2.10. La famille des éléments Merlins
: 42
I.2.2.11. La Super famille des Transib : 43
II. L'impact des éléments transposables
sur le génome : 43
II.1. La modification de la structure et la fonction des
gènes : 44
II.2. Les réarrangements chromosomiques : 45
II.3. Les éléments transposables et la
régulation de l'expression du gène : 46
II.4. Néogènes (nouveaux gènes) et
recrutement d'élément transposable : 46
II.4.1. Recrutement de fonctions structurelles : 47
II.4.1. Recrutement de fonctions enzymatiques : 47
III. La régulation de la transposition :
49
III.1. L'autorégulation de la transposition :
50
II.1.1. Titration de la transposase par les séquences
délétées: 50
III.1.2. Les régions régulatrices : 50
III.1.3. Synthèse de répresseurs : 50
III.1.4. Inhibition par surproduction: 51
III.1.5. La Co-suppression: 51
III.1.5.1. La Co-suppression transcriptionnelle: 51
III.1.5.2. La Co-suppression post-transcriptionnelle:
52
III.2. Régulation de la transposition par les facteurs
de l'hôte : 52
III.2.1. L'effet de position : 52
III.2.2. La régulation
épigénétique : 52
IV. La transmission des éléments transposables
: 53
IV.1. Le transfert vertical : 53
IV.2. Le transfert horizontal : 53
Partie II : Caractérisations et
fonctionnalité des transposons de type
mariner « une approche
expérimentale » 55
I. Définition et Découverte: 56
I.1.La nomenclature des MLEs: 57
II. Structure et fonctionnalité des
éléments Tc1/mariner : 57
II.1. Structure générale des MLEs et
TLEs: 57
II.1.1. Les mariner- like elements ou MLEs:
58
II.1.1.1. Structure nucléique: 58
II.1.1.2. Structure protéique: 59
II.1.1.2.1. Les motifs caractéristiques du domaine
N-Terminal: 59
II.1.1.2.2. Les motifs caractéristiques du domaine
C-Terminal: 60
II.1.2. Une comparaison entre les MLEs et les TLEs:
61
III. Fonctionnalité des MLEs : 62
III.1. Les MLEs actifs : 62
III.1.1. Le mécanisme de transposition des
MLEs : 62
III.1.1.1. Liaison aux ITRs : 63
III.1.1.2. L'excision de l'élément : 66
III.1.1.3. La réinsertion de l'élément
dans son site cible : 67
III.1.2. L'intérêt de l'étude des
MLEs : 68
III.1.2.1. L'intérêt fondamental : 68
III.1.2.2. L'intérêt appliqué : 68
III.2. Les MLEs potentiellement actifs : 69
III.3. La caractérisation des transposons de
type mariner : 70
III.3.1. Recherche des éléments mariner
à partir de l'ADN génomique par PCR : 70
III.3.1.1. Extraction de l'ADN génomique: 70
III.3.1.1. PCR: 71
III.3.2. Hybridation des éléments par la
technique de Southern Blot : 72
III.3.3. Séquençage par la méthode de
Sanger : 73
III.3.4. Analyse des séquences in silico :
76
III.2.1.4. Analyse fonctionnelle d'un transposon de
type mariner : 78
III.2.1.4.1. Tests de transposition in vitro :
78
III.2.1.4.1.1. Analyse de la liaison des transposase de type
mariner aux ITRs : 81
III.2.1.4.1.2. Test de spécificité de liaison
des transposases de type transposase mariner aux ITRs : 83
III.2.1.4.1.3. Test de coupure des transposases de type
mariner : 84
III.2.1.4.2. Les tests de transposition in vivo :
86
III.2.1.4.2.1. Construction des vecteurs permettant
l'expression de la transposase mariner : 87
III.2.1.4.2.2. La construction de plasmide pBC KS(+) contenant
le pseudo transposon : 88
III.2.1.4.2.3. Réalisation des tests de transposition
in vivo : 90
Conclusion et perspectives 92
La Bibliographie 93
Résumé
« L'ADN » est la structure de base de toute
être vivant. Il était considéré comme une structure
figée, hors la découverte des gènes sauteurs par Barbara
McClintock (1953) a révolutionné le trajet de la
génétique et la biologie moléculaire. Les gènes
sauteurs ou les éléments transposables sont des séquences
répétées dispersées dans les chromosomes. Ils
codent pour des protéines spécifiques, ces dernières
assurent son déplacement par un phénomène appelé
transposition. Les éléments transposables sont subdivisés
en deux classes ; les éléments de la classe I ou les
rétrotransposons qui utilisent un intermédiaire ARN et les
éléments de la classe II qui se transposent directement par un
intermédiaire ADN en utilisant une enzyme spécifique
appelée transposase. Ils affectent leurs génomes hôtes
selon plusieurs façons et ils subissent des systèmes de
régulation très particuliers. Le transposon mariner est
un transposon de classe II qui appartient à la super famille
Tc1/mariner. Il est le transposon le plus simple connu chez les
eucaryotes. Cette simplicité à fait l'objet de plusieurs travaux
pour définir toutes ses propriétés et les
particularités de son mécanisme de transposition. La
caractérisation d'un élément mariner à
partir d'un organisme fait appel aux techniques connues de biologie
moléculaire et l'activité de la transposase peut être
testé in vivo ainsi qu'in vitro.
En effet, les résultats de ces études ont
montré d'une part que mariner est bel et bien un bon candidat
en vectorologie. Et en d'autre part, elles ont ouvert des perspectives
promotrices pour son utilisation large dans le monde de transfert de
gènes.
Introduction et Historique
Les génomes ont été
considérés comme des structures fixes qui obéissent aux
règles simples de transmission des caractères proposées
par Mendel (in Rivière, 2000), cependant, dès les années
1950s, Barbara McClintock qui étudiait le maïs mettait en
évidence la présence de séquences mobiles, responsables de
la création de nouvelles mutations (Renault et al, 1997).
McClintock a analysé les cassures des chromosomes du
maïs (il possède 10 chromosomes numérotés de 1
à 10 du plus long au plus court respectivement) et a attiré
l'attention sur le clivage très fréquent du chromosome 9 dans un
locus très particulier. Elle a constaté que ce clivage
était dû au comportement de 2 facteurs: un facteur appelé
Ds (pour dissociation) qui est situé dans le site de clivage,
et un autre facteur qui est indispensable pour l'induction de la cassure du
chromosome 9 où se localise le Ds, ce deuxième facteur
est appelé Ac (In Griffths et al., 2004). Ces deux
facteurs ont été nommés les éléments de
contrôle suite à leur pouvoir d'activer des gènes dormants
à leurs cotés après excision de ces
éléments. Pendant plusieurs années, le maïs a
été le seul système génétique dans lequel il
a été observé des éléments mobiles. Or
à la fin des années 1960, certaines mutations pléiotropes
(affectant plusieurs fonctions) chez E.coli ont résulté
de la présence de grands fragments d'ADN (appelés IS
pour insertion sequence ou séquence d'insertion) qui s'étaient
intégrés dans le génome (In Watson et al.,
1994)
Des noms très variés désignaient ces
éléments comme: les éléments de contrôle, les
gènes sauteurs, les gènes mobiles, mais les noms les plus
courants actuellement sont: les transposons et les éléments
transposables (In Griffiths et al., 2004).
Les éléments transposables sont des
séquences d'ADN moyennement répétées, capables de
se déplacer le long des chromosomes. Ils codent pour les
protéines nécessaires à leur déplacement (le
phénomène est appelé transposition), ils sont
présents chez tous les organismes dès la bactérie
jusqu'à l'homme. Ils se répartissent en deux principales classes
sur la base de leur mécanisme de transposition:
1. les éléments de la classe 1 ou
rétroéléments qui transposent via la transcription inverse
(par une transcriptase inverse) d'un intermédiaire ARN
(Anxolabéhère et al, 2000).
2. Les éléments de la classe 2 dont une
molécule d'ADN est l'intermédiaire de la transposition, ces
éléments sont caractérisés par la synthèse
d'une transposase permettant toutes les étapes de la transposition
(Renault et al, 1997).
Actuellement, de nombreux chercheurs ont étudiés
les éléments transposables de point de vue structural et
fonctionnel, ainsi que leurs effets sur les génomes hôtes car ils
se révèlent de plus en plus comme des composés
fondamentaux des génomes (In Amara 2008). Plusieurs
éléments transposables ont été
caractérisés et démontrée actifs et d'autres
restent potentiellement actifs. Des procédures bien adaptées sont
actuellement utilisées pour caractériser un élément
et démontré son activité. Ceci est rendu possible à
l'aide de plusieurs expériences comme les tests de transposition in
vivo et les tests de transposition in vitro.
Dans ce travail, une première partie va mettre la
lumière sur les éléments transposables, en citant ses
différentes classes, leurs impacts sur les hôtes ainsi que les
niveaux de régulation de leur transmission. Une seconde partie
détaillera les transposons Mariner et donnera une approche
expérimentale qui permet la caractérisation d'un transposon et le
test d'activité.


I- Classification générale des
éléments transposables:
Les éléments transposables sont divisés
selon leur mécanisme de transposition en deux grandes classes :
I.1. Les éléments de la Classe I : Les
rétrotransposons
Les rétrotransposons ou les éléments de
la classe 1 ont la particularité de transposer par une copie
intermédiaire d'ARN. Ils utilisent pour ceci une reverse transcriptase,
cet ARN possède une double fonction :
· Il est une matrice pour la synthèse des
protéines nécessaires au cycle de rétrotransposition.
· Il sert aussi de matrice pour la synthèse d'un
brin d'ADN complémentaire par transcription inverse. (In Rivière
; 2000).
La classification de ces éléments est
fondée sur un critère structural qui est la présence ou
non de longues répétitions terminales « les LTR (Long
Terminal Repeat) ». De ce fait, les rétreotransposons sont
rangés en deux classes
1. Les rétrotransposons à LTR.
2. Les rétrotransposons sans LTR. (In Klug et
al., 2006). I.1.1. Les rétrotransposons à LTR
:
I.1.1.a. Caractères généraux :
Un élément à LTR est
généralement une portion d'ADN centrale longue de plusieurs Kpb
« constituée d'un seul ou de plusieurs cadre de lecture ouvert ORF
(pour Open Reading Frame) » et flanquée par des LTR longs de
plusieurs centaines à un millier de pb. Ces LTR portent les promoteurs
et les signaux de régulation de la transcription de
l'élément, et ils sont parfois bordés par de courtes
répétitions inversées de quelques pb. (In Singer et Berg,
1992). L'insertion de la majorité de ces éléments produit
une duplication au niveau de leur site cible, dont quelques uns
présentent une spécificité pour sa séquence. (Malik
et Eickbush ; 1999). Les rétrovirus et les rétrotransposons
sont
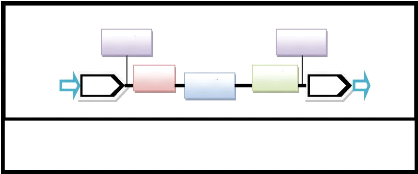
Figure 1 : structure générale d'un
rétrotransposon à LTR
LTR
PBS
(D'après Mhiri et Grandbastien, 2004)
gag
pol
env?
PPT
LTR
structurellement, fonctionnellement et évolutivement
très proches. Ils se diffèrent seulement dans la capacité
des rétrovirus à infecter les cellules adjacentes, parce qu'ils
sont porteurs d'une fonction additionnelle env permettant une
étape extracellulaire (où le virus infecte d'autres cellules).
D'une manière générale les rétrotransposons
à LTR sont caractérisés par :
> A chaque extrémité, des LTR identiques,
dont leurs transcrits portent le motif 5'-R-U5-PBS et PPT-U3-3', sont
retrouvés en orientation directe, PBS (Primer Binding Site) et PPT
(PolyPurine Tract) sont impliqués dans la réplication de ces
éléments.
> Ils portent au moins de 1 à 2 ORF, appelé
gag, pol, le gène gag code pour les
protéines de la capside, et pol code une polyprotéine
donnant naissance la reverse transcriptase (RT) et la ribonucléase H
(RH) qui sont requises pour la réplication du rétrotransposon,
l'intégrase (INT) qui permet à la molécule de l'ADN fille
de s'intégrer dans un nouvel site, et la protéase (PR) qui clive
la polyprotéine aux protéines correspondantes. Etant donné
que la plupart des rétrotransposons sont dépourvus du gène
env qui code pour les protéines de l'enveloppe virale,
toutefois la frontière entre le monde des rétrovirus et celui des
rétrotransposons est parfois très floue, car quelques
éléments comme les éléments gypsy, 297 et
tom de la Drosophile sont porteurs d'ORF additionnels de type env
(figure1) (in Griffiths et al., 2004 ;Mhiri et Grandbastien, 2004
; Renault et al., 1997 ; Pelsy et Merdinoglu, 2002 ; In
Rivière, 2000).
I.1.1.b. La classification :
Comme tous les cadres taxonomiques, la classification au sein
de ces éléments est actuellement soumise à de
fréquentes révisions en raison de l'identification de nouvelles
familles (Havecker et al., 2004). En fonction de l'ordre des domaines
dans leurs séquences Pol, les rétrotransposons à
LTR sont essentiellement divisés en deux grandes super familles, dont la
mobilité est autonome, la première est ty1/copia
(Pseudoviridae) et la deuxième est ty3/gypsy
(Métaviridae) (in Luchetta et al., 2005). Cependant,
Witte et al. (2001) ont pu découvrir une nouvelle
catégorie appelée TRIM (Terminal Repeat
Retrotransposon In Miniature) (Mhiri et Grandbastien, 2004), aussi un
autre groupe de chercheur a identifié des éléments
regroupé sous le terme de LARDs (pour Large Retrotransposon
Derivatives). (Havecker et al., 2004) la mobilité des
éléments au sein de ces dernières familles est
supposée d'être en trans c'est-à-dire qu'elle
nécessite la présence des protéines d'un autre
élément autonome (ces éléments sont décrits
ultérieurement) (Schulman et Kalendar, 2005).
I.1.1.b1. Les éléments autonomes :
I.1.1.b1.1. La super famille ty1/copia : Pseudoviridae
La taille des éléments de cette super famille
est comprise entre 3925pb12088pb, les LTRs sont entre 156pb-1929pb, et
présentent un TSD (pour Target Site Duplication) de 5pb. L'ordre des
domaines des gènes Pol est : PR-INT-RT-RH (Figure1.1) dont les
protéines sont caractérisées par des motifs hautement
conservés (Pelsy et merdinoglu , 2002). Cette super famille est
rangée en trois familles : les Hémivirus, les Pseudovirus, et
enfin les Sirevirus (la nomenclature n'indique pas qu'ils sont vraiment des
virus). Les Sirevirus se distinguent par leur abondance dans les génomes
végétaux et aussi par la séquence protéique de leur
RT. Les Hémivirus et les Pseudovirus sont caractérisés par
les amorces utilisées pour la transcription inverse qui sont un ARNt
complet et un demi-ARNt respectivement (Havecker et al., 2004). Dans
cette Super Famille il existe entre autres les rétrotransposons
Ty1, Ty2 et Ty4 de la levure Saccharomyces
cerevisiae, les copia -like (copia, mdg1, mdg3)
et 1731 de la drosophile (In Halaimia Toumi, 2006).
I.1.1.b1.2. La super famille ty3/gypsy :
Métaviridae
L'ordre des domaines des gènes Pol est :
PR-RT-RH-INT, qui est semblable à celui des rétrovirus (Figure
1.1). Les membres de la super famille ty3/gypsy sont
d'intérêt, parce que, contrairement à leurs voisins les
rétrovirus, ils possèdent parfois une spécificité
remarquable pour leur site d'insertion, par exemple, l'élément
ty3 de la levure S. cerevisiae s'insère exactement aux
environs 5pb du site de début du gène codant pour l'ARN
polymérase Ø, cela s'avère un excellent choix car ce
gène possède des promoteurs internes, de sorte que l'insertion
ait un effet minime sur l'expression génétique (Malik et Eikbush,
1999). Certains éléments de cette super famille peuvent
acquérir un ORF additionnel codant le gène env des
rétrovirus, c'est le cas des éléments gypsy et
ZAM de la drosophile. Il a été démontré
que l'élément gypsy posséde une capacité
infectieuse. Les analyses phylogénétiques des séquences de
la Reverse Transcriptase montrent que la super famille ty3/gypsy est
généralement divisée en trois familles : les
Métavirus dont l'élément Boudicca de l'homme et
Athlia d'Arabidopsis, les Errantivirus dont
l'élément gypsy et les Sémotivirus qui sont
présents aussi chez les nématodes (Havecker et al.,
2004).
I.1.1.b2. Les éléments non autonomes :
I.1.1.b2.1. La famille des TRIMs :
Les TRIMs (pour Terminal Repeat Retrotransposon
In Miniature) ont été décrits pour la première
fois dans les génomes du pomme de terre (Havecker et al.,
2004), ils présentent la plupart des caractéristiques
structurales des rétrotransposons à LTR, mais sont beaucoup plus
petits (300 à 800 pb) composés de petites LTR encadrant un court
domaine central non codant (Figure1.1). L'origine des TRIM est encore
peu claire et il est probable que leur amplification nécessite une
trans-activation par un autre rétrotransposon (Mhiri et
Grandbastien, 2004), un des éléments connu de ce groupe est
appelé Cassandra, il est une séquence de 565-860 pb,
avec des LTR qui varient selon les espèces, leur taille est comprise
entre 240-350pb. Les LTR d'un élément Cassandra

Figure 1.1: L'organisation génomique
des différentes familles des rétrotransposons
à LTR.
(D'après Havecker et al., 2004).
LTR
LTR
LTR
PBS
PBS PPT
PBS PPT LARDs
gag
LTR
gag PR-INT-RT-RH
PBS
PR-RT-RH-INT
Le gène pol
Le gène pol
PPT
LTR
env
TRIM
Ty1/copia
LTR
LTR
PPT
LTR
Ty3/gypsy
totalement séquencé présentent un motif
très conservé : 5'-TG.....CA3'et des ITRs de 6- 12 pb (Kalendar
et al., 2008).
I.1.1.b2.2. La famille des LARDs :
Les LARDs identifiés dans l'orge et d'autre
membre de Triticaeae ont des LTRs de 4.5kb et un domaine interne
non-codant de 3.5kb. Le mécanisme de déplacement de ces
éléments est mal-connu, mais en se basant sur les similitudes des
séquences, il semble que ces éléments se mobilisent sous
l'action d'autres éléments tels que les métavirus
Erika-1 de Triticum monococcum et RIRE3 du riz
(Figure1.1) (Havecker et al., 2004).
I.1.1.c. Cycle de vie d'un rétrotransposon :
Les rétrotransposons à LTR suivent un cycle de
vie analogue à celui des rétrovirus décrit par Echalier en
1989 (In Rivière, 2000), la transcription de l'élément en
ARNm débute dans la région 5'-R à la région R-3'des
LTR, de sorte que l'ARN bicistronique (génère deux
protéines) soit une version tronquée de l'ADN en raison de
l'absence des régions U3 en 5 `et U5 en 3'. Par la suite, cet ARN est
traduit aux différentes protéines des gènes pol
et gag (plus des protéines env dans le cas des
Errantivirus), la polyprotéine pol est clivée par la
protéase « PR », en INT, RH, PR et RT, les protéines
gag se polymérisent pour former les particules viraux, ensuite
l'ARN est assemblé dans ces particules où commence la
transcription inverse par le couple RT-RH, la reverse transcriptase
synthétise l'ADN en utilisant l'ARN comme matrice avec un taux d'erreur
de (2.5×10-5 erreurs/nucléotide/cycle) et la
Ribonucléase H lyse l'ARN matricielle après l'achèvement
de la transcription inverse. Enfin, l'ADN double brin synthétisé
est couplé à l'intégrase INT (Sabot et Schulman, 2006)
(qui est caractérisée par un motif catalytique de type DDE
analogue à celui des enzymes des éléments
mariner) sur les LTRs, ce complexe migre dans le noyau dont INT assure
l'intégration du rétrotransposon dans un nouvel site hôte
(Figure1.2) (Kapitonov et Jurka, 2006 ; Sabot et Shl 2006)

PR
Figure 1.2: Cycle de vie d'un
rétrotransposon. (D'après Sabot et Schulman, 2006)
I.1.2. Les rétrotransposons sans LTR : ou
rétroposons
Ces éléments ont également une phase ARN
dans leur cycle de rétrotransposition, ils ne possèdent pas des
LTR et sont subdivisés en deux familles selon leur autonomité,
les éléments autonomes « LINEs (Long
Interspersed Nuclear Elements) », et les éléments non
autonomes « SINEs (Short Interspersed Nuclear Elements)
», actuellement une nouvelle famille appelée RTEs
(RetroTransposable Elements) est connue (Figure 2) (In Tempel,
2007).
I.1.2.1. Les LINEs :
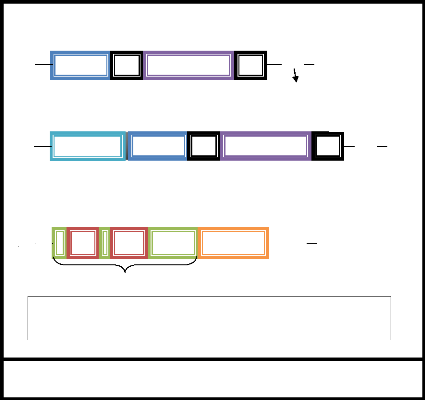
Figure 2 : Organisation structurelle des
rétroposons. (In Tempel, 2007 ; Huchon et
al., 2002)
5'
5'
5
Légende : ENDO : endonucléase, RT : Reverse
Transcriptase,
An : queue poly A ; A.B : sites de fixation à
l'ARN polymérase Ø
ENDO
ORF1
A
Domaine ARNt
B
ENDO
ORF2
RT
ORF2
O
RT
Queue poly A
An 3'
An
SINEs
RTEs
3'
LINEs
An
3'
Les éléments autonomes de type LINEs sont
représentés majoritairement dans les génomes des
mammifères y compris l'homme, ils constituent 40% du génome
humain, et il existe plusieurs éléments
LINEs : L1, L2, L3 le premier étant le seul
élément actif décrit dans le génome humain. Un
élément L1 complet est une séquence d'environ
6kpb, les éléments incomplets présentent des troncations
en 5', de réarrangement interne ou de mutations. Un TSD de 9-19pb soit
présent, ou non selon les espèces, à chaque
extrémité de l'élément. L1 possède
en 3' une séquence riche en A (poly A), il se trouve des
séquences allant de séries d'A presque ininterrompues
jusqu'à une répétition en tandem de la séquence
TAAA ou de séquences similaires (Gilbert, non publié ; in Singer
et Berg, 1992), il existe aussi deux ORF, l'ORF1 code une protéine de
liaison à l'ARN de 40kDa, qui se lie avec l'ARN du L1 dans le
cytoplasme pour former un complexe ribonucléoprotéine « RNPs
(RiboNucleoprotein Particles) >>, tandis que l'ORF2 code une
protéine multifonctionnelle présentant des activités
Endonucléase et Reverse Transcriptase (RT), l'endonucléase
génère des trous dans l'ADN chromosomique, et la RT transcrit
inversement l'ARNm en ADN en utilisant le résidu 3' hydroxyle
libéré comme amorce, il semble que la transposition d'un
élément L1 s'effectue par un processus appelé
« TPRT (Target-Primed Reverse Transcription) >> (In Zhan, 2007)
c'està-dire que l'endonucléase réalise une coupure simple
brin dans l'ADN de l'hôte, et la queue poly A de l'ARN de
rétroposon se fixe à une région poly T. D'une façon
plus générale la rétrotransposition du L1 peut se
résumer dans deux étapes. La première est la transcription
et la traduction d'un élément L1 actif pour former le
RNP. La deuxième est la transcription inverse et l'intégration
qui sont concomitantes à un nouveau site chromosomique (Figue2.1)
(Gilbert, non publié).
Figure 2.1 : Modèle de
rétrotransposition de l'élément L1 (d'après
Gilbert,
non publié)
Protéine de liaison
à l'ARN La protéine
multifonctionnelle
I.1.2.2. Les éléments SINEs :
Ils sont des rétroposons non autonomes
dérivés principalement des ARN de transfert ou de l'ARN
cytoplasmique 7SL (Figure 2), le SINE le plus abondant chez l'homme
est appelé Alu, parce qu'il contient un site de clivage pour
l'enzyme de restriction Alu1. Un élément Alu
complet est d'environ 200 nucléotides, et contient deux
répétitions d'environ 120pb encadrant une séquence de
60pb. Le mécanisme de déplacement des SINEs, n'est pas
encore élucidé : dans la mesure où ils ne codent pas pour
les fonctions nécessaires à leur rétrotransposition, les
SINEs pourraient utiliser la Reverse Transcriptase des
éléments LINEs. Ces « ADN égoïstes
» sont avérés des outils très efficaces en
systématique moléculaire : en effet, l'insertion d'un SINE
à un site donné est un événement unique et non
réversible à l'échelle des génomes (Huchon et
al., 2002 ; Renault et al., 1997 ; in Griffiths et
al., 2004).
I.1.2.3. Les RTEs :
Ils sont des rétroposons autonomes d'environ 3,3 kb et
possèdent de larges répétitions d'une centaine de paires
de bases. Contrairement aux autres rétroposons, l'élément
RTE ne code que pour l'ORF2 (Figure 2), ce qui en fait le plus petit
rétroposon autonome connu actuellement (In Tempel, 2007).
I.2. Les éléments de la Classe II: Les
Transposons
Ces éléments transposent d'un site chromosomique
à un autre grâce à l'activité d'une transposase, une
enzyme codée par l'élément sur un ou plusieurs ORF
(Anxolabéhère et al, 2000). Ils sont Essentiellement
caractérisés par la présence des ITR (Inverted Terminal
Repeat ou répétition terminal inversé) en orientation
inverse qui flanquent les transposons à chaque extrémité.
(Renault et al, 1997).
Au cours des 30 dernières années, le nombre des
transposons chez les procaryotes et les eucaryotes identifiés n'a
cessé à croître, avec une grande diversité dans leur
structure et une remarquable conservation des mécanismes qui assurent
leur mobilité (Merlin et Toussaint, 1999).
I.2.1. Structure et diversité des transposons
procaryotes:
La classification des transposons procaryotes se repose sur des
critères fonctionnels et structuraux. De ce fait, ils sont
regroupés en deux classes principales:
· Les séquences d'insertions (IS)
· Les transposons composites (Tn) I.2.1.1.
Les séquences d'insertions: (IS)
La première séquence d'insertion était
découverte chez E-coli pendant les années 60 à
l'occasion d'une mutation qui apparaissait dans les gènes constituant
l'opéron lactose grâce à l'insertion de cette
séquence.
Les séquences d'insertions sont les
éléments transposables les plus simples, de petite taille
(généralement inférieure à 2500pb) (Merlin et
Toussaint, 1999) et bordées à
chaque extrémité par des ITR (< 50pb) (In
Griffiths et al., 2004) indispensables pour le phénomène
de transposition et qui sont reconnus spécifiquement par la transposase
( figure 3) (In Singer, Berg, 1992).
Figure 3 : La structure d'une séquence
d'insertion (IS1) (d'après Singer, Berg.,
1992).
GGTGATGCTGCCAACTTACTGAT
CCACTACGACGGTTGAATGACTA
722pb
ORF
ORF
ATCAATAAGTTGGAGTCATTACC
TAGTTATTCAACCTCAGTAATGG
Selon les différences dans leurs séquences, les
IS ont été cataloguées en 17 familles dont les
membres des 12 entre elles contiennent un seul ORF qui code pour la
transposase, à partir de ce même cadre ouvert de lecture, les
IS de certaines familles codent pour une autre protéine, une
version tronquée de la transposase mais douée d'une
activité régulatrice pour la transposition. Pour les membres de
la famille IS3, la synthèse de la transposase (ORF AB)
résulte d'un déphasage programmé de la traduction entre
deux cadres de lecture ouvert qui codent pour une protéine
régulatrice de la transposition (ORF A) et une protéine de
fonction inconnue (ORF B) (Merlin et Toussaint., 1999). La séquence du
site d'insertion varie pour une même IS mais pas sa longueur,
aussi, certains éléments manifestent une
spécificité pour une séquence en palindrome (par exemple
elle est 5'-CTAG pour IS5), lors de l'intégration d'une IS une
duplication du site cible se produit, ce qui conduit à en trouver une
copie à chaque extrémité de l'élément
(Merlin et Toussaint., 1999; In Singer et Berg., 1992; Renault et al.,
1997) ( figure 3.1).
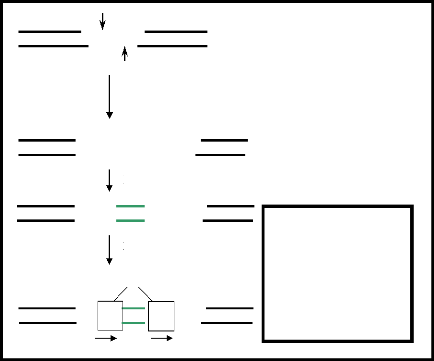
AGGTAAGGTA
AGG TCCATTCC
TAAGGTAG
ATC
TCCATTCCATC
La Transposase coupe le site cible
L'insertion de l'élément transposable
Figure 3.1 : La
duplication du site cible lors de l'insertion d'un
élément transposable par la coupure cohésive
effectuée par la transposase. (d'après Griffiths et al.,
2004)
AGG TCCATTCC
TAAGGTAG
ATC
AGGTAAGG
TCCATTCC
TAAGGTAG ATTCCATC
L'hôte répare les brèches
Un ITR de 5 pb flanque l'élément
I.2.1.2. les transposons composites: "Tn"
Ils sont des entités avec des structures complexes
(peuvent compter jusqu'à plusieurs milliers de pair de base), ils
contiennent une variété des gènes encadrés par deux
IS en direction directe ou inverse (figure 3.2) (In Watson et
al., 1994; In Singer et Berg, 1992), très souvent seule une des
deux séquences d'insertion code une transposase fonctionnelle, tandis
que l'autre code majoritairement pour un régulateur de la transposition,
le choix de transposition d'un Tn entier ou d'une seul IS se
fait en fonction de la taille de la séquence située entre les
deux IS c'est-à-dire que plus un Tn est court plus qu'il a
tendance à être mobilisé en entier (Merlin et Toussaint,
1999; Renault et al., 1997). Le segment d'ADN bordé par les
deux IS peut coder pour n'importe quelle fonction comme la
résistance aux antibiotiques (kanamycine chez Tn5,
tétracycline chez
Tn10), ou une fonction métabolique (catabolisme
du citrate chez Tn3411) (Tableau 1) (Merlin et Toussaint, 1999).

Figure 3.2: La structure du transposon Tn10,
les IS sont orientées en sens inverse et
forme des ITR
(d'après GRIFFITHS et al., 2004).
IS10 IS10
Résistance à la
tétracycline
Tranposon Tn10
Tableau 1: Transposons caractéristiques
d'E.coli (Tableau d'après Singer et Berg,
1992)
|
Transposon
|
Taille (pb)
|
Extrémités IS
|
Orientation des IS
|
Réplique terminale inverse
(pb)
|
Duplication du site
cible
|
Gènes
portés
|
|
Tn3
|
4957
|
Aucun
|
|
38
|
5
|
Ampr
|
|
Tn9
|
2638
|
IS1
|
Identique
|
23
|
9
|
Camr
|
|
Tn10
|
9300
|
IS10
|
Inverse
|
1329
|
9
|
tetr
|
Ces transposons peuvent passer du chromosome bactérien
au génome d'un phage ou dans un plasmide conjugatif. Pour ces raisons,
ces transposons peuvent être transmis à d'autres bactéries.
Ce type de transposon est une cause naturelle d'acquisition de
résistance aux antibiotiques pour les bactéries (in Halaimia
Toumi, 2006).
I.2.1.3. Les mécanismes de transposition: Deux
modèles de transposition des IS et donc des Tn ont été
observés
v' La transposition réplicative:
(appelé aussi la co-intégration ou
copier-coller)
Ce processus implique la formation d'une structure
moléculaire de type "crossing-over" appelée
co-intégrat. Cette structure résulte de la fusion entre un
réplicon (molécule circulaire capable de se répliquer)
donneur et un réplicon récepteur avec une copie du transposon
à chacune des deux jonctions entre ces deux molécules (In Singer
et Berg, 1992; Renault et al., 1997), généralement cette
structure ne persiste pas et une recombinaison entre les deux copies du
transposons résout le co-intégrat. La transposase produit une
coupure à un site bien précis de chaque côté du
transposon ainsi qu'au site d'insertion, ensuite ces extrémités
sont liées et une réplication se produit en utilisant les
fonctions réplicative de la cellule hôte pour former le
co-intégrat qui est détaché par une résolvase
codée par le transposon et qui agit au niveau du site res
également porté par le transposon. Dans certains cas, une
intégrase achève la résolution du co-intégrat en
agissant sur un site appelé att (figure 3.3) (Merlin et
Toussaint, 1999; In Singer et Berg, 1992; Renault et al., 1997).
v' La transposition conservative: "non
réplicative" "couper-coller" cut-paste":
Ce modèle correspond à la simple insertion, la
transposase se fixe aux ITRs pour former une structure compacte appelée
complexe synaptique ou transpososome, qui rapproche les ITRs, ensuite une
coupure double brin est effectuée par la transposase conduisant à
la formation de l'intermédiaire de transposition (une molécule de
transposase fortement associée au transposon). En présence d'ions
calcium ou magnésium, la transposase coupe au site cible d'une
façon décalée et une structure branchée de type
crossing-over est formée entre les brins d'ADN du transposons
donneur et la séquence cible. La réparation des
extrémités décalées conduit à la duplication
de la séquence cible. Cette transposition se distingue de la
précédente par le fait que l'intégration du transposon
dans le site cible se fait sans le passage par une étape de
réplication (Figure 3.4) (In Singer et Berg, 1992; Renault et
al., 1997; In Watson et al., 2004).

31
Figure 3.3: la transposition
réplicative
Transposon
Résolution par la résolvase ou
l'intégrase
La transposase coupe le réplicon donneur
Fusion entre les deux réplicons et
réplication
du transposon
Le réplicon donneur


1
2
3
4
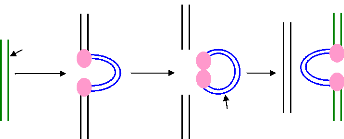
Transpososome
7 8
5
6
Intermédiaire de
transposition
9


Figure 3.4: Le mécanisme
général de la transposition conservative: 1: la transposase, 2:
l'ADN donneur, 3: le transposon, 4: liaison de la Tnp(pour transposase) aux ITR
du transposon, 5: l'ADN récepteur, 6: rapprochement des ITRs pour former
le transpososome, 7: détachement du transposon de son hôte en se
liant à la transposase et la formation de l'intérmédiaire
de transposition, 8: liaison de la transposase à la séquence
cible de la molécule réceptrice, 9: intégration du
transposon dans son nouveau hôte.
1' La transposase et son mode d'action:
Les transposases sont peu conservées dans leur
structure primaire, mais contiennent très souvent un motif
conservé D,DxE/D où le résidu glutamate (E) ou aspartate
(D) est généralement séparé des deux premiers
résidus aspartate par un nombre connu d'acide aminé
fréquemment conservé pour les membres d'une même famille,
ce motif est impliqué dans la liaison de deux ions métalliques
bivalents (magnésium ou
calcium) et fait partie du site catalytique de l'enzyme, le mode
d'action de la transposase est généralement comme suit:
1' Les ions bivalents piégés par le motif
D,D-E/D, interagit avec les extrémités de l'élément
et fragilise un pont phosphodiester qui devient plus sensible à une
attaque nucléophile par une molécule d'eau qui libère des
extrémités 3'-OH .
1' Une seconde attaque se lance contre les ponts phosphodiester
de l'ADN cible y libérant des extrémités 5'-P
sortantes.
1' Selon que la transposase a clivé ou non
l'extrémité 5' du transposon, la transposition sera conservative
ou réplicative. Dans le premier cas, les deux brins du transposon se
désolidarisent simultanément du site donneur et la
synthèse d'ADN s'arrête au niveau du transposon qui n'est pas
répliqué, en revanche lorsque la transposition est
réplicative, le second brin du transposon n'est pas clivé, la
synthèse à l'extrémité 3'-OH de la cible se
poursuit à travers le transposon et le duplique, en engendrant un
co-intégrat (Shao et Tu, 2001; Merlin et Toussaint, 1999).
I.2.2. Les transposons eucaryotes:
Après la découverte des éléments
de contrôle par Barbara McClintock, plusieurs transposons eucaryotes ont
été identifiés chez divers organismes comme la drosophile,
les levures, les nématodes et divers vertébrés y compris
l'homme. Comme le cas des séquences d'insertion bactériennes, les
transposons eucaryotes contiennent des ITRs qui flanquent la région
codante pour la transposase qui catalyse la transposition d'une manière
réplicative ou conservative. Ils sont divisés en plusieurs
familles individualisées par des similarités de séquence
ainsi que dans la séquence du site cible d'insertion (In Lodish et
al., 2004). Ils sont également divisés en
éléments autonomes pour leur transposition et nonautonomes qui se
déplacent via une transposase d'un élément autonome qui
appartient à la même famille, les familles les plus connues
sont:
1. Hat (h:
hobo de Drosophila melanogaster, a:
Ac du maïs, t: Tam3 du
antirrhnium majus )
2. Le groupe des éléments
PiggyBac.
3. Le groupe des éléments
P.
4. Le groupe des
MITEs/PIF/Harbinger
5. Le groupe EN/Spm
6. L'élément helitron
7. Le groupe Mutator (Mu)
8. Le groupe de la super famille
IS630/Tc1/maT/mariner
9. La famille Polintons(Mavericks)
10. La famille des éléments
Merlins
11. La Super famille des Transib
I.2.2.1. Le groupe des éléments hat:
Ils sont des transposons endogènes mobiles (In
Léonard, 2005), ces éléments contiennent un seul ORF qui
code la transposase, leurs ITRs sont généralement courts
(11-17pb), l'insertion de l'un de ces éléments dans le site cible
conduit à une duplication

Halaimia-Toumi, 2006). Les éléments de
ce
groupe représentent entre eux un
domaine très conservé
constitué de 50aa
situé sur la région C-terminal de
la
transposase, ce domaine est impliqué
dans le
phénomène de dimérisation de
Figure 4: structure de l'élément
Ac. Modèle
d'après
www.scibd.com
de 8pb (Renault et al. , 1997; in
ITR
ITR
deux molécules de transposase pendant le processus de
transposition (In Halaimia Toumi, 2006).
Chez le maïs, les éléments Ac
(pour activateur) ont une longueur de 4563, l'ARNm de 3500 pb transcrit
à partir de ces éléments comprend 5 exons, il code une
transposase de 807aa (figure 4) (Renault et al., 1997). La
présence de l'élément Ac au sein du gène
de la synthèse de l'anthocyane conduit à l'apparition de
nouvelles mutations susceptibles d'être reversée suite au
déplacement d'Ac, ces mutations sont exprimées par une
couleur tacheté des grains (où Ac est inseré)
avec la présence des grains de couleur normale (là où la
mutation est reversée). L'élément Ds est un
dérivé de l'élément Ac par
délétion du gène de la transposase, donc cet
élément ne peut jamais être transposé qu'à la
présence d'Ac c'est-à-dire qu'en absence d'Ac,
Ds reste en place et on n'observe pas de réversion de la
mutation induite. En revanche, lorsque Ac est présent
l'élément Ds se transpose en trans via le
transposase d'Ac ce qui permet l'évacuation du Ds sous
forme d'un transposon complexe permettant au gène de l'anthocyane de
retrouver sa fonction (In Watson et al., 1994 ).
I.2.2.2. Le groupe des éléments PiggyBac:
Le premier élément de cette famille a
été découvert chez le baculovirus qui a
infecté des cultures des cellules du lépidoptère
Trichoplusiani, ils sont d'une longueur comprise entre 2.3-6.3 kb,
avec des ITRs d'une taille 12-19 pb, leur site d'insertion est
caractérisé par la séquence conservée TTAA (In
Halaimia Toumi, 2006). Le seul élément autonome de cette famille
est appelé PiggyBac, le séquençage
moléculaire a montré qu'il est un transposon de 2500pb et porte
des ITRs parfaites de 13pb, en outre ils existent des ITRs internes
asymétriquement localisées aux extrémités de
l'élément, la transposase (64kDa) est codée par deux ORFs
traduits comme un seul ORF, son motif catalytique est estimé
d'être DDE?. Les éléments PiggyBac ont
été caractérisés dans le génome
d'espèces diverses, depuis les insectes jusqu'aux mammifères
(Feschotte et Pritham, 2007; in Halaimia Toumi, 2006).
I.2.2.3. Le groupe des éléments P:
La dysgénésie hybride est une singularité
génétique à certains souches de Drosophila
melanogaster, ses propriétés s'expliquent par
l'activité d'éléments
transposables, plusieurs syndromes sont connus chez cette
espèce. La dysgénésie
de type P, est bien comprise aux plans
génétiques et moléculaires (In Singer et Berg, 1992), le
croisement des mâles de cytotype (type cellulaire) P (
Paternel ayant de 30-50 copies du transposon P) avec des femelles de
type M (maternel dépourvu de P) produit une descendance F1
stérile caractérisée par des gonades atrophiées, le
croisement réciproque produit une descendance fertiles ( Renault et
al., 1997) étant donnée que la cause principale de cette
mutation ou dysgénésie est la transposition de
l'élément P dans la lignée germinale de la
mouche (Koslovski Sassi et al., 2005). Comme dans le
cas du système Ac/Ds, cette famille comporte des
éléments autonomes et d'autres nonautonomes qui
|
|
|
L'élément P
ARNm1 ARNm2
|
|
|
L'intron 3 reste
|
|
|
L'ARNm
|
|
somatique Cadre de
Represseur
|
lecture
de 66kDa
|
|
L'ARNm
|
|
germinal Cadre de lecture
transposase
|
de 88kDa
|
|
Figure 4.1 : La structure et
l'épissage
somatique et germinale de l'élément
P
(D'après
www.scribd.com)
|
requièrent la transposase des éléments
autonomes.
Les éléments P les plus longs sont des
séquences de 0.5-2.9kb, ils présentent des
répétitions terminales inversées de 31pb.
L'élément P autonome porte 4 exons, il est transcrit
dans les cellules somatiques et germinales, mais ne transpose que dans la
lignée germinale parce que la transposase active de 88KDa n'est
synthétisée que dans cette catégorie cellulaire, dans la
lignée somatique la protéine synthétisée
résulte des 3
premiers exons et sert de répresseur de la
transposition de 66KDa (figure 4.1). La transposition des
éléments P est conservative et se produit par un
mécanisme proche de celui des IS non réplicatifs
(Renault et al., 1997; In Watson et al., 1994).
I.2.2.4. Le groupe des MITEs/PIF/Harbinger:
I.2.2.4.1. Les MITEs : Pour
"Miniature Inverted Repeats Transposable Elements" sont initialement
considérés parmi les éléments non-classés
mais par son homologie avec les éléments de la famille
Tc1/mariner, ils sont désormais intégrés
aux éléments de la classe 2 (in Rivière, 2001). Ils sont
des petites séquences d'une taille (100-500pb) et avec un TSD (pour
Target Site Duplication, ou la duplication du site cible) de 3pb riche en A et
T, et ne possèdent aucune capacité codante, ces
éléments sont donc non autonomes, et semble transposé en
trans. Les MITEs sont décrites dans une
variété d'espèces animales et végétales.
Dans les plantes ces éléments peuvent être divisés
selon les similarités qui existent dans leurs ITRs et TSD en deux
groupes principaux:
· Les Tourist like MITEs: l'élément
le plus répandu est appelé tourist qui a été
découvert sous la forme d'une insertion de 128pb dans un gène du
maïs.
· Stowaway like MITEs (in Halaimia Toumi, 2006;
Zhang et al., 2001)
I.2.2.4.2. Les PIFs (pour P
Iinstability Factor) : ils ont été
caractérisés par des insertions dans le gène R de
régulation de la production des anthocyanes dans divers tissus du
maïs. L'insertion de ces PIFs dans le second intron bloque la
production qui est rétablie après excision,
révélant un système de transposition par couper-coller.
PIF-12 a été séquencé, mais les
ORFs présentes ne ressemblent à rien de connu.
PIF-12 et PIF-5.2 transposent activement,
mais sont cependant non autonomes. Ceci laisse donc perplexe. La solution est
apparue quand l'équipe de Wessler a séquencé un nouvel
élément actif de la famille, PIFa. C'est un
élément de 3728 pb qui participe à la mobilisation
d'autres éléments de la famille. Il code, en effet, une ORF de
331 acides aminés codant potentiellement une transposase qui ressemble
à celles présentes dans beaucoup d'autres eucaryotes très
disparates. Cette transposase ressemble également de loin à celle
d'IS5. Un fait curieux est que cet élément est plus
court que PIF-5.2. Une version miniature de PIF
d'une taille de 364 pb est présente à environ 6 000 exemplaires
dans la garniture
haploïde du maïs, mais elle est absente dans les
génomes du riz et du sorgho. . Elle a les caractéristiques des
MITEs de type Tourist. Cet élément est donc
très probablement un élément dérivant des
éléments plus grands et fortement simplifié par
délétion. Il est donc obligatoire de réunir biologiquement
ces éléments de taille pourtant très différentes.
Leur site d'insertion est une séquence de 9 pb (dont seul leTTA1 central
est dupliqué lors d'une insertion) qui est un palindrome imparfait, Il
en existe environ 10 000 dans le génome du maïs. Il reste donc des
places libres dans le génome pour une mobilité. Les PIFs
sont probablement à l'origine et contribuent à la mobilisation
des MITEs de type Tourist.
I.2.2.4.3. Harbinger : il est un
transposon de 5 382 pb découvert lors du séquençage total
d'Arabidopsis. Il possède des répétitions
terminales de 25 pb, crée une répétition de 3pb au site
d'insertion et contient deux phases de lecture potentielles (ORFs) dont l'une
code une transposase ressemblant à celle de la séquence
d'insertion bactérienne IS5. Cet élément est donc
probablement autonome. Les éléments
PIF/Harbinger code pour une transposase
caractérisée par une triade catalytique DD(35-37/47-48)E et
constitué par 350-550aa (Feschotte et Pritham, 2007).
I.2.2.5. Le groupe En/Spm:
L'élément En/Spm
(Enhancer/Supressor-Mutator) a été découvert par
Barbara McClintock et Peterson en 1953, il a une longueur de 8.3kb avec une
capacité de coder au moins deux transcrits alternativement
épissés de 5.8kb et 2.5kb à partir de deux ORFs, le
dernier est appelé TnpA, il contient 11 exons du gène
TnpA dont il tire son nom et il est 100 fois plus fréquent que
le premier. Le transcrit de 5.8kb est nommé TnpD, outre les 11
exons il comporte aussi un large ORF dans sa région 5'. Le TnpA
code une protéine nécessaire pour le phénomène
excision/intégration tandis que le transcrit TnpD code pour une
protéine de 67kDa qui fonctionne comme une protéine de liaison
à l'ADN (DNA binding protein). La séquence de l'ITR de cet
élément est hautement conservée (5'- CACTACAAGAAAA) et une
duplication du site cible résulte au cours de l'intégration de
cet élément dans son nouveau site hôte (In Halaimia Toumi,
2006).
|
I.2.2.6. L'élément Helitron
:
|
|
|
|
Il a été identifié par Kapitonov et Jurka
(2001) dans les génomes d'Arabidopsis thaliana, Caenorhabditis
elegans et Oryza sativa. Les analyses << in silico
»
|
|
|
|
|
|
ont montré
représente
|
que 2%
|
cet du
|
élément
génome
|
Figure 4.2: Structure de
l'élément Helitron du
maïs. (D'après
www.plantcell.org)
|
d'Arabidopsis et caenorhabditis. Helitron
est un transposon de 500-15000 pb ne possédant pas des ITRs mais porte
des motifs 5'-TC et 3'-CTRR, des structure palindromique peuvent former une
structure de type << hairpin loop » (épingle à
cheveux) de 16-20pb proche de la région 3' (figure 4.2). Les
Helitrons autonomes codent une hélicase et une ou deux copies
d'une nucléase/ligase, ils s'insèrent précisément
entre les deux nucléotides A et T de l'hôte et sont
supposés de transposer selon un mode de réplication en cercle
roulant semblable à celui décrit chez des transposons
bactériens (IS91, IS801, IS1294) (in Halaimia Toumi, 2006).
I.2.2.7. Les éléments Mutator (Mu)
Les éléments transposables de type
Mutator ont été connus par Robertson parce qu'ils ont
élevé la fréquence de mutation chez le maïs Zea
mays, les analyses moléculaires de ces éléments ont
révélé la présence d'au moins 8 classes de
Mutator (Mu1 à Mu8) autonomes et
nonautonomes, ils présentent des ITRs de 210pb extrêmement
conservées et un TSD de 9pb, leur taille et comprise entre 1000-2200pb.
MuDR est le premier élément Mu autonome qui a
été identifié. Il est de 4.9Kb et peut être
transcrit en deux ARNm différentiellement épissés, ils
sont appelée mudrA et mudrB, le premier code un
polypeptide de 823 aa appelé MURA et qui fonctionne comme une
transposase, le deuxième code la MURB indispensable pour l'excision de
MuDR mais son rôle dans le phénomène de la
transposition reste encore inconnu (Bénito et Walbot, 1997).
I.2.2.8. Le groupe des éléments de la super
famille IS630/Tc1/maT/mariner :
La taille des éléments de cette super famille
peut atteindre des milliers de paires de bases, les ITRs sont de 19 à
69pb, la transposase est codée par un gène
généralement sans intron, leur site d'insertion est le
dinucléotide TA (.Halaimia.Toumi et al., 2004). Les
éléments de la famille IS630 qui existent chez les
bactéries code une transposase qui contient une triade catalytique de
type DDXE (X indique une distance variable). Les TLEs (Tc1
Like-Elements) identifiés dans les champignons, les
vertébrés et les invertébrés portent le motif D,
D34E. Les MLEs (mariner like-elements) présente la triade D,
D34D à l'exception de Soymar1 qui est identifié dans le
soja ainsi que dans le riz et les nématodes et qui est
caractérisé par le motif D, D39D (Shao et Tu, 2001). La famille
des maTs est une famille intermédiaire entre les MLEs
et TLEs (<< ma >> pour mariner et
<< T >> pour Tc1) et qui a été
définie par Claudianos et al. (2002) possède le motif
D,D37D (In Halaimia Toumi, 2006). D'après les analyses des domaines
catalytiques un autre élément appelé pogo
décrit chez Drosophila melanogaster et contient le motif D,D30D
peut être classé parmi les membres de la famille
IS630/Tc1/maT/mariner (Shao et Tu, 2001).
(a)
Tc1
(D,D34E)
maT
(D,D37D)
ITmD41D
(D,D41D)
mariner
(D,D34D)
Plantes
(D,D39D)
Bactéries/ Champignons
(D,DxE/D) (x: distance
variable)
(b)
41
Figure 4.3 : Les différentes familles de
la superfamille
IS630/Tc1/ITmD41D/maT/mariner/Pogo/Ant1.
(a) : Positionnement des familles
IS630/Tc1/ITmD41D/maT/mariner/Ant1
(c) : Positionnement de la famille Pogo par rapport
à mariner et Tc1. (in Halaimia Toumi, 2006)
I.2.2.9. La famille Polintons(Mavericks) :
Les éléments de cette famille ont
été découverts plus récemment (2006), ils sont les
transposons les plus complexes identifiés jusqu'ici. Les polintons,
ou aussi les mavericks, sont des géants
éléments d'environ 10-20 kb qui peuvent coder au moins pour 10
protéines différentes dont une ADN polymérase B (POLB),
une intégrase rétrovirale (Int), une protéase
adénovirale (PR), et une ATPase. Ils ont un TSD de 6pb qui flanque des
ITRs longs contenant des terminaisons 5'-AG et TC-3' (Figure 4.5). Kapitonov et
Jurka (2006), Pritham et al. (2006) ont développé un
modèle de transposition unique à ces éléments,
selon ces auteurs la molécule d'ADN de polinton/mavericks
excisée par l'intégrase, au cours de la réplication du
génome de son hôte, sert comme une matrice pour une
synthèse extra-chromosomique du brin d'ADN complémentaire par
l'ADN polymérase B, la molécule d'ADN double brin s'insère
dans le génome sous l'action de l'intégrase. Ces
éléments sont largement répandus dans les génomes
eucaryotes comme les génomes des insectes, des poissons et aussi des
oiseaux (Kapitonov et Jurka, 2006 ; Pritham et al., 2006)
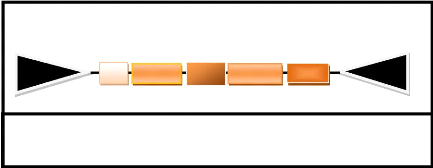
ITR Int ORFs Pro ATPase POL ITR
Figure 4.4 : Structure des
éléments de la famille Polintons/Mavericks
(D'après Tempel, 2007)
I.2.2.10. La famille des éléments Merlins
:
Ces éléments sont des homologues des
séquences d'insertion IS1016, le premier élément
Merlin était identifié en octobre 2001 dans le
nématode Caenorhabditis briggsae, il est une séquence
d'environ 1914pb et possède un TSD de 8pb (Feschotte, 2004). Les
éléments merlin décrits sont des séquences
de 1.4-3.5 kb, avec des ITRs entre 21 et 462 pb flanqués par des TSD de
8-9 pb, leurs ORFs codent pour une transposase de
270-330 aa caractérisées par le motif DD(36-38)E.
Ils sont présents dans les génomes de divers
invertébrés et vertébrés (Feschotte et Pritham,
2007)
I.2.2.11. La Super famille des Transib :
Cette super Famille a été reconstituée
in silico à partir des éléments
dégénérés dans les génomes de Drosophila
melanogaster et Anopheles gambiae (Chen et Li, 2007), ils sont
des éléments d'une longueur comprise entre 3 et 4kb bordés
par des ITRs de 9- 60pb, ils produisent lors de son insertion un TSD de 5pb. Un
élément Transib code pour une transposase de 650-700aa
avec une triade catalytique de type DD(206-214)E (Feshotte et Pritham, 2007).
Kapitonov et Jurka (2005) ont démontré que les protéines
RAG1/2 impliquées dans la recombinaison V(D)J sont des
dérivés de ces éléments (Kapitonov et Jurka, 2005).
Les transposons Transib sont aussi présents dans le
génome de l'Hydre, de l'oursin et du moustique de la fièvre jaune
(Chen et Li, 2007).
II. L'impact des éléments transposables sur
le génome :
L'intérêt porté aux éléments
transposables se focalise sur deux points principaux : l'évolution des
éléments transposables eux-mêmes et leur effet sur les
génomes des hôtes. Au cours des dernières années,
les auteurs ont orienté leurs recherches sur ce deuxième point,
c'est-à-dire sur le rôle des éléments transposables
dans l'évolution de la structure et du fonctionnement du génome
de l'hôte (Anxolabéhère et al., 2000). Ce ne sont
cependant pas des éléments génétiques purement
égoïstes dans la mesure où l'acquisition de nouveaux
gènes sous la forme d'élément transposable est une voie
majeure d'adaptation des organismes qui les hébergent (Merlin et
Toussaint, 1999), d'une manière générale les
éléments transposables influencent le trajet
évolutionnaire de leurs hôtes selon 3 façons principales
:
/ La modification de la structure et la fonction des
gènes par le processus excision/intégration.
/ L'induction des réarrangements
chromosomiques.
v' Ils constituent une source importante du
matériel codon et non-codon qui permettent l'émergence de
nouveauté génétique (néogène et des
régions régulatrices) (Feschotte et Pritham, 2007).
II.1. La modification de la structure et la fonction des
gènes :
L'intégration d'un élément transposable
à proximité d'un gène a un effet modeste voire novateur au
sens de l'évolution. Dans certains cas, l'intégration conduit
à un changement de la séquence du gène hôte et
à la production d'une protéine modifiée, par exemple chez
le maïs l'insertion d'un élément au début d'un exon
génère un nouvel site de reconnaissance qui produit l'excision du
début de cet exon avec l'intron adjacent. La protéine
codée est ainsi plus courte de 30 acides aminé mais reste
fonctionnelle et produit une modification de la couleur des grains (Renault et
al., 1997). Un autre exemple bien étudié est le cas du
gène nia2 du Tabac, ce gène code l'apoenzyme de
la nitrate réductase (NR), l'analyse des mutants instables de
phénotype [NR-] permet de mettre en évidence
l'insertion du rétrotransposons à LTR Tnt1, dans une
région codante de ce gène. Le phénotype [NR-]
est dû à des modifications qui dépendent de l'orientation
de l'élément par rapport au sens de transcription du gène
: (figure 5)
v' Lorsque l'élément Tnt1 est
inséré en orientation directe par rapport au sens de
transcription, des transcrits tronqués sont obtenus correspondant
à l'arrêt de la transcription dans les LTR 3' et 5'.(figure 5
A).
v' Par contre quand l'élément est
inséré en orientation inverse par rapport au sens de
transcription, un transcrit composite est obtenu correspondant au transcrit
classique du gène plus la séquence de rétrotransposon. Ce
transcrit donnera, après traduction, une protéine tronquée
s'arrêtant au niveau da la LTR3'. (Figure 5 B).
v' Outre que la production d'un transcrit composite,
l'excision d'un intron soit perturbé par la présence de
l'élément, là encore une protéine tronquée
est obtenue (Figure 5 C) (In Luchetta et al., 2005).
UTR5'
Figure 5: Effet de l'insertion du
rétrotransposon à LTR, Tnt1, sur les transcrits du
gène
nia2 codant la nitrate réductase chez
Nicotiana tabacum
UTR5'
UTR5'
B
A
C
Exon1 Exon2 Exon3 Exon4 UTR3'
Exon1 Exon2 Exon3 Exon4 UTR3'
Exon1 Exon2 Exon3 Exon4 UTR3'
Transcrit 1.8kb
(d'après Luchetta et al. ; 2005)
Transcrit 6.7kb
Transcrit 10kb
Transcrit 12kb
Dans des cas plus graves, l'intégration de
l'élément induit une mutation caractérisée par
l'acquisition d'une nouvelle fonction, chez E-coli le gène
sauvage Eco DXXI code pour une enzyme de restriction de type
I qui reconnait spécifiquement les séquences de
type TCA(N7)RTTC, une mutation provoquée par l'insertion du Tn5
dans le gène correspondant, conduit à une modification
sévère de la fonction enzymatique : l'ancien site n'est plus
identifié et la nouvelle enzyme reconnait les séquences de type
TCA(N8)TGA (Renault et al., 1997).
II.2. Les réarrangements chromosomiques :
Les mécanismes de transposition des
éléments transposables impliquent des processus de coupures
double brin et de réparation. Ce qui induit différents types de
réarrangements chromosomiques, soit en liaison avec leur
mobilité, soit parce qu'ils
fournissent des séquences répétées
favorisant des recombinaisons intra-génomiques voire
intra-chromosomiques. L'excision imprécise de certains
éléments génère ainsi de nombreuses
délétions ou translocations dans le génome hôte
adjacent. Chez la drosophile plus de 50kb du génome hôte peuvent
ainsi être transporté lorsqu'ils sont pris en sandwich entre des
éléments transposables. D'autre part, les recombinaisons entres
séquences répétées produisent des
délétions ou des inversions selon le sens direct ou
inversé des motifs répétés. Ces
réarrangements peuvent n'avoir aucune incidence sur le phénotype
externe du porteur, ou au contraire provoquer une mutation visible lorsque les
séquences d'un gène sont déplacées et mises sous le
contrôle d'un autre promoteur (Anxolabéhère et
al., 2000 ; Feschotte et Pritham, 2007 ; Renault et al.,
1997).
II.3. Les éléments transposables et la
régulation de l'expression du gène :
Les éléments transposables possèdent un
répertoire important de séquences régulatrices qui
confèrent aux gènes de nouveaux profils d'expression
spatio-temporels, en s'intégrant le plus souvent dans les régions
5' noncodante des gènes. Au cours de l'évolution, une
recombinaison entre les LTRs d'un rétrotransposon peut exciser la
majorité de l'élément en ne laissant qu'un seul LTR
à proximité du gène de l'hôte. Les signaux de
régulation du LTR sont alors capables de prendre le contrôle de ce
gène. La modification de l'expression d'un gène peut poser
cependant la question de la survie de la cellule et de l'organisme en l'absence
de la fonction primitive. Une des solutions repose en la duplication du
gène avant que la modification n'intervienne, celle-ci ne provoquera
donc pas la perte de la fonction préexistante et au cours de
l'évolution l'organisme pourra même en acquérir de
nouvelles (Renault et al., 1997).
II.4. Néogènes (nouveaux gènes) et
recrutement d'élément transposable :
Les éléments transposables peuvent participer
à l'évolution des gènes non seulement par la
création de systèmes de régulation mais aussi en
fournissant de nouvel types de gènes ainsi que de nouvelles fonctions
cellulaires. La transition moléculaire d'une séquence codante
apparentée à un ancien élément transposable vers un
gène immobile a été présentée comme une
« domestication moléculaire ». Elle correspond au recrutement
d'élément transposable par génome, deux types de
recrutement peuvent être
observés : le recrutement de fonction structurelle et
recrutement de fonction enzymatique (Anxolabéhère et
al., 2000 ; in Luchetta et al., 2005).
II.4.1. Recrutement de fonctions structurelles :
Le cas le plus étudié est
l'intégrité des extrémités chromosomiques chez la
drosophile. A l'inverse de la plupart des organismes, cette
intégrité n'est pas assurée par la
télomérase qui catalyse la synthèse des courtes
répétitions terminales, elle est assurée par deux
rétrotransposons de types non rétroviral (sans LTR), les
LINEs TART (pour Telomerase Associated RetroTransposon) et
Het-A, ils sont d'environ 7kpb, les TART codent pour leur
propre transcriptase inverse (TI), ils sont donc autonomes tandis que les
Het-A doivent être transposés en trans par une
TI fournit par un TART ou d'autres rétrotransposons.
Après chaque cycle de réplication, les extrémités
chromosomiques sont érodées par une réplication
incomplète, ensuite elles sont régénérées
grâce à la rétrotransposition des TART et
Het-A. De façon tout à fait remarquable, un
équilibre dynamique s'est installé dans ce système
où le taux moyen d'érosion est égal au taux moyen de
transposition. Par ailleurs, structurellement le domaine transcriptase inverse
de la télomérase présente des similarités avec les
transcriptases inverses des rétrotransposons ce qui soulève la
question de l'ancêtre commun : était-ce la RT d'un
rétrotransposon ou celle d'une télomérase ?
(Anxolabéhère et al., 2000 ; in Luchetta et
al., 2005 ; Renault et al., 1997).
II.4.1. Recrutement de fonctions enzymatiques :
L'exemple le plus frappant d'un tel recrutement est
probablement apporté par le système immunitaire des
vertébrés. Ce système doit être capable de
reconnaître des millions d'antigènes différents, cette
diversité est générée au niveau des lymphocytes au
cours de leur développement, suite à des remaniements de l'ADN au
niveau des gènes codants les immunoglobulines et les récepteurs
des cellules T, le processus conduisant à ces remaniements a
été appelé Recombinaison V(D)J (In Luchetta et
al., 2005 ; in Watson et al., 1994). Dans ce système,
deux protéines RAG1 et RAG2, sont essentielles pour la recombinaison,
elles possèdent en effet à la fois la propriété de
reconnaître des séquences nucléotidiques spécifiques
correspondant à des signaux de recombinaison
placés au voisinage des segments V, D et J, et celle de
cliver de l'ADN immédiatement à proximité de ces signaux,
ces derniers, composés d'héptamères et de nonamères
très conservés et séparés les uns des autres par
des séquences relativement homogènes de 12 ou 23
nucléotides, évoquent les répétitions terminales
inversées de nombreux éléments transposables. Donc RAG1 et
RAG2 se comportent exactement comme des transposases c'est-à-dire
reconnaissance de sites de coupures et activité endonucléase. Par
ailleurs le mécanisme de réparation de la coupure double brin de
l'ADN après excision de fragments V(D) ou (D)J est similaire à
celui observé pour la réparation chez les transposons Ac/Ds
de Zea mays . L'ensemble de ces observations suggèrent que les
gènes rag1 et rag2 dérivent d'un ancien
élément transposable (l'élément Transib)
de classe 2 possèdant des ITR semblables aux actuels signaux de
recombinaison entre les séquences V, D et J. L'élément
à l'origine de rag1 et rag2 serait devenu immobile par
la perte de ses ITR (Figure 5.1) (Anxolabéhère et al.,
2000 ; in Luchetta et al., 2005).

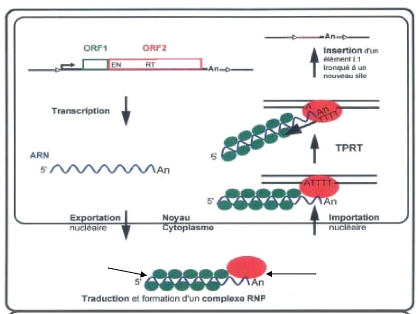
Figure 5.1 : Origine du système
immunitaire des mammifères par coaptation de l`activité
transposase d`un élément transposable : Les
éléments transposables à l'origine de la recombinaison
V(D)J devaient posséder des répétitions terminales
inversées (RTI) comme ceux des transposons de type II, et deux
gènes (rag1 et rag2) codant les deux propriétés
essentielles d'une transposase : la reconnaissance des sites de coupures et
l'activité endonucléasique. Selon le modèle, les
recombinases RAG1 et RAG2 et les motifs répétés du
système V(D)J dérivent de ces anciens éléments
transposables (Anxolabéhère et al., 2000).
Élément transposable d'origine
Cluster des gènes immunoglobulines
V
RTI ITR
RTIa
RAG1
RAG1
ITRa
rag]
rag]
D RTIa ITRa J
rag2
rag2
RAG2
RAG2
III. La régulation de la transposition :
Il est possible de constater que le nombre
d'élément transposable est régulé et ce à
différents niveaux. En effet, des analyses populationnelles
intraspécifiques ainsi que des analyses interspécifiques montrent
que le nombre d'éléments transposables dépend de
l'espèce voire de la population considérée, et de type de
l'élément lui-même. L'étude de la transposition a
permis de mettre en évidence différents mécanismes
expliquant la
régulation du nombre de copies, ceux assurés par
l'élément et d'autre par le génome hôte (In Luchetta
et al., 2005).
III.1. L'autorégulation de la transposition :
Les éléments transposables utilisent
différents modèles pour effectuer une telle régulation
pour leur transposition, soit par la titration de la transposase, ou par des
régions régulatrices, la synthèse de répresseurs ou
aussi l'inhibition par surproduction et enfin la Co-suppression (In Luchetta et
al., 2005)
III.1.1. Titration de la transposase par les
séquences délétées:
Ce premier modèle a été
évoqué pour les éléments de la classe II, en effet,
la transposase issue directement et seulement des éléments
complets autonomes assure le déplacement des éléments
complets en cis et les éléments
délétés (nonautonomes) en trans. Alors s'il
existe un plus grand nombre d'élément
délétés que les éléments complets, la
protéine sera alors insuffisante car titrée par les
éléments délétés, la quantité par
élément sera alors limitée pour assurer la transposition,
d'une façon générale ce modèle n'a jusqu'à
présent jamais pu être confirmé ou infirmer (in Luchetta et
al.; 2005).
III.1.2. Les régions régulatrices :
Chez les rétrotransposons à LTR, les
séquences nécessaires à une transcription basale sont
localisées dans les LTR où une transcription bidirectionnelle
(transcription du brin sens et du brin antisens) peut se faire, ceci aboutit
à la production des messagers antisens pouvant créer des hybrides
ARN-ARN avec les messagers sens et induire une répression.
Néanmoins, la transcription du brin sens est majoritaire, dans un
rapport 20:1 (In Rivière, 2000),
III.1.3. Synthèse de répresseurs :
Le cas le plus argumenté d'un tel modèle est le
cas de l'élément P responsable de la
dysgénésie des hybrides chez Drosophila melanogaster ,
le croisement entre des femelles M dépourvus de P
et des mâles P qui en possèdent aboutit
à une génération F1 stérile (les gonades sont
atrophiés), le croisement réciproque ne produit aucune
mutation
ce qui montre clairement l'existence d'une régulation
à effet maternel liée à la présence de
l'élément P dans la lignée germinale. Ainsi,
l'enzyme principale de la transposition de l'élément P
est une transposase de 87KDa obtenue depuis l'ARN complets après
épissage de tous les introns, IVS1, 2 et 3. Cette protéine est
spécifique de la lignée germinale car le dernier intron IVS3
n'est pas épissé dans la lignée somatique. De ce fait,
pour la lignée somatique, la traduction produit une protéine
tronquée de 66KDa en raison de la présence d'un codon stop dans
ce dernier intron. Cette protéine joue le rôle d'un
répresseur de la transposition en agissant sur le même site
nucléotidique de la transposase (in Luchetta et al.; 2005).
III.1.4. Inhibition par surproduction:
Appelé aussi OPI (pour Over Production
Inhibition), ce mécanisme de régulation a été
mis en évidence avec les transposons mariner de la drosophile.
Selon ce modèle la transposase de l'élément agirait sous
forme d'un monomère. Cependant quand, la production de la transposase
augmente, des multimères pourraient se former, l'activité
transposase serait quant à elle inversement proportionnelle au niveau de
polymérisation. Dans un tel schéma, plus il y a production de
transposase, plus il y a polymérisation et moins la transposase est
efficace (in Luchetta et al.; 2005).
III.1.5. La Co-suppression:
Elle était mise en évidence vers les
années 1995 par son importance dans la transgénèse du fait
que le nombre de séquences transgéniques insérées
est inversement proportionnel avec leur expression. Il semble que la
Co-suppression puisse être transcriptionnelle ou
post-transcriptionnelle:
III.1.5.1. La Co-suppression transcriptionnelle :
La diminution de la transcription se fait par des interactions
entre les copies homologues des éléments transposables qui
existent sur le même génome. Ainsi, ces copies pourraient se
rapprocher et créer des régions hétérochromatiques
(où la compaction de la chromatine est importante) inaccessibles
à la machinerie de la transcription ou favoriser la méthylation
des séquences.
III.1.5.2. La Co-suppression post-transcriptionnelle:
Dans ce cas, la transcription n'est pas affectée, mais
les messagers synthétisés sont ainsi susceptibles de former des
duplex, sensibles à une dégradation par les RNaseH (In
Rivière, 2000).
III.2. Régulation de la transposition par les
facteurs de l'hôte :
Le génome hôte peut aussi contribuer à la
régulation de la transposition et c'est par différents
mécanismes, les plus importants sont mentionnés ci-dessous :
III.2.1. L'effet de position :
Il est connu depuis longtemps que la position des gènes
eucaryotes peut constituer un facteur capable de modifier leur expression. En
ce qui concerne les éléments transposables, l'expression d'une
séquence peut être modifiée du fait de la proximité
d'une séquence << enhancer >> ou <<
silencer >>, celle-ci ayant la particularité de pouvoir
agir à une grande distance, et dans les deux orientations. Ainsi il est
probable que lorsqu'un élément soit inséré à
l'intérieur d'un gène, il peut être transcrit avec le
gène dans lequel il est inséré par un mécanisme de
<< readthrough » (l'ARNpolymérase continue la
synthèse au-delà du stop de transcription de
l'élément) ensuite l'élément est
épissé du messager, comme s'il s'agissait d'un intron (In
Rivière, 2000).
III.2.2. La régulation
épigénétique :
La régulation épigénétique est un
phénomène qui ne modifie pas la séquence du gène
mais touche principalement la conformation de la chromatine et la
méthylation des séquences. Toutefois, il est aussi possible que
cette régulation fasse intervenir des facteurs cytoplasmiques. La notion
d'hétérochromatinisation observée chez la drosophile
correspond à un état très condensé du
matériel génétique. L'hétérochromatine est
constituée essentiellement de séquences hautement et moyennement
répétées. Du fait de son importance compaction, elle
constitue un cimetière pour les éléments transposables, ce
phénomène n'est pas remarqué chez les plantes et les
vertébrés. Un autre mécanisme de régulation est
utilisé par ces organismes, la méthylation des séquences.
La méthylation est un processus qui concerne la cytosine suivie d'une
guanine (se sont les îlots CpG),
plus ces séquences sont méthylées moins
le gène porteur est exprimé, le cas le bien connu est le cas de
l'élément IAP de la souris, ces éléments
sont exprimés principalement dans les testicules parce qu'ils sont
hypométhylés dans ce tissu, en revanche, ils sont
hyperméthylés dans les autres tissus et donc inactivés (In
Rivière, 2000).
IV. La transmission des éléments
transposables :
Dans la plupart des analyses détaillées des
éléments transposables, il est montré que leur
phylogénie ne suit pas toujours la phylogénie des espèces
hôtes. En effet, deux mécanismes illustrent la manière de
la transmission des éléments transposables entre les individus
:
IV.1. Le transfert vertical : C'est le cas le
plus simple, les éléments transposables sont transmis des parents
vers leurs descendances.
IV.2. Le transfert horizontal : Il suppose
que du matériel génétique peut passer d'une espèce
à une autre sans qu'il y ait fécondation, dont les vecteurs
peuvent être des virus, des bactéries intracellulaires ou encore
des parasites. (In Halaimia-Toumi, 2006). Trois situations sont
utilisées pour expliquer le transfert horizontal. La première
semble offrir la meilleure preuve, elle se repose sur la détection des
éléments transposables avec un grand degré de similitude
dans des taxons très divergents, les auteurs proposent
que ceci pouvait résulter d'un transfert horizontal
entre les ancêtres de ces espèces. La seconde situation se
réfère aux différences observées entre les
phylogénies des espèces établies par les
éléments transposables, et celles crées par un
caractère plus conventionnel tels que les gènes de structure. La
dernière situation s'appuie sur la distribution inégale des
éléments transposables, c'est-à-dire la présence
d'un élément transposable dans une espèce et son absence
dans une espèce soeur, cette démonstration se repose sur
l'assomption que la première espèce a acquis
l'élément par un transfert horizontal qui n'a pas impliqué
l'espèce soeur (Silva et al., 2004).
L'introgression, la polyspermie et divers vecteurs sont trois
mécanismes estimés d'être à l'origine du transfert
horizontal. L'introgression est la possibilité de l'hybridation des deux
espèces dont l'un des sexes est fertile ; aussi une grande partie du
matériel génétique d'une espèce peut être
transmis à l'espèce jumelle. La polyspermie est la
fécondation d'un oeuf par deux spermatozoides
d'espèce différentes, cela n'ai été jamais
clairement démontré. Les vecteurs qui peuvent participer dans ce
type de transfert sont les parasites et parasitoïdes, les baculovirus, les
éléments transposable et des bactéries comme les
rickettsies (In Halaimia Toumi, 2006).

I. Définition et Découverte:
Les éléments mariner sont des
transposons de la classe 2 appartenant à la super famille des
Tc1/maT/MLEs et à la famille des MLEs (pour
mariner like elements). Ils sont très largement répandus
dans les génomes des eucaryotes; ils sont dits "des ubiquistes". Le
premier élément mariner était découvert
par Jacobson et Hartl (1985) dans le génome de la mouche Drosophila
mauritiana parce qu'il y avait causé une mutation qui était
exprimé par une couleur mutée des yeux suite à
l'expérimentation suivante: (Hartl, 2001)
Un Croisement entre les deux espèces

Drosophila simulans × Drosophila
mauritiana
Une première génération hybride,
fertile et non mutée
Croisement entre les individus de la
1ère génération
Une 2ème génération
présentant une mutation instable dans les couleurs des yeux
Au niveau de l'allèle
wpch (pour white-peach)

Clonage de l'allèle
wpch
Observation d'une séquence
étrangère insérée dans l'allèle qui
était nommée mariner
Figure 6 : Résumé des
expériences conduisant à la découverte du transposon
mariner
(D'après Hartl, 2001)
I.1. La nomenclature des MLEs:
Les transposons mariner suivent actuellement la
nomenclature suivante:
· Xymarz:
X: la 1ère lettre du genre de
l'individu qui l'héberge.
y: la 1ère lettre de l'espèce de
l'individu qui l'héberge.
z: c'est un chiffre qui désigne l'ordre de
l'élément selon sa découverte. mar: pour
mariner
Exemple: le 1er élément mariner
qui a été découvert est appelé: Dmmar1
pour Drosophila mauritiana
mariner n°1 (Halaimia.Toumi et
al; 2004).
II. Structure et fonctionnalité des
éléments Tc1/mariner : II.1. Structure générale des
MLEs et TLEs:
Un MLE ou TLE typique est une
séquence d'une taille comprise entre 1300 et - 4000 pb, elle est
constituée d'un seul ORF (dépourvu d'intron ou avec un ou
plusieurs introns) codant la transposase (environ 345 aa). (In Augé
gouillou, 2003; In Toumi, 2001- 2002; Halaimia Toumi et al, 2004).Cet
ORF est bordé par deux ITRs d'une taille comprise entre 19-750 pb, les
5' ITR et 3' ITR sont séparés de l'ORF par une séquence
non traduite appelée UTR (UnTranslated Region), (figure 6.1) (In
Halaimia Toumi, 2006).

ITR5'
UTR
ITR3'

ORF (gène de la transposase)
ORF: cadre de lecture ouvert (Open Reading Frame)
ITR: répétition terminale inversée (Inverted
Terminal Repeat) UTR: région non traduite (UnTranslated Region)
Figure 6.1: La structure
générale des TLEs et des MLEs.
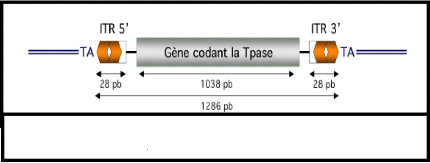
Figure 6.2: La structure d'un transposon
mariner, le cas du Mos1
(D'après
www.univ-lemans.fr)
II.1.1. Les mariner- like elements ou
MLEs: II.1.1.1. Structure nucléique:
La majorité des éléments mariner
décrits jusqu'ici sont d'environ 1300pb dont le gène de la
transposase (ORF) est d'environ 1050pb (Figure 6.2), la taille des ITRs varie
de 19pb (comme dans le cas de l'élément Acmar1 de
l'abeille apis cerana) à 69pb (élément Ahmar1
du lépidoptère Adoxophyes honmai) (In Halaimia
Toumi, 2006). D'une manière générale, les MLEs
présentent une homogénéité structurale plus que
celle des TLEs à l'exception de l'élément
Mcmar1, isolé du nématode meloidogyne chitwoodi
qui porte des ITRs de 355pb et un ORF qui code pour une transposase de 350 aa
(In Augé Gouillou, 2003).
II.1.1.2. Structure protéique:
La transposase active des MLEs est un polypeptide de
350aa organisé en deux domaines (In Toumi, 2001-2002; In Halaimia Toumi,
2006), un domaine N terminal responsable de la liaison de la protéine
aux ITRs et un domaine C terminal doué d'une activité catalytique
permettant la transposition (In Augé Gouillou, 2003). Ce domaine est
caractérisé par la présence d'une triade catalytique
D,D(34)D dont le premier et le second acide aspartique (D) est localisé
à proximité des positions 158 et 250 respectivement et le dernier
est séparé du second par 34 acides aminé (In Toumi,
2001-2002).
Ces deux domaines comprennent des motifs qui semblent
indispensables à la transposition:
II.1.1.2.1. Les motifs caractéristiques du domaine
N-Terminal:
1. Un motif hélice-tour-hélice HTH (pour
Helix-Turn-Helix)
localisé dans la région bordée par les acides aminé
87 et 109 de la transposase, il est de type CRO ; Cro est une
protéine qui ne se fixe à l'ADN que sous une forme
dimérique dont chaque monomère est constitué de 03
hélices á et porte 66 acides aminés.
2. Huit motifs hélicesá (á1 à
á8) dont l'hélice á1 est responsable de l'accumulation de
la transposase sur l'ITR3' et l'hélice á2 participe à la
liaison de la transposase à l'ADN mais en s'associant avec le HTH.
3. Un feuillet â démontré qu'elle joue un
rôle dans la dimérisation des transposases (In Halaimia Toumi,
2006).
4. Des signaux d'adressage nucléaire appelé NLS
(Nuclear Localisation
Signal) permettant l'internalisation de la transposase dans le
noyau après sa synthèse dans le cytoplasme cellulaire. En effet
la localisation des NLS des TLEs a été identifiée
expérimentalement tandis que celle des MLEs reste
hypothétique et est basée sur des homologies de séquence
(In Augé Gouillou, 2003).
5. Un motif WVPHEL relativement conservé et
paraît impliquer dans un phénomène de
trans-dimérisation des molécules de la transposases (In
Augé Gouillou, 2003).
II.1.1.2.2. Les motifs caractéristiques du domaine
C-Terminal:
Outre que la triade catalytique D,D(34)D, ce domaine comporte
un motif YSPDLAPD très conservé dont le dernier D de ce motif
correspond au dernier D de la triade catalytique (figure 6.3) (In Halaimia
Toumi, 2006).
La transposase est une phosphoryletransférase dont
activité catalytique (de la triade D,D(34)D) requiert des cations
divalents qui est le magnésium, les trois résidus acides sont
associés dans une poche catalytique capable de contenir le ou les
cations, la présence du magnésium apporte des charges positives
permettant le recrutement des groupes nucléophiles impliqués
à chaque coupure de brin d'ADN. Le motif DDD est une signature qui
spécifie les éléments mariner tandis que les
TLEs sont spécifiés par une triade DDE (E pour le
glutamate) (In Augé Gouillou, 2003).
WVPHEL YSPDLAPD
â1 á1 á2 á3 HTH
á4 á5 á6
NLS NLS
D D 34 D
Domaine N-terminal Domaine C-terminal
â1: Feuillet Beta
á1 à á8 : Hélices
alpha
NLS : Signal potentiel de Localisation
Nucléaire
Figure 6.3 : Structure générale
de la transposase des MLEs (ici la transposase de
Mos1)
(modèle d'après Halaimia Toumi, 2006)
II.1.2. Une comparaison entre les MLEs et les
TLEs:
Selon les structures nucléiques, ils se diffèrent
dans (voir tableau 2) :
Tableau 2 : Les différences
nucléiques entre les MLEs et les TLEs
|
Taille de
|
MLEs
|
|
|
|
TLEs
|
|
ORF
|
1050
|
|
|
|
1050#177;90
|
|
ITR
|
19-69 Mcmar1
|
à
|
l'exception
|
de
|
24-756
|
|
UTR
|
|
|
|
|
150-1300 en 5' 100-2200en 3'
|
|
Taille totale
|
1300
|
|
|
|
1500-4700
|
Selon leurs structures protéiques, ils se distinguent
dans: (voir tableau 3)
Tableau 3: les différences
protéiques entre les MLEs et les TLEs
|
Domaine D Terminal
|
MLEs
|
TLEs
|
|
HTH
|
Un seul motif
|
Deux motifs
|
|
Les hélices á
|
Huit hélices á
|
Deux hélices dont chacune est associée à un
HTH
|
|
Les motifs conservés
|
WVPHEL
|
WVPHEL
|
Dans le domine C terminal les deux éléments se
diffèrent dans la triade catalytique qui est D.D(34)E chez les
TLEs (Figure 6.4)

1 7 45 68 108 153 244 279 340
á1, á2: Hélices Alpha
HTH : Hélice tour Hélice
NLS : Signal de localisation
nucléaire.
Figure 6.4 : Structure de la transposase des
TLEs (ici la transposase de Sleeping
Beauty) (In Halaimia
Toumi, 2006)
(á1 + HTH) (HTH + á2) NLS D
D34E
Domaine N-terminal Domaine C-terminal
YSPDLAPE
III. Fonctionnalité des MLEs : III.1. Les MLEs
actifs :
Le premier élément reconnu comme un MLE
actif est Mos1 (pour mosaic) qui était isolé
par Jacobson et Hartl en 1986, (Hartl, 2001) à partir du génome
de Drosophila mauritiana, un autre élément appelé
Himar est construit à partir d'un élément
muté isolé de la mouche Haematobia irritans, un
troisième élément Famar1 était
découvert chez le perce-oreille Forficula auricularia. (In
Halaimia Toumi, 2006)
III.1.1. Le mécanisme de transposition des MLEs
:
Tous les MLEs et aussi les TLEs transposent
via un mécanisme de type « coupercoller » homologue à
celui décrit chez quelques IS et Tn, ce
mécanisme est constitué par trois étapes principales :(
figure 6.5)
1. Reconnaissance et liaison de la transposase aux ITRs.
2. L'excision du transposon de son site donneur.
3. Réinsertion du transposon dans le site cible (In
Augé Gouillou, 2003).

1 - Reconnaissance et liaison
Figure 6.5: Schéma du mécanisme
de transposition des MLEs (Modèle d'après
Augé
Gouillou, 2003)
2 - Excision 3 - Reinsertion
aux ITRs
Site
donneur
AT
TA
AT
TA
Transpososome
TA
AT
nn
TA
AT
nn
Intermédiaire de
Transposition
AT
TA
Site
accepteur
AT
TA
AT
TA
III.1.1.1. Liaison aux ITRs :
La liaison aux ITRs est assurée par le domaine
N-Terminal (plus précisément par les HTH, l'hélice
á1 et á2) qui reconnait
spécifiquement chaque ITR, cette liaison aboutit à la formation
d'une structure nucléoprotéique active qui est << le
transpososome >>.
Deux types de dimérisation protéine-protéine
peuvent avoir lieu lors de la formation des complexes ITR/transposase :
1- Interface de dimérisation << Cis
>> : C'est la liaison de deux monomères de transposase
côte à côte sur une seule molécule d'ADN, cette
interface est localisée dans les premiers 20 aa.
2- Interface de dimérisation << trans
>> : la liaison de deux molécules de transposases liées
à deux molécules d'ADN différentes.
Ainsi, la région de la transposase située entre
les aa 116-143 et qui comprend le motif WVPHEL est directement impliquée
dans le passage d'une conformation cis à une conformation
trans et la stabilité de cette dernière.
La transposase se lie aux ITRs au niveau d'une séquence
cible palindromique aboutissant à la formation d'un complexe
appelé cis-SE (Single End Complex 2), trois voies principaux
sont proposées pour la destination de ce complexe :
1' La dissociation d'un monomère de transposase du
complexe SEC-2 conduisant à la formation d'un complexe SEC1. (SEC1 : une
molécule de transposase liée à un ITR, SE : 2
molécules de transposase liées à un ITR.)
1' Un monomère du complexe SE subit une modification
conformationnelle caractérisée par l'acquisition d'une nouvelle
conformation plus stable dite << Trans-SE >> dont les deux
monomères possèdent une interface cisdimérisation libre.
Un monomère du trans dimère peut recevoir un autre ITR
en formant le complexe PEC1 (Paired End Complex 1). Enfin via les interfaces de
dimérisation libres les deux transposases de ce complexe sont
susceptibles de se lier à deux autre transposases pour former le
complexe synaptique final dit PE.
1' Le complexe final PE résulte directement de la
dimérisation de deux complexes cis-SE (Figure 6.6) (In Halaimia
Toumi, 2006).

trans-SE
1 trans- dimère
Figure 6.6: Représentation
schématique des différents complexes
transposase/ITR
SEC: Single End Complex.
PEC: Paired End Complex
(Modèle in Halaimia Toumi,
2006).
Cis-SE 1 cis-dimère
SEC1 ITR
Tnp
Voie 2
Voie 1
Voie 3
PE
1 trans-dimère
2 Cis-dimères
et 2
trans-
dimères
PEC1
L'exemple de Mos1 :
Les tests in vitro utilisant une protéine
recombinante montrent qu'elle nécessite la présence des cations
divalents (Mg+2, Mn+2, ou Ca2+) et un Ph
élevé (autour du 9) et que la liaison Tnp/ITR est hautement
spécifique parce que la transposase Mos1 ne se lie que sur ses
propres ITRs ce qui explique l'absence d'un phénomène de
transposition croisée (la transposition in trans). Les ITRs du
Mos1 sont imparfaits parce que la séquence de l'ITR5'
diffèrent de celle de l'ITR3' en 4 positions sur 28pb (position 1, 16,
18 et 26). Il a été démontré que les positions 1,
18 et 26 sont des contacts stabilisants entre la transposase et l'ITR parce
qu'ils ont un faible impact sur la qualité et la quantité de la
transposition, en revanche une forme mutée d'un ITR dans la position 16
a conduit à une perte de 90% la liaison Tnp/ITR ce qui suggère
qu'elle est un point d'ancrage de la transposase sur l'ITR (In Augé
Gouillou, 2003).
III.1.1.2. L'excision de l'élément :
Les coupures réalisées par les transposases des
MLEs et ceux des TLEs aux bornes des ITRs pour faire
libérer l'élément sont assurées par la poche
catalytique démontrée qu'elle est très importante dans la
catalyse par mutagénèse dirigée, chez de nombreux
transposons bactériens apparentés aux MLEs et
TLEs. En effet, chez les MLEs cette étape se
déroule en deux phases (figure 6.7).
(1)

3'
5'
Figure 6.7 : Les étapes de l'excision
d'un élément de type mariner (D'après
Augé
Gouillou, 2003)
TA
AT
TA
AT
A TA A AT
AT
AT
TA
A TA
(1)
ITR
5'
3'
(2)
Coupure du premier brin : Chez la transposase
Mos1, cette coupure est imprécise et peut être
localisée majoritairement à trois bases à
l'intérieur de l'ITR ou même à 2 ou 1 base dans l'ITR, ou
aussi à la hauteur du << TA », un phénomène
identique a été observé chez Himar.
(2) Coupure du second brin : Dans ce cas la
coupure est extrêmement précise et prend place au niveau du
dinucléotide << TA » et produit une extrémité
3'-OH sortante, la localisation de cette coupure est compatible avec la
cicatrice de l'excision laissée dans le génome de l'hôte,
au site du donneur.
Le comportement de la transposase mariner dans le
processus de l'excision est semblable à celui des protéines
RAG1/RAG2 impliquées dans la recombinaison V(D)J permettant l'assemblage
des gènes pour la production des immunoglobulines. Des structures en
boucles comme celles observées dans ce système ont
été recherchées après excision de
l'élément, cependant la présence des boucles n'a pas pu
être établie ainsi que la structure de l'intermédiaire de
transposition (In Augé Gouillou, 2003 ; in Halaimia Toumi, 2006). En
général plusieurs solutions sont envisageables :
" L'intermédiaire de transposition
présente une forme linéaire avec des extrémités
libres comme le cas du transposon Tn7.
" Il est également linéaire
mais avec des extrémités en « hairpin » comme
dans IS10, IS50, au moment de l'insertion ces structures vont
être résolues par la transposase.
" L'intermédiaire de transposition est circulaire
où les ITRs sont en position TêteBêche et associées
par une jonction de quelques paire de bases (le cercle est résolu par la
transposase avant l'insertion).
Les produits de l'excision des TLEs et des
MLEs se ressemblent, alors que la chronologie des étapes semble
légèrement différente, les TLEs ont
également faits l'objet de travaux pour identifier la structure de leur
intermédiaire de transposition, mais les résultats sont
paradoxaux, par exemple :
L'élément Tc1 a un intermédiaire
de transposition qui est soit linéaire, soit circulaire selon les
auteurs. Enfin la structure de l'intermédiaire de transposition dans la
famille Tc1/mariner reste donc à établir (In Augé
Gouillou, 2003).
III.1.1.3. La réinsertion de l'élément
dans son site cible :
Au cours de cette étape le transposon est
transféré par la transposase dans un site accepteur du
génome hôte. Ce site est caractérisé par la
présence d'un dinucléotide TA dupliqué lors de
l'insertion, les mécanismes moléculaires intervenants pendant
cette étapes sont encore inconnus. (In Halaimia Toumi, 2006).
III.1.2. L'intérêt de l'étude des
MLEs :
Mariner est le parasite génétique le
plus ubiquiste connu chez les eucaryotes, cette propriété
explique l'intérêt fondamental de l'étude des MLEs
ainsi que les perspectives de valorisation biotechnologique et
médicale d'un tel modèle biologique.
III.1.2.1. L'intérêt fondamental :
Les MLEs peuvent jouer un rôle dans la
plasticité des génomes hôtes, en favorisant les
réarrangements chromosomiques par exemple : chez la drosophile les
changements de place des transposons étaient essentiellement
associés à des réarrangements chromosomiques qu'à
de la transposition. Sous cette hypothèse, toutes les copies même
inactives participent majoritairement dans l'évolution des
génomes. Présents dans de nombreux génomes eucaryotes et
souvent avec un nombre élevé de copies en grande partie inactive,
les MLEs pourraient être des acteurs importants de la
plasticité des génomes eucaryotes. La présence des copies
active favorise et renforce leur impact sur les génomes en affectant
directement la régulation de l'expression des gènes comme dans le
cas de Dmmar1. Un exemple du rôle possible des MLEs
dans la recombinaison a d'autre part été illustré dans le
génome humain par : la maladie de Charcot-Marie Tooth qui est due
à un crossing-over inégal sur le chromosome 17p,
un élément mariner a été
identifié à proximité du point de recombinaison et les
auteurs suggèrent que la recombinaison pourrait avoir été
favorisé par une cassure de l'ADN due à la transposase. Il semble
que les éléments mariner aient, ou ont eu, la
capacité de se perpétuer en colonisant de nouveaux
génomes, suite à des transferts horizontaux, alors les MLEs
pourraient jouer un rôle dans l'évolution des
génomes.
III.1.2.2. L'intérêt appliqué :
Grâce à leurs ubiquité et activité,
les éléments mariner offrent les bases
intéressants d'un vecteur de transfert d'ADN. Une transposase
hyperactive dérivée de l'élément Himar1 a
été obtenue par ingénierie moléculaire,
démontrant qu'il est possible d'améliorer les MLEs
naturels peu efficaces en transposition. Ainsi de nombreuses publications
décrivant l'obtention, grâce à mariner des
organismes ou de lignée de cellules transgéniques chez les
protozoaires, les insectes, les poissons et les oiseaux
confirment que mariner est un bon candidat pour
transférer l'ADN exogène chez les eucaryotes (In Augé
Gouillou, 2003 ; in Halaimia Toumi, 2006). Par exemple chez la souris, il est
possible de transfecter des cellules souches avec un élément
mariner modifié (le gène d'intérêt
flanqué par des ITR d'un élément mariner), les
cellules transformées seront par la suite transplantées dans une
femelle, et le gène d'intérêt sera exprimé dans la
génération suivante.

Figure 6.8 : Les différentes
étapes de la transgénèse à l'aide de
mariner chez la souris
(d'après Halaimia Toumi et
al., présentation GBM ).
III.2. Les MLEs potentiellement actifs
:
Plusieurs éléments mariner ont
été découverts mais la majorité d'entre eux sont
inactifs parce qu'ils accumulent des mutations ponctuelles ou des
délétions.
Ainsi il existe 9 transposons potentiellement actifs,
c'est-à-dire que leurs ORFs sont
complets mais leur activité
de transposase n'a pas été démontrée. Ces
éléments sont
présents dans le tableau ci-dessous : (In Halaimia Toumi,
2006)
Tableau 4 : Quelques éléments
mariner potentiellement actifs (In Halaimia Toumi,
2006).
|
Elément
|
Espèce hôte
|
|
Ccmar1
|
Ceratitis capitata
|
|
Crmar1
|
Ceratitis rosa
|
|
Momar1
|
Metaseuilis occidentalis
|
|
Mcmar1
|
Meloidogyne chitwoodi
|
|
Tvmar1
|
Trichomonas vaginalis
|
|
Mdmar1
|
Musca domestica
|
|
Bgmar1
|
Blatella germanica
|
|
Mbomar
|
Messor bouvieri
|
III.3. La caractérisation des transposons de
type mariner :
La caractérisation d'un transposon se fait en suivant 4
étapes principales : III.3.1. Recherche des
éléments mariner à partir de l'ADN
génomique par PCR :
III.3.1.1. Extraction de l'ADN génomique:
L'ADN est extrait à partir des organismes entiers et de tissus
congelés à -80°C ou dans l'azote liquide.
L'échantillon sera broyé dans du tampon de préservation.
Les protéines et les débris cellulaires peuvent être
éliminés par un traitement au phénol/chloroforme. Les
acides nucléiques seront ensuite précipités à
-80°C par de l'acétate de sodium et de l'éthanol absolu. Un
traitement à la RNAse A et une ultracentrifugation sur gradient de
Chlorure de Césium permettent d'achever la purification. Après
ultracentrifugation, l'ADN peut être récupérer
sous rayonnement ultraviolet. Le Bromure d'Ethidium est
ensuite éliminé par des lavages successifs à l'isopropanol
saturé par une solution de NaCl. L'ADN se précipite par addition
de l'eau ultra pure et de l'isopropanol pendant 2 heures à -20°C.
Après centrifugation, le culot sera repris dans l'acétate de
sodium et l'ADN sera reprécipité en ajoutant de l'éthanol
absolu pendant 2 heures à -80°C. Après centrifugation 15
minutes à 15000g, le culot d'ADN doit être lavé avec de
l'éthanol 70°C (In Halaimia Toumi, 2006).
III.3.1.1. PCR: Après extraction de
l'ADN génomique d'un organisme, les transposons mariner seront
recherchés par PCR (Polymerase Chain Reaction ou l'amplification en
chaîne par polymérase) (In Halaimia Toumi, 2006). Inventée
par Kary Mullis (prix Nobel de Chimie 1993), cette technique est
utilisée pour amplifier une séquence d'ADN cible en utilisant une
paire d'amorces d'oligonucléotides, chacune complémentaire
à une extrémité de la séquence cible. Celles-ci
subissent une élongation par une ADN polymérase thermostable (la
Taq polymérase), dans un cycle de réaction à
trois étapes : dénaturation, hybridation de l'amorce et enfin
polymérisation (Figure7) (In Turner et al., 2002). Les amorces
utilisées pour la recherche de ce type du transposon sont les amorces de
Robertson décrites en 1993 et qui correspondent aux motifs
conservés des transposases mariner (In Halaimia Toumi,
2006).
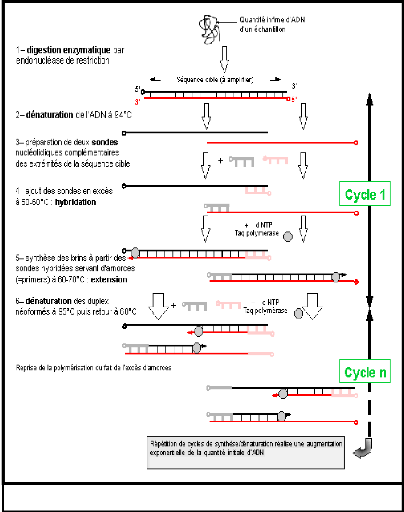
Figure 7 : Les différentes étapes
de la technique PCR (D'après
www.inrp.fr)
III.3.2. Hybridation des éléments par la
technique de Southern Blot : Elle est utilisée
pour
confirmer s'il existe vraiment des transposons dans le génome
d'intérêt ou non.
Cette technique fut inventée par
Southern (1975-1979b) est basée sur le transfert des
acides nucléiques sur une membrane pour le
repérage des séquences complémentaires à une
séquence spécifique d'ADN ou d'ARN appelée sonde, la
membrane d'hybridation est placée en sandwich entre le gel et un papier
absorbant qui assure l'entraînement de solution-tampon à travers
le gel (Figure 8) (In Primose et al., 2004). Les sondes
utilisées pour le repérage des transposons mariner sont
des transposons mariner apparentés car ils présentent un
taux d'homologie très importants (In Halaimia Toumi, 2006).

Autoradiogramme
Figure 8 : Schéma démontrant le
principe de Southern Blot (D'après Griffiths et al. 2004)
Marqueur de taille
Électrophorèse
ADN ou ARN
Migration
La solution passe à travers le filtre
Éponge
Gel
Solution salée Filtre de nitrocellulose
Filtre
Hybridation avec une sonde
Lavage pour enlever les liaisons non
spécifiques
Le transfert de l'ADN
au filtre
Hybridation des
sondes et
séquences
complémentaires
III.3.3. Séquençage par la
méthode de Sanger : Les fragments obtenus par les techniques
précédentes seront envoyés au séquençage,
une des méthodes de séquençage la plus utilisée est
celle de Sanger. Cette méthode fait intervenir une étape de
dénaturation des deux brins, une hybridation avec une amorce
marquée radioactivement et une élongation par une ADN
polymérase en présence de
didésoxyribonucléosides
triphosphate (ddNTP). Ces molécules peuvent être
normalement incorporées dans un brin d'ADN en élongation
grâce à leur extrémité 5' triphosphate mais ne
peuvent pas former de liaison phosphodiester avec le nucléoside
triphosphate suivant. Lorsqu'une faible quantité d'un ddNTP est
présente (ddATP par exemple) en plus des quatre dNTP nécessaires
pour la polymérisation (dATP, dCTP, dGTP et dTTP), plusieurs fragments
marqués sont générés. Ils correspondent tous
à des fragments portant un résidu ddATP à
l'extrémité 3'. En répétant l'expérience
pour les autres ddNTP et en séparant les différents fragments
d'ADN de chaque réaction de polymérisation par
électrophorèse sur gel de polyacrylamide (séparation selon
la taille des séquences), la séquence du fragment d'ADN de
départ peut être déduite. Aujourd'hui, des
séquenceurs automatiques de l'ADN existent sur le marché. Ils
sont capables d'effectuer l'électrophorèse et de
déterminer directement la séquence nucléotidique par une
lecture d'un ou de plusieurs fluorochromes (Figure 9) (In Primose et
al., 2004).

75
ADN polymérase + 4dNTP
Gel de polyacrylamide
Brin d'ADN
ADN
Amorce marquée
Amorce marquée ADN polymérase
+4dNTP+ ddATP
La séquence du gel
La séquence originale
Figure 9: Le séquençage selon
la méthode de Sanger. (a) : l'addition d'une amorce marquée et
ddNTP et poursuit de l'élongation. (b) : les fragments résultants
des différentes réactions sont séparés par
éléctrophorèse. (c) : le gel de séquençage
de Sanger (d'après Griffiths et al., 2004)
III.3.4. Analyse des séquences in
siico : Les séquences obtenues seront analysées
in silico par des logiciels de traitement de séquences
spécifiques. Ces derniers comparent les nouvelles séquences par
rapport aux autres séquences existantes dans la base des données,
ainsi ils permettent la recherche des motifs caractéristiques. Par
exemple, l'analyse d'un élément mariner issu d'un
organisme des sources hydrothermales océaniques (l'élément
Alvcmar du ver Alvinella caudata) a montré la
présence des motifs conservés WVPC(R/X)NL et YSPDL(A/T)(P/H) au
lieu des motifs WVPHEL et YSPDLAP définis par Robertson 1993. Ainsi ces
analyses ont permis la mise en évidence de deux codons start,
cette remarque permet d'émettre l'hypothèse que ce transposon
peut coder pour deux isoformes différentes de la transposase (Figure
10). Cette transposase ressemble aux autres transposases des
éléments mariner dans la triade catalytique D.D(34)D, le
premier D se trouve au niveau d'un motif conservé DETWV, le second est
localisé dans le motif HDN et le dernier se trouve juste après le
motif YSPDL(A/T)(P/H). De plus, elle présente un feuillet â dans
leur domaine N-terminal, et des hélices « HTH », enfin un
signal potentiel de localisation nucléaire NLS a été
observé dans la région C-terminal (Figure11) (In Halaimia Toumi,
2006).
ITR5' 1298pb ITR3'
UTR5' UTR3'
36pb 37 pb

TA
ORFII = 1050 (350 aa)
ATGI ATGI
ORFI = 1140 (380 aa)
Figure 10 : Structure nucléique de
l'élément Alvcmar (D'après Halaimia
Toumi,
2006)
CGGTTAGGACTGGTTCTGAC AWG TTC ACA AGC AGC GCC GCT CCG CTC CCT CGC
TCC CTC 130 X F T S S A A P L P R S L 13
CTC AGT CTT AAG GGA GCG AGC AGT GGA CGY GGT TGG CAG GAG TCG GCA
ACA ATG 184

L S L K G A S S G R G W Q E S A T M 31
GAC AAG ATC GAG TAC CGT GCA GTG ATC AAG TTC TTG ACA AAA GAG GGG
AAG AAC 238
|
D K I E Y R A V I K F L T K
|
E G K N 49
|
Hélice á
GCG AAG GAG ATC CAC GAC AGG CTG GTT GCG GTG TAC AAC GAC ACT GCC
CCT TCG 292
|
A K E I H D R L
|
V A V Y N D T A P S 67
|
Hélice á
TAT GCC ACA GTC ACC CGC TGG CAC AAG GAA TTT CGT CAT GGC CGT GAG
TCC CTT 346
|
Y A T V
|
T R W H K E
|
F R H G R E S L 85
|
Hélice á
GAA GAC GAC CCC CGT GTG GGA CGC CCC TCC GAG GCG ACC TCC GAA GAC
ATT GTT 400
|
E D D P R V G R P S E A T S
|
E D I V
|
103
|
GAT CGT GTG GAG GCA ATG ATC ATG GAG AAT CGG CGA GTG AAG GTG GAG
GAA ATT 454
|
D R V E A M I M E
|
N
|
R R V K V E E I
|
121
|
|
Hélice á
|
|
|
|
TCG TTG GAG ATT GGA ATT TCT CAT GGA AGC GTT TGC ACC ATT ATT CAT
CAT CAC 508
|
S L E I G I S H G S V C T I I
|
H H H 139
|
Hélice tour hélice
CTG GGC ATG AGC AAA GTT TCT GCT CGT TGG GTG CCC YGA AAT CTT TCT
CTG CAT 562
L G M S K V S A R W V P X N L S L H 157
GAT CGC CTT CAG CGC CGC ACA AGT TCG GAG GAG CTG CTG ACT CTG TAC
AAC GCA 616
|
D R L Q R R
|
T S S
|
E E L L T L Y N
|
A 175
|
Hélice á Hélice á
GAC CCA GCG GGA TTC GAG TCG AGG GTC GTG ACA GGT GAT GAA ACG TGG
GTT CAC 670
D P A G F E S R V V T G D E T W V H 193
CAC TGG GAC CCA GAG ACG AAG CTC GAG AGC ATG GCC TGG AAA CAC AAG
GGA TCC 724
H W D P E T K L E S M A W K H K G S 211
CCG ACA CCG CTC AAG TTT CGG ACC CAA CCA TCG GCT GGC AAG ATC ATG
GCC ACC 778
P T P L K F R T Q P S A G K I M A T 229
ATC TTC TGG GAC GCC GAG GGA GTG CTG CTG GTG GAC TTC CTG CCG CGT
GGC TCT 832
I F W D A E G V L L V D F L P R G S 247
ACG ATC ACG GGG GAG TAC TAC GCC GGM GTA CTC GGT CGC TTG AGG GAC
TCC ATC 886
T I T G E Y Y A G V L G R L R D S I 265
CGT CAG AAG AGG CGG GGC AAG TTG ACC CGT GGT GTC CTC CTC CTC CAC
GAC AAC 940
|
R Q K R R G K
|
L T R G V L L L H D N 283
|
NLS
GCT CCG GTC CAC AAG GCC CGC CGT GCC CAG GCT GCT CTG AGG GAC TGC
GGC TTC 994
A P V H K A R R A Q A A L R D C G F 301
GAG CAG CTC AAT CAC CCA CCC TAC AGT CCG GAC CTG GCC CCC AGT GAC
TAC TTT 1048
E Q L N H P P Y S P D L A P S D
Y F 319
CTG TTC CGC CAG CTC AAG TCC TCG TTG CGG GGG CGG AGG TTC GAC GAC
GAT GAT 1102 L F R Q L K S S L R G R R F D D D D 337
GAG GTC AAG GAG GCT GTG ATG ATG TGG TTG GAG GAG CAG TCG GAA TCC
TTC TGG 1156 E V K E A V M M W L E E Q S E S F W 355
CTG GCA GGA ATC CAG AGC CTT CGC GAC AAG TGG TTC AAG TGT ATT CAA
GTC AAG 1210 L A G I Q S L R D K W F K C I Q V K 373
GGT AAT TAC ATT GAA AAA
TGATGTGGTTATCATTTTCATTCCTCCGAAATAAAATACATACCGAA 1275 G N Y I E
K * 379 TTGCAGAACTTTCTGACCGCCCCTCGTA
III.2.1.4. Analyse fonctionnelle d'un transposon de type
mariner :
L'étude fonctionnelle des différentes transposases
mariner peut être réalisée selon deux
stratégies : les tests de transposition in vitro, et les tests
de transposition in vivo.
III.2.1.4.1. Tests de transposition in vitro :
Ces tests permettent l'analyse et l'interprétation des
différentes étapes de la transposition indépendamment
chacune de l'autre (voir figure 6.5 page 63). Pour réaliser cette
approche il est obligatoire de produire la transposase, cette étape
nécessite le clonage d'un transposon dans une bactérie. Il doit
être véhiculé par un vecteur d'expression en se liant en
aval d'un promoteur fort. Le vecteur et le gène codant la transposase
seront clivés par la même enzyme de restriction pour assurer la
complémentarité des bases. Le plasmide pMAL est largement
utilisé, Il est caractérisé par la présence d'un
gène de la résistance à l'ampicilline (Permettant une
sélection) et permet la production de la transposase sous une forme
fusionnée avec la MBP "Maltose Binding Protein" (Figure 12).
L'expérience permettant la production des transposases est citée
ci-dessous:
1' Préparation du gène codant la transposase en
le fusionnant avec le gène de la MBP porté par le plasmide pMAL
de sorte qu'il soit insérer en aval d'un promoteur inductible
(Figure13).
v' Transformation d'une bactérie par le plasmide pMAL
recombinant.
v' Sélection des bactéries transformées
selon leur résistance à l'antibiotique.
v' Culture des bactéries dans un milieu nutritif et
l'induction de l'expression de la transposase par l'IPTG (l'IsoPropyl-B-D
ThioGalactopyranoside).
v' Récupération des bactéries par
centrifugation.
v' Extraction des protéines recombinantes par la lyse
des parois bactériennes en utilisant les lysosymes, puis centrifugation
pour l'élimination des débris cellulaires et enfin le dosage des
protéines obtenues.
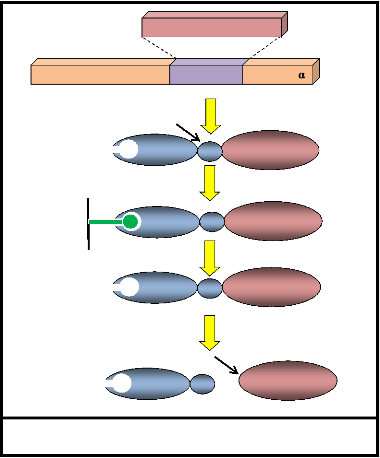
Figure 12 : Expression de la transposase
fusionnée à la MBP par le plasmide
pMAL.
Amylose
Site spécifique de clivage
mal E
MBP
MBP
MBP
MBP
Gène de la transposase
MCS
Chargement et lavage
Elution avec le maltose
Clonage et expression
Clivage avec une
protéase spécifique
Transposase
Transposase
Transposase
Transposase
v' La transposase fusionnée sera purifiée par la
chromatographie d'affinité : la fixation des protéines s'effectue
sur une résine amylose, l'élution sera réalisée par
le maltose (Figure 12).
v' La technique SDS-PAGE donne une estimation sur la taille
des protéines fusionnées, par exemple la transposase
fusionnée d'Alvcmar mesure 80KDa sachant que la MBP seule
mesure 40KDa, alors la transposase d'Alvcmar peut mesurer 40KDa
(Figure 14) (In Halaimia Toumi, 2006).

Plasmide pMAL
Gène de la MBP
Résistance à
l'ampicilline
Le transposon mariner
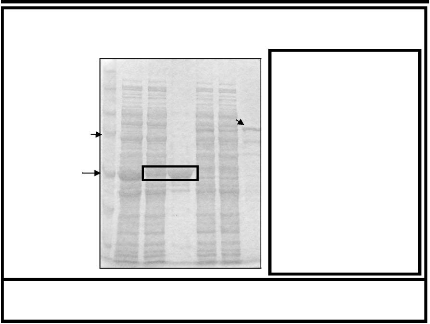
Figure 14: Analyse de la production des
protéines sur gel SDS -PAGE (d'après
Halaimia Toumi, 2006)
75 kDa
40 kDa
M MBP Transposase A (A pour Alvcmar)
1 2 3 4 5 6
-Extrait protéique brut pMAL vide (puit1). pMAL-A (4)
(A : pour Alvcmar).
-La flèche noire indique La transposase A
-M : marqueur de taille.
- Surnageants récupérés après la
purification de la MBP (2), Transposase A (5).
- Protéines purifiées : MBP (3), Transposase A
(6)
Figure 13 : Le clonage du gène de la
transposase mariner ligué avec le plasmide
pMAL dans une
bactérie (
perso.univ-lyon2.fr)
La production de diverses transposases mariner a
révélé l'existence de plusieurs versions tronquées,
en effet, Erwan et al. (2005) ont montré pendant la production
d'une transposase IS911 la présence de plusieurs formes
tronquées. Une de ces formes s'est avérée un inhibiteur
pour la transposition, alors le phénomène de la transposition est
vraisemblablement limité si des transposases tronquées sont
disponibles. (In Halaimia Toumi, 2006).
III.2.1.4.1.1. Analyse de la liaison des transposase de
type mariner aux ITRs :
Ce test sera assuré par une technique appelée
l'analyse de retard sur gel (In Halaimia Toumi, 2006), cette
technique mis en évidence la liaison d'une protéine à une
séquence d'acide nucléique marqué, la migration des
complexes ADNmarqué/protéine, sur un gel de polyacrylamide non
dénaturant, sera retardée par rapport à celle de l'ADN
seul (In turner et al., 2002) (Figure 15). Cette technique est
basée sur l'incubation de la transposase mariner
indépendamment avec les ITR3' et 5' marqué avec du dATP
(ãP 32), soit par le marquage de l'ADN double brin en
effectuant une phosphorylation avec la T4 nucléotide kinase, ou par le
marquage des ITRs monocaténaires puis leur hybridation pour l'obtention
d'une structure bicaténaire. Les complexes ITR3'/Tnp, ITR5'/Tnp,
seront formés dans un milieu contenant du tampon de colonne,
glycérol (pour le maintien de la stabilité), de l'ADN du sperme
de Saumon (compétiteur non spécifique), tous ça en
présence ou absence d'EDTA (chélateur de cations) et de
Mg2+. La séparation sera réalisée sur un gel
composé de deux gels de polyacrylamide superposés : un gel de
séparation au dessus d'un gel de résolution, la migration doit
s'effectuer à des températures adaptées aux conditions de
vie des organismes hôtes (In Halaimia Toumi, 2006).

ITR seul
(Sonde libre)
Figure 15 : Principe de la technique de retard
sur gel (d'après Halaimia Toumi,
2006)
Différents
Complexes
Produit de coupure

Figure 16 : Analyse de la liaison de la
transposase
Mos1 (d'après Augé Gouillou et
al., 2005)
Produits de clivage
En effet, il a été montré par
Augé-Gouillou et al, 2001 que la transposase de Mos1
reconnaît et se fixe spécifiquement à ses ITRs en
présence de cations Mg2+. Ces auteurs ont observé
aussi que l'ajout d'EDTA, qui chélate les ions Mg2+ dans le
milieu réactionnel au cours des expériences de retard sur gel
diminue légèrement le taux de liaison Tnp/ITR, ce qui
suggère que la transposase Mos1 utilise les ions
Mg2+ dans la catalyse. De plus, ils ont pu visualiser les
différents complexes formés par la transposase
Mos1 grâce à la technique du retard sur gel (Figure
16).
III.2.1.4.1.2. Test de spécificité de
liaison des transposases de type transposase mariner aux ITRs
:
Le principe de ce test est la vérification de la
spécificité de la liaison d'une protéine à son
ligand, dans ce cas, de la transposase mariner aux ITR3' et 5'. Cette
vérification peut être réalisée par l'addition d'ADN
compétiteur dans le milieu réactionnel (Figure 17). Sur un gel de
polyacrylamide homologue à celui de l'analyse de retard sur gel, cette
spécificité sera analysée c'est-à-dire s'il existe
une spécificité, le même profil de migration sera obtenu en
présence ou absence d'un ADN compétiteur, pour la confirmation,
la transposase doit être incubée avec un ITR radioactif et un
autre non radioactif mis en excés ; alors en cas de
spécificité, la fixation de la transposase avec l'ITR froid sera
plus importante qu'avec l'ITR radioactif. (Figure 17). Un exemple d'une telle
spécificité est fournit par l'élément
Bytmar issu du crabe Bythograea thermydron qui provient des
sources hydrothermales océaniques, le test de spécificité
de sa transposase a montré une spécificité satisfaisante
à ses ITR car le profil de migration n'a pas été
changé au cours de l'addition de l'ADN compétiteur non
spécifique, aussi la réaction a été
légèrement déplacée en présence des ITR non
radioactif (Figure 18) (In Halaimia Toumi, 2006).

Cas 1 Cas 2 Cas 3
Figure 17 : Schéma explicatif du
principe de l'analyse de spécificité : Dans le premier cas la
transposase est incubée avec l'ITR radioactif, dans le deuxième
cas la transposase est incubée avec l'ITR radioactif et un ADN
compétiteur mis en excès et dans le troisième cas la
transposase est incubée avec de l'ITR radioactif et de l'ITR non
radioactif mis en excès. (D'après Halaimia Toumi, 2006).

Avec l'ITR5' Avec l'ITR3'
Figure 18 : Analyse de la
spécificité de la fixation de la transposase Bytmar avec
leurs ITRs (Conditions expérimentales 30°C-2h)
3 : Tnp + ITR marqué + ITR non marqué mis en
excès (D'après Halaimia Toumi, 2006).
2 : Tnp + ITR marqué + ADN compétiteur mis en
excès
1 : Tnp + ITR marqué
III.2.1.4.1.3. Test de coupure des transposases de type
mariner :
Pour l'analyse des phénomènes de coupure, un
test de coupure peut être réalisé. La transposase
mariner sera mise en contact avec 3 plasmides contenant un
pseudotransposon indépendamment, le premier porte à ses
extrémités des ITR3' et 5' (appelé construction 5T3), le
deuxième porte à chaque extrémité un ITR5'
(construction 5T5) et le troisième porte à chaque
extrémité un ITR3' (construction 3T3). Chaque construction sera
additionnée à un milieu réactionnel permettant sa coupure
par la transposase. L'ADN coupé sera ensuite analysé dans un gel
d'agarose. 3 formes plasmidiques seront attendues (Figure 19) :
· La forme surenroulée << SC pur Super Coiled
>> : elle migre plus vite et elle est normalement la forme la plus
présente au début de l'expérience.
· La forme Open Circle << OC >> : elle est
obtenue après une coupure simple brin.
· La forme linéaire << L >> : cette
forme révèle une coupure double brin.

Plasmide linéaire contenant le pseudo
transposon
Plasmide surenroulé
(Super coiled)
pBC+ pseudotrans psonTet
ITR
Plasmide circulaire ouvert à un seul
brin
ouvert (Open circle)
Figure 19 : Schéma explicatif du test de
coupure
(D'après Halaimia Toumi, 2006)
Tnp
Plasmide linéaire sans pseudotrans
poson
Pseudo transposon
M T1 T2 t0 t1 t2 t4 t6
OC (Open Circle) Lin (Linear)
SC (Super Coiled)
Figure 20 : Analyse en test de coupure de la
transposase Alvcmar avec la construction 5T5. M : marqueur de Taille,
T1 : témoin de linéarisation, T2 : témoin de test de
coupure, t0h à t6h : les incubations faites en cinétique de temps
0h à 6h (D'après Halamia Toumi, 2006)
Dans le cas de la transposase d'Alvcmar, ce test a
montré une spécificité assez satisfaisante. Ceci a
été confirmé par la dominance de la forme linéaire
avec la construction 5T5, en plus les trois formes plasmidiques ont
été observées avec une
hétérogénéité des pourcentages avec les deux
autres constructions (Figure 20) (In Halaimia Toumi, 2006).

1
Figure 21 : Test de transposition in vivo
: Un plasmide permettant l'expression de la transposase (pMAL ou pBAD Tnp)
et un plasmide portant un mini-transposon constitué du gène de
résistance à la tétracycline (rectangle mauve), sans son
promoteur (pBC Tet) sont cotransformées dans une bactérie
(rectangle bleu) (1). La transposase (rond noir) se fixe sur les ITR
(flèches roses) du mini-transposon (2) et permet l'excision du
minitransposon (3) qui se réintegrera soit dans un plasmide, soit dans
l'ADN génomique (4) (D'après Halaimia Toumi, 2006).
3
Chl r
Tet r
Chl r
Tet r
Tet r
Tnp
Tnp
2
4
Chl r
Tet r
Tet r
Tet r
ou
Tnp
III.2.1.4.2. Les tests de transposition in vivo :
Ces tests ont pour objectif l'obtention de réponses
directes concernant l'activité ou non d'une transposase de type
mariner. Ces tests sont réalisées à l'aide des
bactéries co-transfomées par deux plasmides ; un plasmide
contenant un pseudotransposon et un autre portant le gène de la
transposase. Si la transposase est active, elle peut exciser et
réinsérer le pseudotransposon. En cas où, la
réinsertion est effectuée en aval d'un promoteur, des
événements de transposition peuvent être détectables
par un système de sélection spécifique (In Halaimia Toumi,
2006). (Figure 21).

Figure 22 : Les différents
étapes permettant la construction de vecteur porteur de
gène
da la transposase (D'après Halaimia Toumi, 2006).
ITR5'
Etape1 : Amplification du gène de la transposase
cloné dans du pGEMT par PCR
en utilisant les amorces 1 et 2. et représentent les
séquences des sites
de restriction des enzymes E1 et E2 respectivement
Etape 4 : Clonage dans un vecteur d'expression
préalablement digéré à l'aide des
enzymes E1 et
E2
E1 E2
Etape 2 : Clonage dans pGEMT pour vérification au
séquençage
Amorce 1 Amorce 2
pGEMT
Etape3 : Digestion avec les enzymes de restriction E1 et E2
ATG Gène transposase TCA
Vecteur d'expression pGEMT
pGEMT
pGEMT
ITR3'
Selon la figure, les bactéries résistantes
à la tétracycline résultent de la transposition du
gène de la tétracycline en aval d'un promoteur, pour effectuer ce
test, il faut passer par les étapes suivantes :
III.2.1.4.2.1. Construction des vecteurs permettant
l'expression de la transposase mariner : (Figure 22)
Deux types de vecteurs sont utilisés :
Un plasmide pMAL c2 : (déjà
décrit dans la partie ) Il permet l'expression de la transposase en
fusion avec la MBP via un promoteur lacI inductible à l'IPTG,
la transposase sera produite sous une forme fusionnée ce qui facilite sa
purification par une réaction d'affinité, ce vecteur porte un
gène de résistance à l'ampicilline
Un plasmide pBADR : Il permet l'expression de la
Tnp via un promoteur inductible à l'arabinose et possède aussi un
gène de résistance à l'ampicilline.
Les deux vecteurs seront digérés par des
endonucléases de restriction, et le gène de la transposase sera
amplifié par PCR, des nucléotides seront additionnés aux
amorces de polymérisation afin de reconstituer des sites d'enzymes de
restriction aux extrémités 5' et 3', ces sites correspondent aux
sites de restrictions connus par les enzymes utilisées pour la digestion
des deux vecteurs. Enfin le gène de la transpsase sera
inséré dans les vecteurs d'expression par ligation en utilisant
une DNA ligase. (In Halaimia Toumi, 2006).
III.2.1.4.2.2. La construction de plasmide pBC KS(+)
contenant le pseudo transposon :
Le pseudo-transposon est le gène de la
résistance à la tétracycline (TetR) sans son
promoteur, flanqué par un ITR en position 5' et 3'. Les ITRs double brin
peuvent être produits par hybridation d'un ITR simple brin up et
un ITR simple brin down, et c'est en ajoutant des
oligonucléotides qui constituerons après hybridation des sites de
restriction, ces derniers permettent le clonage des ITRs dans le plasmide pBC
KS(+) (Figure 23), trois constructions différentes peuvent être
constituées dont chacune contient une combinaison des ITRs
différents (Figure 24), les ITRs à insérer seront
digérées par les mêmes enzymes de restrictions
utilisées pour la digestion du vecteur (In Halaimia Toumi, 2006).

Figure 23 : Les différentes
étapes de construction du vecteur pBC KC (+) porteur
du
pseudotransposon. (D'après Halaimia Toumi, 2006)
pBC
Chl r
Tet r
Chl r
Chl r
Tet r
Double digestion par
HindIII et Xba I
Double digestion par
KpnI et XhoI
Double digestion par
NotI et SacI
Chl r
Tet r
Tet r
Chl r
Chl r
Tet r
Chl r
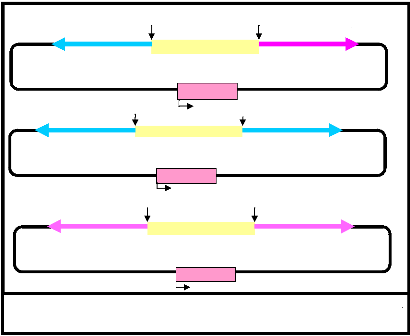
Figure 24 : Les différentes
constructions de pBC porteur de pseudo-transposon
réalisées
(D'après Halaimia Toumi, 2006)
ITR 3' en position 5'
ITR 5'
ITR 5' ITR 3'
Codon stop Codon ATG
Codon stop
Codon stop
pBC 5T5
Tet.R
pBC 3T3
Chl r
Tet.R
Tet.R
Chl r
Chl r
Codon ATG
Codon ATG
ITR 5'en position 3'
ITR 3'
III.2.1.4.2.3. Réalisation des tests de
transposition in vivo :
Les bactéries co-transformées par les deux
plasmides construits précédemment seront mises en culture, puis
sélectionnées sur des boites LB-tétracycline. Dans les
clones contenant le plasmide pMAL, la transcription de la trasposase sera
induite par l'IPTG, tandis que celle des clones contenant le plasmide pBAD sera
induite par l'arabinose. Le taux de transposition est calculé par le
rapport :
Nombre de bactéries sur les boites
LB-tétracycline
Nombre de bactéries sur les boites LB
Les clones qui présenteront une résistance
à la tétracycline seront analysés par extraction de leurs
plasmides et ses linéarisations à l'aide d'une enzyme de
restriction pour la vérification de l'existence d'un tel
évènement de transposition. La résistance à la
tétracycline peut être expliquée par le
fait que la transposase a effectuée l'intégration du
pseudo-transposon en aval d'un promoteur qui doit être du
côté du codon ATG du gène de la tétracycline (In
Halaimia Toumi, 2006).
Augé Gouillou et al., (2001) ont mis en
évidence une activité de transposition in vivo avec la
transposase Mos1, le taux de cette transposition était variable
avec les constructions utilisées dont la construction 3T3 a
manifesté le meilleur taux (3.2×10-5). Ils ont
également montré que la transposition était
inter-plasmidique car la transposition dans le génome bactérien
n'a pas été observée (Figure 25) (Augé Gouillou et
al., 2001).
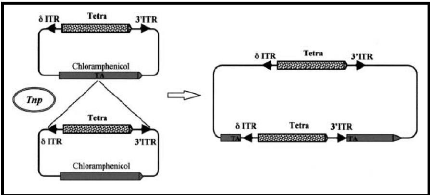
TA
TA
AT AT
Figure 25 : Une représentation
schématique de la transposition inter-plasmidique obtenue avec la
transposase Mos1 au cours des tests de transposition in vivo,
la transposase assure le transfert du pseudo-transposon en l'insérant
dans son site cible qui se duplique au cours de l'insertion et qui est
porté par le gène du chloramphénicol. (d'après
Augé Gouillou et al., 2001).
Conclusion et perspectives
Le rôle des éléments transposables est de
deux aspects distincts. Un premier aspect évolutif parce qu'ils
contribuent d'une façon très significative dans la
plasticité et l'évolution des génomes. Ils participent
également à la création des nouveaux gènes donc
à la création de nouvelles fonctions. Le deuxième aspect
est orienté vers les domaines d'application de ces
éléments dans les biotechnologies. Il s'est avéré
que les propriétés des différents éléments
leur confèrent l'aptitude d'être des bons candidats en
vectorologie.
Une des familles des transposons s'avère de plus en
plus intéressante comme des outils très convenables aux
applications en biotechnologie et en thérapie génique, elle est
la famille mariner. Sa découverte permet d'envisager la mise au
point d'un système de transgénèse ubiquiste facile
à utiliser (Renault et al., 1999), En effet, la mise en
évidence des transposases résistantes à des
températures élevées issues des organismes des sources
hydrothermales océaniques constituerait un plus pour les manipulations
in vivo et in vitro. De toute façon, les transposons
mariner sont des transposons dont les différents composants
nucléiques et protéiques ne contiennent pas les solutions
optimales pour la transposition. Cette propriété permet
l'amélioration de ses aptitudes à intégrer de l'ADN
exogène par ingénierie moléculaire, soit par modification
des ITR pour l'augmentation de l'intégration, ou aussi par modification
de la transposase par mutagénèse pour obtenir une activité
catalytique plus efficace. L'utilité des transposons mariner
dans la transgénèse est conférée par le fait qu'ils
assument les étapes les plus importantes permettant le transfert du
gène d'intérêt, ainsi in vitro des transposases
mariner suggèrent qu'elles n'exigent pas des cofacteurs pour
leur catalyse au sein des cellules vivantes (Laraespada, 2003).
La Bibliographic
1. Amara.A (2008) : Les éléments transposables
chez l'homme. Mémoire bibliographique. pp 1-21. Université de
Monastir. Tunisie.
2. Anxolabéhère.D, Nouaud.D, Miller.W.J (2000)
: Eléments transposables et nouveautés génétiques
chez les eucaryotes. Societé française de
génétique. Vol 16, n°11, 1-9.
3. Augé-Gouillou. C, Hamelin. MH, Demattei. MV,
Periquet. M, Bigot. Y (2001). The wild type conformation of the Mos-1
inverted terminal repeats is suboptimal for transposition in bacteria.
Molecular Genetics and Genomics 265: 51-57.
4. Augé Gouillou.C (2003) : Habilitation à diriger
des recherches. Université de tours CNRS.
5. Augé Gouillou.C, Brillet.B, Hamelin.MH, Bigot.Y
(2005): Assembly of the Mos1 synaptic complex. Molecular and
cellular biology 25: 2861-2870.
6. Bénito.M.I, Walbot.V, (1997): Characterization of
the Maize Mutator transposable element, MURA transposase as a
DNA-Binding Protein. Molecular cellular biology. Vol 17 n°9,
5165-5175.
7. Chen.S, Li. X (2007): Molecular characterization of the first
intact Transib transposon from Helicoverpa zea.
Gene. Vol 408, issues 1-2, 51-63.
8. Feschotte (2004): Merlin, a new Superfamily of
DNA transposons identified in diverse animal genomes and related to bacterial
IS1016 insertion sequences. Molecular biology and evolution
21(9): 1769-1778.
9. Feschotte. C, Pritham.E.J (2007): DNA transposons and the
evolution of eukaryotic genomes. Annual review of genetics 41:
331-368.
10. Gilbert.N (non publié): Insertion d'ADN dans le
génome de cellule humaine, instabilité génomique
associée à la rétrotransposition. Commission de
génie moléculaire : 30-35.
11. Griffiths.A.J.F, Wessler S.R, Lewontin R.C, Gelbart.W,
Suzuki.D.T, Miller.J.H (2004): An introduction to genetic analysis,
8th edition, pp 423-445.
12. Halaimia.Toumi.N, Casse.N, Demattei.M.V, Renault.S,
Pradier.E, Bigot .Y, Laulier.M (2004): The GC-Rich transposon Bytmar1
from the deep-sea hydrothermal crab Bythrograea thermydron, may
encode three transposase isoforms from a single ORF. Journal of molecular
evolution 59: 747-760.
13. Halaimia Toumi.N, Pradier.E, Casse.N, Laulier.M :
Intérêt des éléments génétiques
mobiles marins en biotechnologie, outils de transfert de gènes.
Présentation GBM LBGE.
14. Halaimia.Toumi.N (2006): Caractérisation et
analyse fonctionnelle des éléments transposables de type
mariner issus des deux espèces des sources hydrothermales
océaniques (Bythograea thermydron et Alvinella
caudata). Thèse pour l'obtention du diplôme de doctorat. UFR
des sciences et technique. Université du Maine-Le Mans. France. PP
180.
15. Hartl.D.L (2001) Discovery of the transposable element
mariner. Genetics 157 (2): 417-476.
16. Havecker.E.R, Gao.X, Voytas.D.F (2004): The diversity of LTR
retrotransposons. Genome biology: 5: 225.
17. Huchon.D, Nikaido.M, Okada.N (2002): Eléments mobiles
SINE en phylogénie. Medecine/Sciences : 18 : 1276-81.
18. Kalendar.R, Tanskman.J, Chang.W, Antonius.K, Sela.H,
Pelg.O, Schulman.A.H : (2008) : Cassandra retrotransposons carry
independently transcribede 5S RNA. National academy of science of the
USA. Vol 105, n° 15, 5833-5838.
19. Kapitnov.V.V*, Jurka.J (2005): RAG1 core and V(D)J
recombination signal sequences were derived from transib transposons.
PLos Biology. Vol 5, issue 6, e 181, 1-14.
20. Kapitnov.V.V*, Jurka.J (2006): New kids on the block:
self synthesizing DNA transposons. Comparative and evolution genomics and
proteomics. Part 5, 167- 170.
21. Klug W.S, Cummings.M.R, Spencer.C.A (2006):
Génétique, 8ème edition. pp: 175-190.
22. Koslovski Sassi.A, Herédia.F, Loreto.E.L.da.S,
Valente.V.L.da.S, Rohde.C (2005). Transposable elements P and
gypsy in natural populations of Drosophila willistoni.
Genetics and molecular biology. 28 (4), 734-739.
23. Laraespada.D.A (2003): Generating and manipulating
transgenic animals using transposable elements. Reproductive biology and
endocrinology 7:1:80.
24. Léonard Widemann-Goiran.B (2005): Etude de la
P70S6 kinase chez Arabidopsis thaliana. Thèse pour l'obtention
du diplôme Doctorat en Bioligie Cellulaire et Moléculaire.
Institut national AGRONOMIQUE. PARIS. GRIGNON. France.
25. Lodish, Berk, Kaiser, Krieger, Scott, Betsher, Ploegh,
Matsudaira (2007): Molecular cell biology. 6th edition. pp 1296.
26. Luchetta.P, Marel.M-C, Hiquet.D, Vervoot.M, (2005):
Evolution moléculaire preface d'Axel KAHN. Edition DUNOD. pp 82-100.
27. Malik.H.S, Eikbush.T.H (1999): Modular evolution of the
integrase domain in the Ty3/gypsy class of LTR retrotransposons. Journal of
Virology, vol: 37, n°6, 5186- 5190.
28. Merlin.C, Toussaint.A (1999): Les éléments
transposables bactériens.
SociétéFrançaise de
génétique, vol 15, n°89, 1-13.
29. Mhiri.C, Grandbastien.M.A (2004) : Eléments
transposables et analyse de biodiversité végétale. La
génomique en biologie végétale. Edition INRA. 377-
401.
30. Pelsy.F, Merdinoglu.D (2002): Complete sequence of
Tw1, a family of Ty1-copia retrotransposons of Vitis
vinifera L., reconstituted by chromosome walking. Theor App Genet
102: 614-621.
31. Primose.S, Twyman.R, Old.R (2004): Principes de génie
génétique. Edition De boeck. Pages 400.
32. Pritham.E.J, Putliwala.T, Feschotte.C (2006):
Mavericks a noval class of giant transposeble element widespread in
eukaryotic and related to DNA viruses. Gene: 390: 3-17, pp 1-15.
33. Renault.S, Bonnin-Rouleux.F, Periquet.G (1997):
Eléments transposables et évolution des génomes.
Virologie, Vol 1, n°12, pp 133-144.
34. Rivière Borie.N (2000) : Expression du
rétrotransposons 412 dans les populations naturelles de
Drosophila melanogaster et D.simulans. Thèse pour
l'obtention du diplôme du Doctorat. Université Claude Bernard-
Lyon. France.
35. Sabot.F, Schulman.A.H (2006): Parasitism and the
retrotransposon life cycle in plants, a hitchicker's guide to the genome.
Heredity. 1-8.
36. Schulman.A.H, Kalendar.R (2005): A movable feast: diverse
retrotransposons and their contribution to Barley genome dynamics.
Cytogenetic and Genome Research. 110: 598-605.
37. Shao.H, Tu.Z (2001): Expanding the diversity of the
IS360-Tc1-mariner SuperFamily: Discovery of a unique DD37E transposon
and reclassification of the DD37D and DD39D transposons. Genetic Society of
America: 159. 1103-1115.
38. Silva.J.C, Loreto.E.L, Clark.J.B (2004): Factors that affect
the horizontal transfer of transposable elements. Current issues in
molecular biology. 6(1): 57-71.
39. Singer.M, Berg.P (1992): Gènes et génomes.
40. Tempel.S (2007) : Dynamique des helitrons dans
les génomes d'Arabidopsis thaliana, Développement de
nouvelles stratégies d'analyse des éléments transposables.
Thèse pour l'obtention du diplôme Doctorat en Biologie.
Université de Rennes. France.
41. Toumi.N (2001-2002) : Etude du transposon
mariner chez les organismes des sources hydrothermales
océaniques. DEA de Biologie, évolution et contrôle des
populations. Faculté des sciences et techniques. Université de
Tours- France.
42. Turner.P.C, Lenman.A.G, Bates.A.D, White.M.R.H (2002):
L'essentiel en biologie moléculaire. Berti 2dition : 386.
43. Watson, Gilmaw, Witkowski, Zoller (1994): L'ADN recombinant.
2ème edition. pp 175-190.
44. Yu.Z (2007): Identification of putative
retrotransposition in genes. A thesis submitted to the faculty of graduate
studies and research in partial fulfilement of the requirement of the master
degree of science. Departement of Biology. Mc Gill University. Montreal.
45. Zhang.X, Feschotte.C, Zhang.Q, Jiang.N, Eggleston.W.B,
Wessler.A.S (2001): P instability factor, an active Maize transposon
system associated with the
amplification of Tourist-like MITEs and a new
Superfamily of transposases. Genetics, Vol 98, n°22,
12572-12577.
Les sites Web
46.
www.biomedcentral.com
47.
www.inra.fr
48.
www.inrp.fr
49.
www.ncbi.nlm.nih.gov
50.
perso.univ-lyon2.fr
51.
PiggyBac-bio.nd.edu
52.
www.plantcell.org
53.
www.scribd.com
54.
www.univ-lemans.fr



