Cette partie est consacréà l'étude
conceptuelle de notre projet, oùnous présentons les
différentes étapes réalisées durant
l'implémentation de notre portail. Nous commençons par
décrire la conception de la base de donnée, ensuite nous
définissons les fonctionnalités et les services de notre
portail.
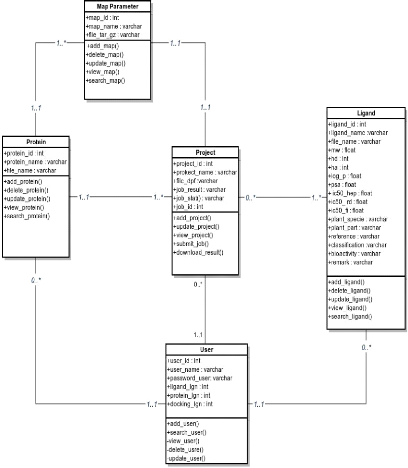
Nous avons élaborénotre base de donnée
pour le portail avec MySQL. Cinq tables font partie de notre conception :
· Ligand, elle contient toutes les
informations nécessaire concernant le ligand. L'at-tribut file name
est le fichier que l'utilisateur l'a
déjàpréparéavec AutoDockTools, et qui sera utiliser
lors du docking.
· Protein, cette table contient les
informations à propos du protéine. Elle un attribut important qui
sera utiliser dans le docking. Il s'agit de file name, qui est un
fichier de protéine que l'utilisateur l'a
déjàpréparéavec »ADT».
· Map Parameter, oùl'attribut
file tar gz contient les fichiers de paramètres de la grille,
que l'utilisateur les a déjàpréparéavec
autogrid4.
· Project, cette table contient tout les fichiers
nécessaires qui seront utilisés pour lancer des jobs de docking
sur la grille de calcul. L'attribut file dpf est le fichier de
paramètres de docking préparer par l'utilisateur avec
AutoDockTools.
· User, cette table contient les
credentials des utilisateurs enregistrés.
Afin de faciliter la tâche aux utilisateurs, nous avons
ajoutéune table pour les paramètres de grille. Au lieu de faire
entrer manuellement les coordonnées (X,Y et Z) des paramètres de
la grille, l'utilisateur ne doàýt que préparer les
fichiers des paramètres avec l'outil AutoDockTools.
La conception de la base de donnée pour notre portail
web est présentédans le schéma ci-dessous.
42
FIGURE 18 - Diagramme de classe du portail
web
mw : poids moléculaire hd : donneur
d'hydrogène ha : accepteur d'hydrogène log p : coefficient de
partition
· Cas d'utilisation
Les diagrammes ci-dessous présentent les cas
d'utilisation, afin de montrer les différentes interactions entre les
utilisateurs et le système. 4 types d'utilisateurs sont présents
dans notre conception, celui du ligand, du protéine, du docking et
l'administrateur du site.
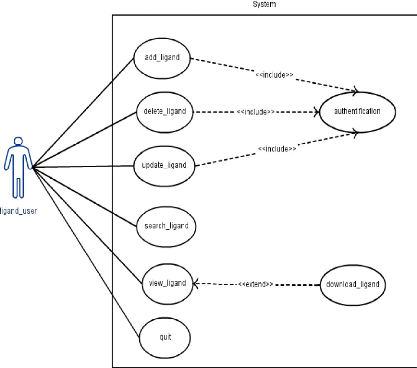
Le premier cas d'utilisation appartient au Ligand.
Oùl'utilisateur peut ajouter, modifier, supprimer un Ligand, mais cela
nécessite une authentification de l'utilisateur s'il possède un
compte, sinon il do^~t s'enregistrer avant d'effectuer ces opérations.
Tous les utilisateur peuvent consulter le catalogue des ligands disponibles,
voir les informations concernant ces ligands et chercher un ligand.
43
FIGURE 19 - Cas d'utilisation pour le
Ligand
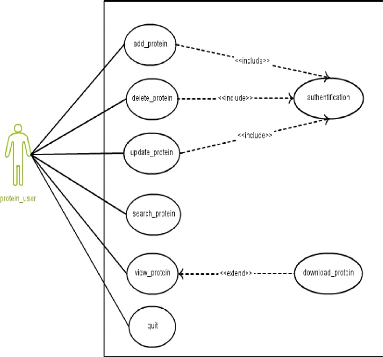
Le deuxième cas d'utilisation concerne la
molécule de protéine. Les utilisateurs authentifiés sont
capables d'effectuer les opérations d'ajout, suppression et
modification. Et les autres utilisateurs non-authentifiés peuvent
consulter la liste des protéines disponibles sur le portail, et
d'effectuer une recherche sur une protéine.
44
FIGURE 20 - Cas d'utilisation pour la
Protéine
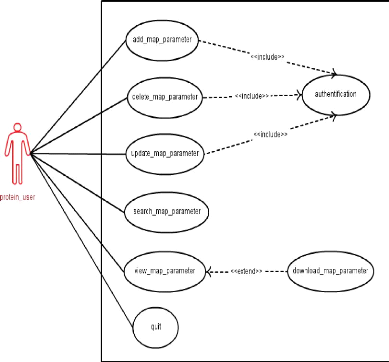
Le troisième cas d'utilisation appartient aux
paramètres de grille (map parameter). Tel que, une
protéine peut avoir plusieurs paramètres de grille. Les autres
utilisateurs qui ne possèdent pas de compte peuvent consulter la liste
des paramètres de grille.
45
FIGURE 21 - Cas d'utilisation pour les
paramètres de grille
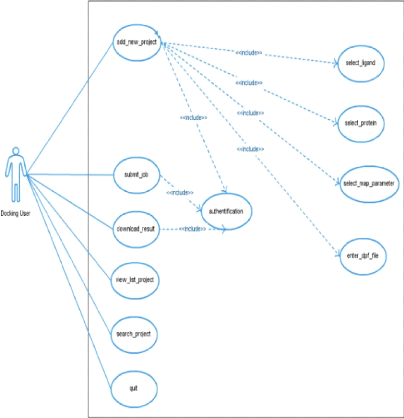
L'avant dernier cas d'utilisation, est le cas crucial dans
notre projet. Il s'agit d'effec-tuer l'amarrage. L'authentification des
utilisateurs est requises pour pouvoir créer un projet, soumettre le job
de docking et télécharger les résultats. Tout d'abord
l'utilisateur accède au portail, il s'authentifie s'il a l'intention
d'effectuer le docking. Puis il crée un projet, en choisissant les
fichiers dont il a besoin pour réaliser l'amar-rage, et après il
soumet son job sur la grille de calcul. À la fin, il peut
récupérer le résultat du job en
téléchargeant le fichier de docking.
46
FIGURE 22 - Cas d'utilisation pour le
docking
47
Le dernier cas d'utilisation concerne l'administrateur du
site. Ce dernier possède les droits de gérer la liste des
utilisateurs, des ligands, des protéines, des paramètres et les
projets de docking.
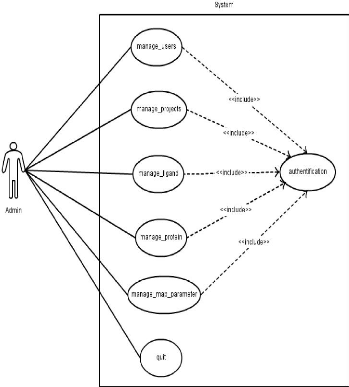
FIGURE 23 - Cas d'utilisation pour
l'administrateur du portail
3.4 Développement du portail du web
Nous avons eu recours à plusieurs technologies du web
pour le développement du portail. Parmi ces technologies : PHP comme
cadre de base et les services web JAX-WS avec JAVA pour interagir avec le
système du portail, tout en recevant les requêtes du client. Le
portail interagit avec le système via l'intermédiaire du web
service. Notre application est réalisée sous Linux et avec
Netbeans 8.0. Nous détaillons, dans ce qui suit, chacun des outils et
langages utilisés pour la manipulation des données ainsi pour
l'implémentation du portail web.
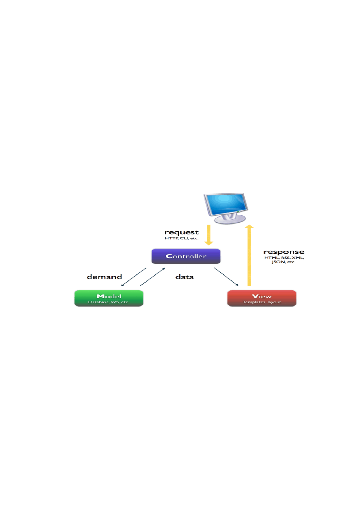
Le portail web a étédéveloppéen
utilisant le framework Yii (Yes It Is), qui est un framework PHP
basésur des composants ultra performant qui a
étédéveloppépour créer des application Web
de grande qualité, dont le but d'accélerer le
développement des applications Web. Yii est développéen
respectant le modèle MVC (Model-View-Controller).
FIGURE 24 - Modèle MVC
3.4.1 Les services web
Nous avons développétrois services web en Java
sous NetBeans 8.0 avec le serveur GlassFish
et Apache Axis2. Chaque service est responsable à une
fonctionnalitépour notre projet. Le premier service prend en
entrée les fichiers dpf & gpf , pour
générer les fichiers nécessaires (jdl et
shell), et il soumet les job de docking sur la grille de calcul via DIRAC
à partir des fichiers jdl qu'il les a générer. Le
deuxième service prend en entrée l'identifiant du job
(jobID) qu'il a soumet, et donne comme sortie l'état du
job. Si l'état du job est à (Failed), alors le
service re-programme le job et il re-soumet à nouveau le job avec la
commande de DIRAC (dirac-wins-file-reschedule jobID). Le dernier
service repose sur la récupération du résultat du job
depuis la grille de calcul. Il prend en entrée l'identifiant
»jobID» et l'état du job.
48
49
Si le statut du job est à l'état (Done),
alors le service récupère le résultat du job
àpartir de l'espace de stockage de la grille de calcul.
Après avoir exécuter le service web sous Netbeans, un script WSDL
(Web Service Description Language) a
étégénéré. L'URL de WSDL sera
utilisépour créer des workflows avec l'outil Taverna.
Le tableau ci-dessous présente en détail les
services web que nous avons implémenté, (les paramètres
d'entrées, les sorties et une description du rôle de chacun).
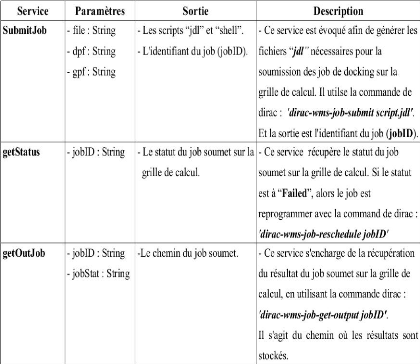
FIGURE 25 - Description des services web
implémentés
- Le service »submitJob», ce
service récupère les fichiers d'entrées à partir du
portail, puis il génère les scripts »jdl» dans
un dossier. Après la génération des scripts, les jobs sont
soumis sur la grille de calcul à travers l'intergiciel
»DIRAC» en utilisant la commande
»dirac-wins-job-subinit».
- Le service »getStatus», le
rôle de ce service est de suivre l'état des jobs soumis sur la
grille avec la commande »dirac-wins-job-status». Si
l'état du job est à»Failed»,
alors le service re-programme la soumission du job avec la commande de
DIRAC »dirac-wins-job-reschedule».
50
- Le service »getOutJob», sert
à récupérer le résultat du docking à partir
de la grille via la commande DIRAC »dirac-wins-job-get-output»
si et seulement si l'état du job est à
»Done».
Toutes les opérations de service (SubmitJob,
getStatus, getOutJob) ont ététestées en utilisant Taverna.
Et chaque opération dispose de son propre workflow. Nous passons
à l'étape crucial de notre travail, et qui repose sur le
déployant de l'outil Taverna, qui est un outil très
utilisépour créer et visualiser des workflows scientifique. Le
workflow a étémis en oeuvre en utilisant l'adresse de service
(http: // localhost: 8080/ TavernaWS/ WSSubmitJob? Tester) avec l'outil
Taverna Workbench, pour être exécutéà
travers l'intergiciel »DIRAC».
51
La figure suivante illustre le workflow des services web
implémentés pour le portail web avec Taverna Workbench
:
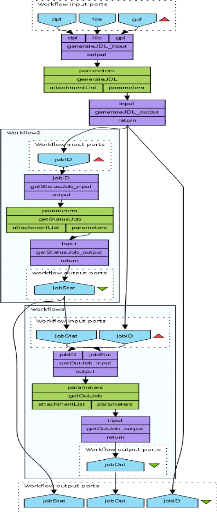
FIGURE 26 - Workflow des services web du
portail
52
4 Expérimentation & Résultats
Cette partie se focalisera sur la démonstration du
portail web et les résultats du docking. La figure ci-après
présente la fenêtre principale de notre portail. Ce portail a
étéconçu pour les utilisateurs qui ne sont pas
forcément des experts en informatique (biologistes, chimistes,
bio-informaticiens,...), pour qu'ils puissent effectuer le criblage virtuel
in-silico sur la grille de calcul. Les visiteurs peuvent
accéder au portail afin de consulter la liste des ligands,
protéines, les paramètres de grille et les projets qui ont
étédéjàsoumet sur la grille. Mais s'ils veulent
effectuer une tâche ils doivent, tout d'abord, s'enregistrer ou
s'authentifier en fournissant leur nom et leur mot de passe.
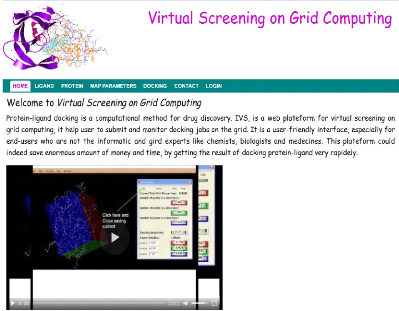
FIGURE 27 - Interface d'accueil du portail
web
L'utilisateur accède au portail par une simple
authentification (nom & mot de passe). La figure ci-dessous montre
l'interface pour s'enregistrer auprès du portail afin de profiter de ses
services. L'utilisateur entre son nom et son mot de passe, et il doit choisir
sa catégorie (soit ligand, soit protéine ou docking).
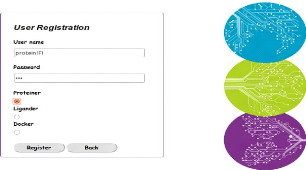
FIGURE 28 - Interface de création d'un
nouveau compte
Après la phase d'enregistrement, l'utilisateur do^~t
s'authentifier pour bien profiter des privilèges qu'un visiteur normal
ne possède pas.

53
FIGURE 29 - Interface d'authentification
L'administrateur du site possède tous les droits pour
la gestion du site. Il gére les utilisateurs, les ligands, les
protéines, les paramètres de la grille et les projets de
docking.
La figure ci-dessous sollicite la liste des utilisateurs.
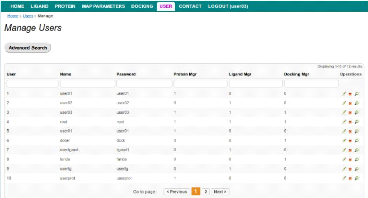
FIGURE 30 - Interface de gestion des utilisateurs
Après avoir créer un compte, l'utilisateur peut
accéder au portail en fournissant ses credentials pour accomplir sa
tâche. Si ce dernier possède un compte, et il appartient à
la catégorie de »ligand», alors il peut ajouter,
modifier son ligand, et il peut le supprimer. La capture d'écran
ci-dessous montre l'ajout d'une nouvelle molécule de ligand par
l'utilisateur.
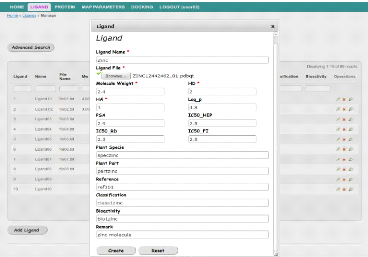
FIGURE 31 - Interface d'ajout d'un nouveau Ligand
54
Les autres visiteurs du portail non-authentifiés
peuvent consulter la liste des ligands disponibles sur le portail et effectuer
une recherche sur une molécule de ligand.
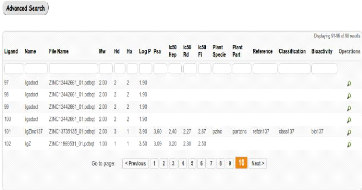
FIGURE 32 - Interface de liste des Ligands disponibles
L'administrateur de protéine peut gérer la
liste des molécules de protéines, tout en effectuant les
opérations d'ajout, de modification et de suppression. Ci-dessous, une
figure qui montre la fenêtre pour la gestion des protéines.
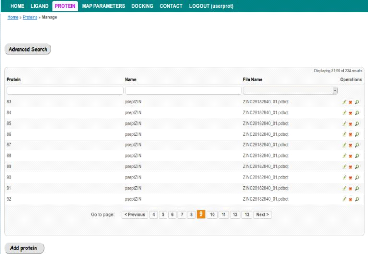
FIGURE 33 - Interface de gestion des protéines
55
56
La modification d'une protéine nécessite
l'authentification de l'administrateur de protéine, comme c'est
illustrédans la capture ci-après.
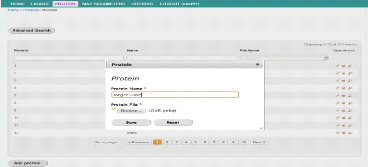
FIGURE 34 - Interface de modification d'une
protéine
Comme nous avons citédans la partie
implémentation, la relation entre la protéine et les
paramètres de la grille est une relation (1 :n). Tel que, une
protéine peut avoir plusieurs paramètres, mais un
paramètre de la grille appartient à une et une seule
molécule de protéine.
La capture suivante illustre l'ajout d'un fichier de
paramètre de la grille.
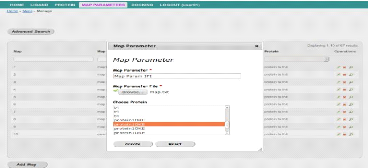
FIGURE 35 - Interface d'ajout de fichier de
paramètres de la grille
On arrive à la partie importante dans ce projet, elle
consiste à créer un projet pour le docking et soumettre les jobs
de docking sur la grille de calcul. Afin de pouvoir créer un nouveau
projet, l'utilisateur doit s'authentifier en fournissant ses credentials (son
nom et son mot de passe), sinon il do^~t créer un compte. Après
avoir authentifier, l'utilisateur est capable de créer un nouveau
projet, pour cela il do^~t choisir un ligand, une protéine, un fichier
de paramètre et aussi le fichier de paramètres pour le docking
(dpf). Pour ces paramètres d'entrées sélectionnés,
soit il les a déjàpréparer lui même, ou par d'autres
utilisateurs. La capture ci-après illustre la création d'un
nouveau projet pour le docking.
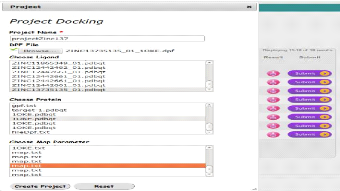
57
FIGURE 36 - Interface d'ajout d'un nouveau projet de docking
En retournant à la liste des projets qui ont
étécréés, on peut vérifier que le projet a
étéajoutéavec succès.
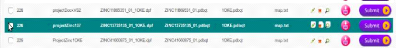
FIGURE 37 - Vérification d'ajout du nouveau projet
Après avoir créer un projet et choisir les
fichiers nécessaires pour effectuer le docking. L'utilisateur n'a
qu'appuyer sur le bouton »submit» pour soumettre son job sur
la grille. Le bouton va récupérer les fichiers à partir du
portail, puis, il fait appel à Ta-verna client qui
récupère le workflow contenant les services web. Après, il
soumet le job sur la grille de calcul. Nous allons présenter les
résultats que nous avons obtenus lors de soumission des jobs de docking
sur la grille, et la récupération des résultats à
partir de la grille à travers de ce portail web. Nous nous sommes servis
de la base de donnée ZINC ( http: //
zinc. docking.
org) , qui est une base de
donnée de composés disponibles pour le criblage virtuel (1OKE
pour la protéine, et ZINC pour le ligand), oùle fichier de
ligand comprend 10256 composants. Les fichiers de paramètres pour la
grille grid parameter et pour le docking dock parameter(dpf
& gpf) ont déjàétépréparé.
L'étape de docking moléculaire est réalisée
grâce au sous-programme AutoDock, qui recherche toute les solutions
d'amarrage en fonction des paramètres du fichier »dpf»
que l'utilisateur à déjàpréparer. Après
l'achèvement du docking, les résultats ont
étégénérés dans un fihier log avec
l'extension (glg & dlg). Le fichier »glg» contient les
affinités calculer entre les différentes types d'atomes de la
protéine et le ligand. Et le fichier »dlg», qui fournit les
coordonnées atomiques des 10 meilleurs positions du ligand dans le site
de la protéine, leur énergie d'in-teraction ainsi que les
différentes valeurs de l'écart quadratique moyen (Root Mean
Square Deviation ou le »RMSD»).
La figure ci-après montre la soumission de job de
docking, tout en sélectionnant le projet »projectZinc137».
Dans ce projet nous avons choisi: le fichier de paramètre de
docking ZINC13735135 01 1OKE.dpf , le ligand ZINC13735135
01.pdbqt, la protéine 1OKE.pdbqt et le fichier de
paramètre de la grille map.txt.
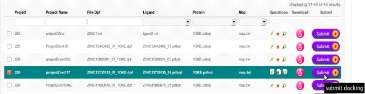
FIGURE 38 - Soumission de job de docking
Après avoir soumettre le job avec le bouton
submit, le résultat consiste en fichier zip,
oùnous l'avons spécifier dans le script jdl. Le fichier
est stokés sur l'espace de stockage de la grille de calcul, puis nous le
récupérons depuis le SE de la grille via la commande DIRAC
»dirac-dms-get-file». Ce dernier contient le fichier de
docking »dlg». La capture ci-après montre le
résultat du job stocker sur notre espace de stockage de la grille, comme
c'est illustrédans la capture ci-dessous.

Le résultat est compresséet stockésur
l'élément de stockage de la grille de calcul, puis, le
résultat est récupéréà partir de la grille
de calcul à l'aide de la commande DIRAC, pour que
l'utilisateur puisse le télécharger. La figure ci-après
montre le téléchargement du résultat du job de docking
soumis. Le fichier contient les fichiers log de docking et de grille
(»dlg» & »glg»).
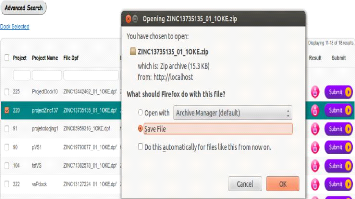
FIGURE 39 - Téléchargement du résultat de
docking
58
59
Après avoir télécharger le fichier
»zip», nous avons vérifiési l'opération
du docking a étéeffctuéavec succès, en ouvrant le
fichier log de docking. La capture ci-dessous
illustre le résultat du docking. Comme montre la
figure suivante le docking a étéachevéavec
succès.


FIGURE 40 - Fichier log de docking »dlg»
Nous avons aussi testéce portail pour soumettre un
autre job de docking du projet : ProjectZinc1OKE, qui comprend
les fichiers de paramètres suivants : fichier de paramètres de
docking (dpf) »ZINC4166-0875 01 1OKE.dpf»,
fichier ligand »ZINC41660875 01 1OKE.pdbqt», fichier
protéine »1OKE.pdbqt».

FIGURE 41 - Soumission du projet de docking
ProjectZinc1OKE
60
L'utilisateur peut récupérer son réultat
du job dès que l'opération du docking s'achèvera. Le
résultat est illustréci-après.
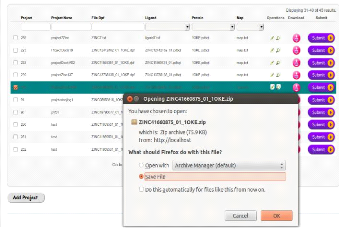
FIGURE 42 - Téléchargement du résultat de
docking
On peut voir que le résultat du docking est bien
enregistrédans l'espace de stockage de la grille de calcul,
oùnous avons compresséles fichiers résultant du
docking.

FIGURE 43 - Enregistrement du résultat du job sur la
grille de calcul
Comme nous avons déjàmentionné, le
résultat du docking consiste en deux fichiers »dlg (docking log
file) 4 glg (grid log file)», mais le fichier le plus important est
le fichier »dlg».
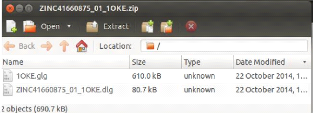
FIGURE 44 - Les fichiers dlg 4 glg du docking
61
La capture ci-après montre que le docking s'est
effectuéavec succès.
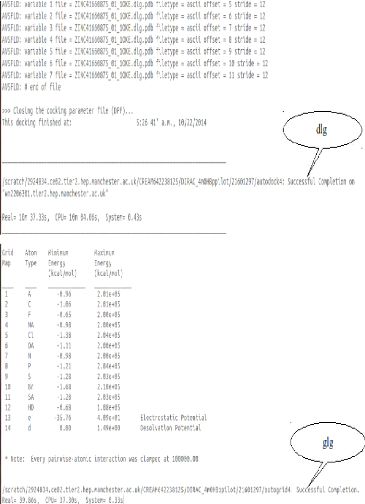
FIGURE 45 - Les fichiers log de docking et de
la grille dlg & glg
Rappelons que le but essentiel de l'utilisation de la grille
de calcul, est la possibilitéde soumettre plusieurs jobs en
parallèle. L'utilisateur peut soumettre plusieurs jobs
de docking sur la grille de calcul via l'intergiciel DIRAC.
Pour cela, il suffit de préparer les fichiers nécessaires pour
réaliser cette opération.
Nous avons préparéles fichiers de
paramètres de docking »dpf» que nous voulons utiliser
afin d'effectuer le docking. Pour soumettre des jobs en parallèle
à l'aide de l'intergiciel DIRAC, nous avons utiliséun workflow.
Ce workflow va générer les
62
fichiers »jdl» essentiels, puis il les
soumets sur la la grille de calcul pour que le Worker Node puisse
exécuter ces jobs. Les résultats des jobs sont
compressédans un fichier zip, ensuite stockésur
l'espace de stockage de la grille. Après que l'opération du
docking s'est terminé, on peut récupérer les
résultat du docking à partir du portail. Afin de montrer la
procédure de docking avec plusieurs fichiers, nous avons préparer
le fichiers de docking »ZINC4.txt», qui comporte les
fichiers ci-dessous :
> ZINC41584388 01 1OKE.dpf >
ZINC41584391 01 1OKE.dpf > ZINC41584955 01 1OKE.dpf >
ZINC41584955 02 1OKE.dpf > ZINC41584983 03
1OKE.dpf
Après avoir soumettre le job, le résultat
consiste en un fichier compresséque nous l'avons récupérer
depuis l'espace de stockage de la grille dès que le docking s'est
achevé. La capture ci-après présente le résultat de
soumission de plusieurs jobs de docking soumis en parallèle.
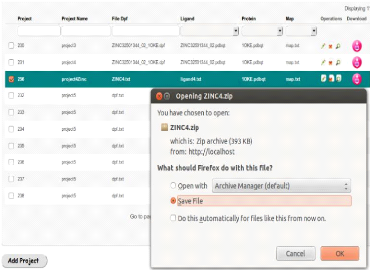
FIGURE 46 - Téléchargement du
résultat des jobs
On accédant à notre espace de stockage de
grille de calcul, on remarque que le résultat du job est bien
stocké. La capture ci-dessous montre le fichiers de paramètre de
docking (ZINC4.txt) et le réusltat (ZINC4.zip).

63
Ce fichier comprend les résultats de tous les jobs
soumet sur la grille de calcul, et chaque fichier contient les deux fichiers
log dlg (docking log file) & glg (grid log file).
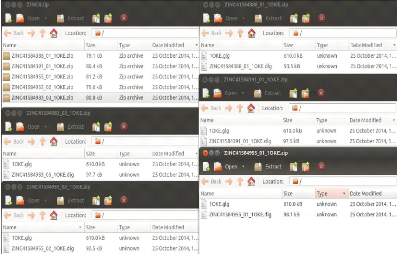
FIGURE 47 - Les fichiers des jobs soumis en
parallèle
Mais parfois dûà une mauvaise connexion et
à la non convivialitédes commandes DIRAC, nous ne pouvons pas
récupérer les résultats depuis la grille. Et lors de
récupération des résultats jobs depuis la grille, on s'est
rendu compte que quelques résultats ne sont pas bonnes. Et cela
s'explique par le fait que les données soit de la molécule du
protéine ou celle des ligands contient des informations erronéet
incompatibles.



