CHAPITRE 2
MATERIEL ET
METHODES
34
Chpaitr 02 : Materiels et méthodes
Matériel et méthodes
Cette étude portant sur l'évaluation de la
résistance aux antibiotiques des entérobactéries est
divisée en deux étapes :
- La première étape se propose de faire une
étude rétrospective des données, de l'isolement des
entérobactéries provenant de malades externes ou
hospitalisés dans les différents services du CHU SAADNA Mohamed
Abdennour de Sétif, durant la période de Janvier 2013 à
Avril 2017. Le logiciel de sauvegarde «Whonet 5.6 » utilisé au
laboratoire a été notre source d'information.
- Dans un deuxième temps, les
entérobactéries BLSE positifs isolées durant
l'année 2017, sont caractérisées du point de vue
moléculaire par l'extraction de plasmides et la détermination des
gènes de résistance codant pour ces â-lactamases en
utilisant une PCR-Multiplex.
1- Prélèvement et
identification
Les prélèvements issus des malades des
différents services hospitaliers ou ceux du milieu extra hospitalier
sont recueillis et guidés selon la nature du prélèvement
à la paillasse approprié comme suit :
- Paillasse Divers(1) : Pus, PB, Crachat.
- Paillasse Divers(2) : PV, KT, Sonde.
- Paillasse ECBU : Urine.
- Paillasse Hémoculture : Sang.
- Paillasse Coproculture : Selles, PR.
- Paillasse Ponction : LCR, LP, Liquide
Sinovial...
Après isolement et purification des
entérobactéries sur milieu sélectif BCP ou Hektoen. Leur
caractérisation est déterminée par une identification
biochimiques en utilisant différentes méthodes, la galerie
classique, la galerie Api 20E (la plus pratique) ou l'automate
SIEMENS MicroScan WalkAway.
1-1- La mini galerie (galerie classique)
La mini galerie repose sur plusieurs tests biochimiques
? L'urée-indole : permettant la recherche
de l'uréase et de l'indole
35
Chpaitr 02 : Materiels et méthodes
· La gélose TSI (Triple Sugar Iron)
:l'utilisation des sucres (glucose, lactose et saccharose) est
détectée par le virage de l'indicateur de pH, et la production
H2S est révélée la présence d'un
précipité noir.
· La gélose Mannitol-Mobilité :
l'utilisation du mannitol (changement de couleur du rouge au jaune) et
de la mobilité du germe.
· La gélose citrate de Simmons :
utilisé pour étudier la capacité de la
bactérie à croître en présence du citrate de sodium
comme seule source de carbone.
· Le milieu Clarck & Lubs : la mise
en évidence des voies fermentaires, voie du butan-2,3-diol (VP+) ou voie
des acides mixtes (RM+).
· Les acides aminés (ADH-ODC-LDC) :
permettant de voir le métabolisme protéique pour
différencier chaque espèce d'une autre.
Les milieux ensemencés sont incubés à
37°C/24h et la lecture se fait selon le test. 1-2- La galerie Api
20E
L'identification par la galerie rapide API 20E
(Analytical profil index) est un système pour l'identification des
entérobactéries utilisant 20 tests biochimiques
standardisés et miniaturisés, ainsi qu'une base de
données.
La galerie est ensemencée, à l'aide d'une
pipette Pasteur, par une suspension bactérienne dont l'opacité
est équivalente à 0,5 McFarland. Les tubes et les cupules de la
galerie sont remplis selon les recommandations du fournisseur. L'incubation se
fait à 37 C° #177; 1C° pendant 18-24h.
La lecture des galeries se fait selon les indications du
fournisseur. Après codification des réactions en un profil
numérique, on se réfère à un catalogue analytique
où l'identification est donnée avec un pourcentage et une
appréciation.
1-3- Le systeme MicroScan WalkAways SIEMENS
Le système WalkAways est un automate-incubateur
relié à un ordinateur comportant le logiciel d'identification des
bactéries, il ressemble à la galerie Api 20E mais de
manière plus sophistiquée. Ce système utilise des plaques
de 96 puits avec des réactifs lyophilisés et chaque
microorganisme à sa plaque spécifique comme suit :
· Combo 54 : bacille Gram négatif non fermentant
(Lactose -).
· Combo 53 : bacille Gram négatif fermentant
(Lactose +).
36
Chpaitr 02 : Materiels et méthodes
? Combo 28 : cocci Gram positif
(Staphylocoques).
? Combo PC1A : cocci Gram positif
(Streptocoques).
La plaque ensemencée sera incubée dans
l'automate pendent 24h/37°C, puis elle sera lu grâce au logiciel et
le résultat est imprimé.
2- Antibiogramme
Une fois la souche isolée et purifiée, sa
sensibilité aux antibiotiques est testée par l'antibiogramme
standard réalisé par la méthode de diffusion des
disques(CA-SFM/EUCAST, 2014) qui permet la détermination du
phénotype sensible (S) de l'espèce et les différentes
résistances acquises (I ou R) avec des termes de diminution
significative de sensibilité (synergie, antagonisme).Une suspension
bactérienne préparée à partir d'une culture pure de
18-24 h est ajustée au standard 0,5 McFarland. A partir de cette
suspension bactérienne diluée au 1/10 dans l'eau physiologique
(0,9 % NaCl) des géloses de Mueller-Hinton sont ensemencées par
écouvillonnage. Après séchage des boîtes, des
disques de papier buvard imprégnés d'antibiotiques sont
déposés à la surface du milieu manuellement ou à
l'aide d'un distributeur automatique. L'incubation se fait 37 °C pendant
18 à 24h. La mesure du diamètre des zones d'inhibition autour de
chaque disque et comparée aux standards du CA-SFM et permet de
déterminer les phénotypes sensibles (S), intermédiaires
(I) et résistants (R).
Par l'automate, l'antibiogramme est
effectué grâce aux antibiotiques lyophilisés dans la
plaque. L'automate peut tester la sensibilité d'une souche envers un
antibiotique. Dans ce cas, l'inoculum préparé est valable
à la fois pour l'identification et l'antibiogramme. La plaque contient
les réactifs pour identifier et pour tester la sensibilité en
même temps, d'où l'avantage de l'automate, identification et
antibiogramme en 24h.
37
Chpaitr 02 : Materiels et méthodes
Tableau 4. Antibiotiques testés.
Antibiotiques
|
Abréviations
|
Charge (jig)
|
Antibiotiques
|
Abréviations
|
CH (jig/UI)
|
Ampicille
|
AMP
|
10
|
|
Ertapeneme
|
ETP
|
10
|
|
Amoxicilline
|
AMX
|
25
|
|
Meropeneme
|
MEP
|
10
|
|
Ticarcilline
|
TIC
|
|
|
Imipeneme
|
IPM
|
10
|
|
Amoxicilline/ Acide
Clavulanique
|
AMC
|
25 /
|
10
|
Acide
Nalidixique
|
AN
|
30
|
|
Piperacilline/ Tazobactam
|
PTZ
|
75 /
|
10
|
Gentamycine
|
GM
|
10
|
|
Aztreonam
|
AZM
|
30
|
|
Amikacine
|
AMK
|
30
|
|
Cefazoline
|
CZO
|
30
|
|
Tobramycine
|
TOB
|
10
|
|
Cefuroxime
|
CXM
|
30
|
|
Tigecilline
|
TGY
|
30
|
|
Cephalothine
|
CEP
|
30
|
|
Minocycline
|
MIC
|
30
|
|
Cefoxitine
|
FOX
|
30
|
|
Ciprofloxacin
|
CIP
|
5
|
|
Cefepime
|
FEP
|
30
|
|
Levofloxacin
|
LVX
|
5
|
|
Cefotaxime
|
CTX
|
30
|
|
Fosfomycine
|
FOS
|
50
|
|
Ceftazidime
|
CAZ
|
30
|
|
Sulfaméthoxa zole-
triméthoprim
|
SXT
|
1.25
|
/ 23.75
|
|
3- Détection phénotypique des
EBLSE
Dans le cas de l'automate, la détection des EBLSE est
automatique par la technique des CMI. Par contre, sur l'antibiogramme standard
la réduction de la sensibilité aux céphalosporines de
troisième génération nécessite la mise en
évidence de l'existence d'une BLSE par la recherche d'une synergie entre
l'acide clavulanique et les céphalosporines de troisième
génération selon les techniques suivantes :
38
Chpaitr 02 : Materiels et méthodes
3-1- Test de synergie
La détection de f3-lactamases à spectre
élargi peut être mise en évidence par la méthode des
disques consistant à rechercher une image de synergie entre un disque
d'antibiotique contenant un inhibiteur de f3-lactamase et les disques de
céphalosporines de troisième génération
(cefotaxime, ceftazidime et cefopérazone) et l'aztréonam. Cette
synergie se traduitpar l'élargissement de la zone d'inhibition en regard
du disque de C3G sous forme d'un "bouchon de champagne" (Jarlier et
al. 1988).
Les disques d'amoxicilline + acide clavulanique (AMC 20/10)
et les disques de C3G (CTX 30ìg, FEP 30ìg et CAZ 30ìg) et
l'aztréonam (ATM 30ìg) sont déposés à la
surface du milieu MH à une distance de 20 à 30 mm entre les
disques. Après incubation à 37°C/18hle test est
considéré positif quand le diamètre d'inhibition du disque
de céphalosporine de troisième génération
appliqué après pré-diffusion du disque de l'AMC est
supérieur ou égale de 4 à 5 mm par rapport au
diamètre d'inhibition du disque C3G.
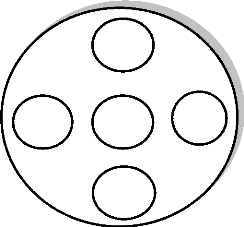
CAZ
AMC
ATM
CTX
FEP
Figure 7. Disposition des disques d'antibiotique
pour le test de synergie.
3-2- Test du double disque
Le test espagnol comme il est appelé, consiste
à rechercher une augmentation de la zone d'inhibition d'un disque de
C3G, précédé par l'application d'un disque contenant
l'AMC, comparé à un autre disque portant la même
céphalosporine et placé côte à côte sur la
gélose
39
Chpaitr 02 : Materiels et méthodes
de MH (Rahal et al. 2005).On procède de la
même manière que la technique d'antibiogramme dans la
préparation de l'inoculum et l'ensemencement, puis on place deux
antibiotiques : un disque d'AMC et un disque de CTX (C3G) à une distance
de 25 mm selon la figure. On laisse diffuser à température
ambiante pendant une heure et on remplace le disque d'AMC par un disque de CTX
(C3G).On incube pendant 18 h à 37°C. Le test du double disque est
considéré positif quand le diamètre d'inhibition du disque
de céphalosporine de troisième génération
appliqué après pré-diffusion du disque de l'AMC est
supérieur ou égale de 4 à 5 mm par rapport au
diamètre d'inhibition du disque C3G.

CTX
CTX
1 heure
Après
AMC CTX
Figure 8. Schéma de détection de
BLSE par le test du double disque
(Rahal et al. 2005).
3-3- Test à la cloxacilline
Le test à la cloxacilline est effectué pour
identifier une BLSE associée à une céphalosporinase
déréprimée. La cloxacilline ajoutée au milieu MH
(0,25 mg/ml) inhibe très fortement toutes les céphalosporinases
hyper-produites (Drieux et al. 2008 ; Philippon et Arlet. 2006). Si un
tel mécanisme de résistance existe, les boites de Pétri
contenant le milieu MH additionné de cloxacilline présentent une
restauration de l'activité de â-lactamases et apparition de
l'image de synergie en bouchon de champagne recherchée.
40
Chpaitr 02 : Materiels et méthodes
4- Détection génotypique des
EBLSE
4- 1- Extraction d'ADN
L'extraction est réalisée par deux
méthodes selon la nature plasmidique ou chromosomique de l'ADN.
a) Extraction d'ADN plasmidique par la lyse
alcaline
La méthode de la lyse alcaline est une technique
rapide permettant l'extraction des plasmides (ADN extra-chromosomiques) de
différentes tailles, de petite et de grande taille (>100kb).Elle met
à profit les propriétés structurales de l'ADN plasmidique,
qui est libérée après la lyse des bactéries sous
des conditions dénaturant l'ADN chromosomique (Kado et Liu. 1981).
L'extraction est réalisée selon le protocole
suivant :
- Des cultures bactériennes sur milieu LB sont
réalisées la veille.1ml de la culture est centrifugé dans
une tube Eppendorf (13000 rpm/1 min). Le culot ainsi obtenu est resuspendu dans
300 ìl du tampon P1 et agité.300 ìl du tampon P2 sont
ajoutés (tampon de lyse) et les tubes sont agités par inversion
environ 6 fois.300 ìl du tampon P3 est additionné et le tube est
agité par inversion (formation d'un précipité).Le
précipité est centrifugé à 13000 rpm/10 mn et le
surnageant est transféré dans un nouveau tube Eppendorf auquel
sont ajoutés 630ìl d'isopropanol refroidie à -20°C
pour précipiter l'ADN. Après centrifugationà13000 rpm/15
mn le surnageant est éliminé et le culot est lavé de ses
sels avec 700ìl d'éthanol à 70%. L'alcool
éliminé, le culot est, ensuite, séché à
l'air libre ou à l'étuve puis il est resuspendu dans 50ìl
de TE (Tris/HCl-EDTA) et conservé à -20°C.
Tableau 5. Composition des différents
tampons d'extraction.
Tampon P1
|
- 50 mM Tris/HCl (pH 8) - 10 mM EDTA (pH 8) - 1 ìl
d'ARNase
|
Tampon P2
|
- 0.2 M NaOH - 1 % SDS
|
Tampon P3
|
- 88.3 g K-acétate
- 15 ml acide formique - 300 ml H2O
|
|
41
42
Chpaitr 02 : Materiels et méthodes
b) Extraction de l'ADN chromosomique par
Boiling
L'ADN des entérobactéries est extrait par la
méthode du boiling. 1.5ml de la culture sur milieu LB est
centrifugé à 12000 rpm/1mn. Le culot bactérien est
resuspendu dans 180 ìl de TE et agité. Ce dernier est
porté à ébullition à 100 °C/10mn puis refroidi
à -20°C/10mn. Après centrifugation à 12000rpm/10 mn,
le surnageant est récupéré dans un nouveau tube Eppendorf
et conservé à 4°C pendant une nuit pour éventuelle
dégradation de l'ARN puis à - 20°C pour une utilisation
ultérieure.
4-2- Amplification par PCR (Polymerase Chain
Reaction)
La recherche des gènes de résistance
blaCTX-M, blaSHV et blaTEM a été
effectué par une PCR-Multiplex.
4-2-1- Principe
La technique de réaction de polymérisation en
chaîne ou PCR permet d'amplifier en un nombre élevé de
copies une séquence particulière d'ADN. La polymérisation
se réalise dans un mélange réactionnel contenant de
faibles quantités d'ADN possédant la séquence à
amplifier, les amorces nucléotidiques complémentaires des
séquences qui encadrent la cible à amplifier, l'ADN
polymérase et un mélange des quatre dNTP (dATP, dTTP, dCTP et
dGTP). Elle débute par une étape de dénaturation thermique
de l'ADN à amplifier. Des séquences oligonucléotidiques
complémentaires ou amorces sont alors hybridées aux
extrémités 3' des deux brins du fragment d'ADN matrice.
L'allongement des amorces dans le sens 5'--> 3' est, ensuite, assuré
par l'ADN polymérase Taq. Les nouvelles molécules d'ADN ainsi
formées sont dénaturées et un nouveau cycle peut
commencer, la réaction se répétant ainsi jusqu'à
plusieurs dizaines de fois (Bonnet et al. 2000b ; Hennequin et
al. 2012).
Tableau 6. Amorces utilisées (Jouini et
al. 2007).
Génes
|
Amorces
|
Séquences
|
Tm°C
|
blaCTX-M
|
For
|
5'-CTC CGC TTT GCG ATG TGA AG -3'
|
59.4
|
|
Rev
|
5'-ACC GCG ATA TCG TTG GT -3'
|
52.8
|
blaSHV
|
For
|
5'-CAC TCA AGG ATG TAT TGT G -3'
|
52.4
|
|
Rev
|
5'-TTA GCG TTG CCA GTG CTC G -3'
|
58.8
|
blaTEM
|
For
|
5'-ATT CTT GAA GAC GAA AGG GC-3'
|
55.3
|
|
Rev
|
5'-ACG CTC AGT GGA ACG AAA AC-3'
|
57.3
|
|
Chpaitr 02 : Materiels et méthodes
La réaction PCR d'un volume final de 25 ìl, est
composée de
- dNTP 0,5ul,
- MgCl2+ tampon 4ul,
- Taq-polymérase (solis biodine) 0,3ul,
- ADN de l'échantillon 5ul,
- Amorces spécifiques du gène à
amplifier : 0,2ul (de chaque),
- H2O (eau ultra pure stérile) qsp 25ul.
L'amplification de l'ADN se déroule selon les
étapes décrites ci -dessous
Tableau 7.Etapes de la PCR-Multiplex.
blaCTX-M/SHV-TEM
|
|
|
Dénaturation initial
|
94°C
|
/ 5 min
|
Dénaturation
|
94°C
|
/ 1 min
|
Hybridation
|
52°C
|
/ 1 min
|
Elongation
|
72°C
|
/ 1 min
|
Elongation final
|
72°C
|
/ 5 min
|
Nombre de cycles
|
30
|
|
|
Pour chaque PCR, il faut toujours un témoin positif et un
témoin négatif. L'amplification se déroule dans un
thermocycleur TC-4000.

Figure 9. Photo du thermocycleur TC-4000.
Chpaitr 02 : Materiels et méthodes
4-3- Electrophorèse sur gel d'agarose
Les produits d'amplification sont analysés sur gel
d'agarose à 1.5% (p/v) contenant 10ul d'une solution de bromure
d'éthidium (0,5 mg/l).Par contre, l'analyse des plasmides se fait sur un
gel à 0.8%. La migration est réalisé pendant 30mn à
100V et le gel est visualisé sous lumière UV et
photographié.

Figure 10. Photo d'une migration d'ADN par
électrophorèse.

43
Figure 11. Photo du transilluminateur
Fisher-Bioblock UV.
| 


