|
N° d'ordre /Master BMC

|
UNIVERSITE Joseph KI-ZERBO
|
Centre National de la Recherche Scientifique et
N° d'ordreMaster IA
Technologique (CNRST)
|
|
|
|
|
|
|
|
|
|
|
|
Unité de Formation et de Recherche en Sciences
de la Vie et de la Terre (UFR-SVT)
Département de
Biochimie-Microbiologie
Laboratoire de Biotechnologie et d'Immunologie
Appliquée
(LaBIA)
|
Institut de l'Environnement et de Recherches N°
d'ordre......
Agricoles (INERA)
|
|
Centre de Recherches Environnementales, Agricoles et
de Formation de Kamboinsé 'ordre...
(CREAF/K)
|
|
Laboratoire de Virologie et de
Biotechnologies
Végétales (LVBV)
|
MÉMOIRE
Présenté par :
AKAKPO Ayékitan
Pour l'obtention du diplôme de :
Master 2 en Biotechnologies
Option :
Biotechnologie Microbienne et
Cellulaire (BMC)
sur le thème
|
Diversité des Begomovirus infectant le
gombo [Abelmoschus esculentus (L.) Moench]
au Togo
|
Soutenu publiquement le 24/10/2019 à
l'Université Joseph KI-ZERBO devant le jury ainsi composé :
Président : Pr Nicolas BARRO,
Professeur Titulaire, Université Joseph KI-ZERBO, Burkina Faso
Membres : Pr Aly SAVADOGO, Professeur Titulaire,
Université Joseph KI-ZERBO, Burkina Faso
Dr Cheikna ZONGO, Maitre de
Conférences, Université Joseph KI-ZERBO, Burkina Faso, Directeur
de mémoire
Dr Fidèle TIENDREBEOGO, Chargé
de Recherche, INERA/CNRST, Ouagadougou, Burkina Faso, Co-Directeur de
mémoire
Année académique : 2018-2019
i
Dédicaces, Remerciements, Table des
matières
Dédicaces
Je dédie ce travail :
À mon père AKAKPO Mamah et
à ma mère ABDOU Zouréhatou : merci pour
votre amour, soutien, encouragement, patience et bénédictions
durant toutes mes années d'études. Permettez-moi
de vous témoigner ma profonde gratitude à travers ce modeste
travail. Puisse Dieu vous garde en bonne santé aussi longtemps
auprès de nous afin que vous puissiez bénéficier de vos
multiples sacrifices.
À ma compagne ADEKPLOVI Akouvi Rosalie
: merci pour ton amour, tes encouragements, ta patience et ta
compréhension tout au long de ces années studieuses.
À mes soeurs adorées : AKAKPO
Améyédé et AKAKPO Tinnon, merci
infiniment de vos soutiens. Je vous souhaite une vie pleine de bonheur et de
réussite.
À mes adorables frères : AKAKPO
Médjidon et AKAKPO Tinin,
AYENA-KASSEGNE Yaovi, que Dieu vous bénisse et comble
votre vie de bonheur et de réussite.
À mon oncle SABI Onyankitan :
merci de votre soutien moral et financier tout au long de mes
études.
II
Dédicaces, Remerciements, Table des
matières
Remerciements
Les travaux présentés dans ce mémoire ont
été effectués à l'Institut de l'Environnement et de
Recherches Agricoles (INERA), au Centre de Recherches Environnementales,
Agricoles et de Formation de Kamboinsé (CREAF/K), plus
précisément au Laboratoire de Virologie et de Biotechnologies
Végétales (LVBV). Plusieurs personnes ont contribué de
près ou de loin à la réalisation de ce mémoire et
nous avons de la reconnaissance pour tous. Qu'il nous soit permis de remercier
:
Le corps enseignant de l'UFR-SVT, Département Biochimie
Microbiologie de l'Université Joseph KI-ZERBO pour l'enseignement de
qualité comme facteur incontournable d'un développement durable
;
Pr Yves TRAORE, Professeur Titulaire, Directeur du Laboratoire
de Biotechnologie et d'Immunologie Appliquée (LaBIA) pour nous avoir
accueilli dans son Laboratoire et nous avoir offert l'opportunité de
faire nos premiers pas dans la recherche scientifique ;
Dr Cheikna ZONGO, Maitre de Conférences, Directeur de
ce mémoire, pour avoir cru en nous et accepté encadrer nos
travaux de mémoire malgré ses multiples occupations. Il
était toujours là pour répondre à nos nombreuses
sollicitations. Son apport sans mesure dans cette étude, ses suggestions
et remarques pertinentes, sa simplicité lors de nos échanges ont
donné au travail toute sa valeur scientifique, nous avons beaucoup
appris à ses côtés, merci pour tout ;
Dr Fidèle TIENDREBEOGO, Chargé de recherche,
Co-Directeur de ce mémoire pour nous avoir donné l'occasion de
réaliser ce travail fascinant et stimulant. Nous lui sommes
reconnaissant pour la qualité de son encadrement, sa
disponibilité pour répondre à nos préoccupations,
son soutien lors des moments difficiles, ses apports techniques et critiques,
son sens élevé de la rigueur, son goût du travail bien fait
et surtout pour l'attention particulière portée à mon
stage. Merci pour toutes les choses qu'il nous a faites et que nous ne pourrons
pas citer ici, mais qui resteront à jamais gravées dans notre
mémoire ;
Dr Kossikouma Djodji ADJATA, Maitre de Conférences,
Université de Lomé, pour son soutien lors de la prospection et
collecte des échantillons, sa collaboration et sa participation à
la réalisation de notre travail ;
Pr Nicolas BARRO Professeur Titulaire, pour nous avoir fait
l'honneur de présider ce jury ; qu'il trouve ici l'expression de notre
profonde reconnaissance, nos remerciements sincères et l'assurance de
notre profond respect ;
III
Dédicaces, Remerciements, Table des
matières
Pr Aly SAVADOGO, Professeur Titulaire, pour avoir
accepté de juger ce travail et pour l'accompagnement financier qu'il
nous a accordé lors du voyage pour la prospection et la collecte des
échantillons. Qu'il trouve ici, l'expression de nos sincères
reconnaissances.
Dr James Bouma NEYA, Maître de recherche, Directeur du
Laboratoire de Virologie et de Biotechnologies Végétales (LVBV)
et du Laboratoire Mixte International (LMI Patho-Bios), pour nous avoir
accepté au sein de son laboratoire (LVBV) pour nos travaux ;
Dr Drissa SEREME, Maître de recherche et co-Directeur du
Laboratoire de Virologie et de Biotechnologies Végétales (LVBV)
pour nous avoir accepté au sein dudit laboratoire ;
Pr Kofi KOBA, Professeur Titulaire, Université de
Lomé, pour ses conseils et son soutien multiforme lors de la prospection
et collecte des échantillons au Togo. Grâce à son appui,
nous avons pu obtenir des données relatives à la production
globale du gombo au Togo auprès de la Direction des Statistiques
Agricoles, de l'Information et de la Documentation (DSID). Qu'il trouve ici
l'expression de notre profonde reconnaissance, nos remerciements
sincères et l'assurance de notre profond respect.
M. Ézéchiel TIBIRI, doctorant du Laboratoire de
Virologie et de Biotechnologies Végétales (LVBV) et du
Laboratoire Mixte International (LMI Patho-Bios), pour son assistance technique
au laboratoire, et les analyses bioinformatiques de nos séquences. Nous
avons beaucoup appris de lui ; qu'il trouve ici l'expression de notre profonde
gratitude ;
Mlle Monique SORO, Ingénieure de recherche, doctorante
du Laboratoire de Virologie et de Biotechnologies Végétales
(LVBV) et du Laboratoire Mixte International (LMI Patho-Bios) pour son
assistance technique lors de nos travaux. Nous lui sommes vraiment
reconnaissants pour sa disponibilité, sa détermination et ses
apports techniques. Nous lui souhaitons une bonne santé et du courage
pour la suite de ses travaux ;
Dr Moustapha KOALA, pour ses conseils et encouragements.
Messieurs, OUATTARA Bakary, SAWADOGO Moumini, KABORE Emmanuel,
DAKUO Thierry, BOUNIOUNOU Damis Yves Patrik, Mlle DRABO Salimata,
MlleFadila TINTAet Mme ZONGO PawindéÉlisabeth, pour
leur précieux conseils, assistance, bonne humeur, amitié, et ces
moments animés de fraternité tout au long de mon stage ; nous
avons vraiment apprécié votre compagnie ;
Mlle Wendpanga TAPSOBA, Technicienne au Laboratoire de
Virologie et de Biotechnologies Végétales (LVBV) et au
Laboratoire Mixte International (LMI Patho-Bios), pour son assistance technique
tout au long de notre stage ;
iv
Dédicaces, Remerciements, Table des
matières
M. Salissou ATAMAO, Ingénieur Agronome, pour sa
participation active à la collecte des échantillons dans la zone
littorale et pour ses vas et viens à Lomé lors des
formalités liées à la demande de données
statistiques auprès de la Direction des Statistiques Agricoles, de
l'Information et de la Documentation (DSID), merci pour tout ;
Tous les producteurs de gombo de la zone d'étude qui
nous ont accueillis et qui ont contribué à la réalisation
de cette étude. Nous voulons leur dire que sans eux ce travail aurait
été impossible ;
Nos Camarades étudiants, SETOR A. Marius pour ses
soutiens multiformes, SOME G. Fabrice, délégué de notre
promotion, pour son courage et sa détermination, KERE Inès,
DIANDA Ida Georgette et SIMPORE Mamouna avec qui nous avons partagé bien
de meilleurs souvenirs mêmes si nous avions connu des hauts et des bas
par moment. Nous leur formulons tous et toutes nos voeux de réussite
dans leur vie future.
Nous ne saurons terminer ces propos sans dire merci à
tous nos autres promotionnaires des Masters du Département
Biochimie-Microbiologie et particulièrement nos collègues du
Master Biotechnologie Microbienne et Cellulaire (BMC) avec qui nous avons
partagé la vie estudiantine. Qu'ils trouvent ici l'expression de notre
profonde reconnaissance.
Que toutes personnes qui de prêt ou de loin ont
contribué à la réussite de ce travail trouve ici
l'expression de nos sincères remerciements.
V
Dédicaces, Remerciements, Table des
matières
Table des matières
Dédicaces i
Remerciements ii
Liste des tableaux ix
Liste des figures ix
Liste des annexes x
Liste des abréviations xi
Résumé xv
Abstract xvi
Introduction 1
Chapitre I: Synthèse bibliographique 3
I. Généralité sur le gombo 3
1. Le genre Abelmoschus 3
1.1. Historique 3
1.2. Données taxonomiques 3
2. Description de l'espèce Abelmoschus esculentus
(L.) Moench 3
2.1. Origine géographique 3
2.2. Systématiques 4
3. Description botanique 4
4. Croissance et développement 6
5. Exigences pédoclimatiques 6
6. Composition biochimique, usages et valorisations du gombo
6
6.1. Composition biochimique du gombo 6
6.1.1. Composition en éléments organiques et
minéraux 6
6.1.2. Composition en mucilages 8
6.2. Usages et valorisation du gombo 8
6.2.1. Valorisations nutritionnelles et sociales 8
6.2.2. Valorisations industrielles et artisanales 9
6.2.3. Valorisations thérapeutiques 9
7. Production et commercialisation du gombo 9
II. Maladies et insectes ravageurs du gombo 10
1. Maladies causées par les champignons 10
2. Maladies bactériennes 10
3. Maladies virales 11
3.1. Virus responsables de la maladie
de l'enroulement des feuilles du gombo (Okra
Mosaic Desease) 11
vi
Dédicaces, Remerciements, Table des
matières
3.2. Maladie de l'enroulement foliaire du gombo 11
4. Insectes ravageurs 12
III. Famille des Geminiviridae 14
1. Généralité et classification 14
2. Origine des géminivirus 15
3. Le genre Begomovirus 15
3.1. Organisation génomique du genre Begomovirus
15
3.2. Association des Begomovirus avec des satellites
17
3.3. Réplication des Begomovirus 18
3.4. Variabilité génétique des
Begomovirus 20
3.4.1. Mutations 20
3.4.2. Survenue des phénomènes de recombinaisons
20
3.5. Dissémination spatiale et temporelle des
Begomovirus 21
3.5.1. Insecte vecteur 21
3.5.2. Activités humaines 22
3.6. Hôtes et symptômes causés par les
Begomovirus 22
4. Principales méthodes de diagnostics des
Begomovirus 23
4.1. Tests sérologiques 23
4.2. Tests moléculaires 24
5. Méthodes de lutte contre les Begomovirus
24
5.1. Pratiques culturales, prophylaxie 24
5.2. Résistance variétale 25
Chapitre II: Matériel et méthodes 26
I. Matériel d'étude 26
1. Cadre et période de l'étude 26
2. Matériel végétal 27
3. Matériel de laboratoire et de prospection 27
II. Méthodologie 28
1. Collecte des échantillons 28
2. Extraction des ADNs totaux 28
3. Détection des Begomovirus par la PCR 30
3.1. Amorces utilisées 30
3.2. Composition des mix et programmes PCR 31
4. Analyse des produits de la PCR 32
4.1. Préparation du gel 32
4.2. Électrophorèse et visualisation 33
5. Séquençage de certains produits PCR 34
VII
Dédicaces, Remerciements, Table des
matières
6. Analyses statistiques et phylogénétiques 34
6.1. Analyses statistiques 34
6.2. Analyses phylogénétiques 34
Chapitre III: Résultats et discussion 35
I. Résultats 35
1. Symptômes observés dans les parcelles de
production de gombo au Togo 35
2. Évaluation de la prévalence de la maladie de
l'enroulement foliaire du gombo au
Togo 37
3. Caractérisation moléculaire des espèces
virales associées à la maladie de
l'enroulement foliaire du gombo 37
3.1. Amplification du gène de la protéine
capsidiale par la PCR 37
3.2. Amplification des Betasatellites 41
3.3. Séquençage des produits PCR du gène de
la protéine capsidiale 41
4. Diversité des virus responsables de la maladie de
l'enroulement foliaire du gombo au
Togo 42
II. Discussion 44
Conclusion 46
Recommandations 46
Perspectives 47
Références bibliographiques 48
Annexes 63
ix
Listes des Tableaux, Figures et
Annexes
Liste des tableaux
Tableau I : Classification de Abelmoschus esculentus
(L.) Moench 4
Tableau II : Composition biochimique des fruits et des feuilles
de gombo pour 100 g de partie
comestible (matière fraîche) 7
Tableau III : Espèces de Begomovirus responsables
de l'OLCD en Afrique 11
Tableau IV : Répartition des échantillons
collectés suivant les zones agroécologiques et les
localités 28
Tableau V: Liste des amorces utilisées pour les
différentes réactions PCR 30
Tableau VI: Composition du Mix PCR pour la détection des
Begomovirus 31
Tableau VII: Composition du Mix PCR pour la détection des
Betasatellites 31
Tableau VIII: Prévalences de la maladie dans les quatre
zones agroécologiques 37
Tableau IX: Récapitulatif des
résultats PCR par zone agro-écologique et par localité
38
Liste des figures
Figure 1: Plant de gombo 4
Figure 2 : Illustration des différentes parties de la
plante du gombo (Abelmoschus esculentus
(L.) Moench): feuilles (a), fleurs (b), fruits (c), et graines
(d) 5
Figure 3: Principaux insectes ravageurs observés sur le gombo.
(A) Altise (Podagrica decolorata); (B) Mouche blanche (Bemisia
tabaci Genn.); (C) Jasside (Jacobiasca sp); (D)
Puceron (Aphis gossypii); (E) Dysdercus sp ;
(F) Syllepte derogata ; (G) Anomis flava 13
Figure 4 :
(a) Reconstruction tridimensionnelle d'une particule géminivirale.
L'échelle représente 10 nm (Zhang et al., 2001). (b)
Particules géminivirales vues au microscope électronique montrant
leur forme icosaédrique associée en doublets. L'échelle
représente 50
nm. 14
Figure 5: Représentation de l'organisation
génomique des Begomovirus (monopartites et bipartites). LIR
(Long Intergenic Region); SIR (Short Intergenic Region); CR
(Common
Region). 16
Figure6: Représentation des ADN
â satellites et á satellites codant respectivement pour les
protéines ßC1 et Rep, et des molécules défectives
sans ORFs. A-rich (Adenine-rich region);
CR (Common Region); IR (Intergenic Region); SCR (Satellite
Conserved Region) 18
Figure 7: Schématisation des modèles de
réplication en cercle roulant (RCR) et de la
réplication dépendante de la recombinaison (RDR)
des Begomovirus . 19
Figure 8: Bemisia tabaci adulte 21
X
Listes des Tableaux, Figures et
Annexes
Figure 9: Symptôme (a) de la maladie de l'enroulement
foliaire du gombo (Okra leaf curl disease, OLCD) observé au
Burkina Faso, (b) de la maladie du jaunissement des nervures du piment (Pepper
yellow vein disease, PYVD) et (c) de la maladie de l'enroulement (et du
jaunissement) foliaire de la tomate (Tomato yellow leaf curl disease,
ToLCD-TYLCD)
observé au Togo 23
Figure 10: Carte de la zone d'étude montrant les douze
(12) localités prospectées 27
Figure 11: (A) broyeur à bille (Tissuelyser II)
utilisé pour le broyage des échantillons ; (B)
balance électronique utilisée pour peser les
échantillons 30
Figure 12: Thermocycleur 2720 (Applied Bioystems) utilisé
pour l'amplification 32
Figure 13: GEL DOC Major Science (ms) UVDI utilisé pour la
visualisation 33
Figure14: (B) et (D) plants de gombo avec des feuilles
déformées; (C) enroulement des feuilles de gombo sur elles
même; (A) et (F) feuilles de gombo en formes de cuillères; (E)
feuilles de gombo présentant des jaunissements.
36
Figure 15 : Electrophorèse des produits d'amplification des
Betasatellites sur gel d'agarose à 1%. 100 paires de bases = marqueur de
taille DNA ladder. 36 ; 37 ; 39 ; 40 ; 41 ; 54 sont
positifs. 41
Figure16: (A) : Arbre phylogénique
généré à partir des séquences de CP des
CLCuGeV,
OYCrV du Togo et d'autres Begomovirus existant dans le GenBank..
43
Liste des annexes
Annexe 1:Procédure d'extraction des ADNs 63
Annexe 2:Procédure de préparation du gel
et de la migration électrophorétique des ADN
amplifiés par PCR 64
Annexe 3:Préparation des Mix et programmes PCR 64
Annexe 4: Composition de quelques tampons utilisés 66
xi
Liste des Sigles, Abréviations et
Acronymes
Liste des acronymes viraux selon le Comité
International de Taxonomie des Virus (ICTV)
Acronyme Dénomination du virus
ACMBFV: African cassava mosaic Burkina Faso
virus
ACMV : African cassava mosaic virus
CLCuGA : Cotton leaf curl Gezira
alphasatellite
CLCuGB : Cotton leaf curl Gezira
betasatellite
CLCuGV : Cotton leaf curl Gezira
virus
EACMV: East African cassava mosaic
virus
HoLCrV: Hollyhock leaf crumple virus
OLCCMV: Okra leaf curl Cameroon virus
OLCuMLA: Okra leaf curl Mali alphasatellite
OLCuMLB : Okra leaf curl Mali betasatellite
OLCV : Okra leaf curl virus
OYCrCMA : Okra yellow crinkle Cameroon
alphasatellite
OYCrV : Okra yellow crinkle virus
ToLCBFV : Tomato leaf curl Burkina Faso
virus ToLCTGB : Tomato leaf curl Togo
betasatellite
ToLCV: Tomato leaf curl virus
TYLCV: Tomato yellow leaf curl virus
Liste des abréviations
a) Organismes et institutions
CNRST Centre national de la recherche
scientifique et technologique
CREAF Centre de recherches environnementales,
agricoles et de formation
DDBJ DNA Data Bank of Japan
DSID Direction des Statistiques Agricoles, de
l'Information et de la
Documentation
EMBL European Molecular Biology
Laboratory
FAO Organisation des Nations unies pour
l'alimentation et l'agriculture
FAOSTAT Food and Agriculture Organization
Statistics
XII
Liste des Sigles, Abréviations et
Acronymes
GenBank Banque de données
génomiques des États-Unis d'Amérique
ICTV International commitee on taxonomy
of viruses (Comité international de
taxonomie des virus)
INERA Institut de l'environnement et de
recherches agricoles
LMI Laboratoire Mixte International
LVBV Laboratoire de Virologie et
Biotechnologie Végétale
NCBI National Center of Biotechnology
Information
RABIOTECH Réseau ouest-africain de
biotechnologie
UFR/SVT Unité de formation et de
recherche/sciences de la vie et de la terre
UL Université de Lomé
b) Autres abréviations
ADN Acide
Désoxyribonucléique
ADN-A Composant A du génome des
Begomovirus
ADN-B Composant B du génome des
Begomovirus
ADNsb Acide désoxyribonucléique
simple brin
APG Angiosperm Phylogeny Group
ARN Acide Ribonucléique
BLAST Basic Local Alignment Search
Tool
bp Paire de bases
cccDNA Forme super-enroulée de l'ADN
viral
CP Protéine de Capside
CR Common Region
dNTP Désoxynucléotide
triphosphate
DO Densité Optique
EDTA Acide éthylène diamine
tétra acétique
ELISA Enzyme Linked Immuno Sorbent
Assay
Genn. Gennadius
ID Immuno-Diffusion
IR Intergenic Region ou
Région Intergénique
Kb Kilobase
XIII
Liste des Sigles, Abréviations et
Acronymes
LIR Long Intergenic Region
MP Protéine de Mouvement
OLCD Okra leaf curl disease
ORF Open Reading Frames
Ori Origine de réplication
PCR Polymerase Chain Reaction
PIB Produit Intérieur Brut
PIP Programme International de Partenariat
qsp20 Quantité suffisante d'eau pour
compléter un volume de réaction à 20 ìl
RCR Rolling Circle Replication
RDR Recombination Dependent
Replication ou Réplication dépendante
REn Replication
Enhancer ou Protéine activatrice de la
réplication
Rep Protéine de Réplication
rpm rotation par minute
SCR : Satellite Conserved Region
TAE Tris-Acétate-EDTA
TrAp Protéine activatrice de la
transcription
xv
Résumé, Abstract
Résumé
La culture du gombo (Abelmoschus esculentus (L.)
Moench, Malvaceae) offre des produits contribuant à l'équilibre
alimentaire, et à la génération de revenus pour de
nombreux producteurs de beaucoup de pays de l'Afrique de l'Ouest et
particulièrement ceux du Togo. Depuis plusieurs années, la
maladie de l'enroulement foliaire du gombo (Okra leaf curl disease,
OLCD) constitue l'une des maladies les plus dommageables dans plusieurs pays de
l'Afrique de l'Ouest. Elle est occasionnée par des virus du genre
Begomovirus (famille des Geminiviridae) et transmise par la mouche
blanche (Bemisia tabaci Genn.) qui constitue le principal vecteur. En
vue de connaître l'identité moléculaire et la
diversité des Begomovirus responsables de cette maladie sur le
gombo au Togo, des travaux de caractérisation moléculaire ont
été effectués sur 118 échantillons de jeunes
feuilles de gombo (symptomatiques et asymptomatiques) collectés dans les
quatre zones agroécologiques du pays (zone littorale, zone
forestière, savane humide et la savane sèche). Les ADNs totaux
ont été extraits selon le protocole Doyle et Doyle modifié
pour l'extraction d'ADN au Cétyl Trimétyl Ammonium Bromide (CTAB)
et le diagnostic moléculaire a été réalisé
par PCR (Polymerase Chain Reaction) en utilisant un couple d'amorces
dégénérées (VD360/CD1266). Un total de 12
échantillons de produits PCR a été séquencé
et des analyses bioinformatiques ont été réalisées.
Il ressort de ces analyses que les isolats du Togo appartiennent à deux
espèces : le Cotton leaf curl Gezira virus (CLCuGeV) avec une
similitude de 88,71 à 96,56% avec CLCuGeV-Burkina Faso (FN554528),
CLCuGeV-Niger (FJ469627) et le Okra yellow crinkle virus (OYCrV) avec
une similitude de 86,52% à 97,70% avec les OYCrV de la Côte
d'Ivoire (KX100572), du Mali (EU024118) et du Cameroun (FM164724). Cette
étude a permis d'identifier les espèces de Begomovirus
infectant le gombo au Togo. Ces virus constituent un véritable
problème non seulement pour la production du gombo, mais aussi celle du
coton (Gossypium sp. L.) au Togo tout comme dans les autres pays
producteurs de l'Afrique de l'Ouest. Au regard des impacts
socio-économiques négatifs que ces virus pourraient causer, la
mise en oeuvre des stratégies de lutte efficaces s'avère
nécessaire.
Mots clés : Begomovirus,
CLCuGeV, OYCrV, PCR, gombo, Togo
xvi
Résumé, Abstract
Abstract
The cultivation of okra (Abelmoschus esculentus (L.)
Moench, Malvaceae) offers products that contribute to a balanced diet and
income generation for many producers in many West African countries,
particularly those from Togo. For many years, Okra Leaf Curl Disease (OLCD) has
been one of the most damaging diseases in several West African countries. It is
caused by viruses of the genus Begomovirus (Geminiviridae family) and
transmitted by the whitefly (Bemisia tabaci Genn.) which is the main
vector. In order to determine the molecular identity and the diversity of
Begomoviruses responsible for this disease on okra in Togo, molecular
characterization works were carried out on 118 samples of young okra leaves
(symptomatic and asymptomatic) collected from the four agro-ecological zones of
the country (littoral zone, forest zone, wet savannah and dry savannah). Total
DNAs were extracted according to the modified Doyle and Doyle protocol for DNA
extraction with Cetyl Trimethyl Ammonium Bromide (CTAB) and the molecular
diagnosis was carried out by PCR (Polymerase Chain Reaction) using a pair of
degenerate primers (VD360/CD1266). A total of 12 samples of PCR products were
sequenced and bioinformatic analyzes were performed. These analyzes indicate
that the isolates from Togo belong to two species: the Cotton leaf curl Gezira
virus (CLCuGeV) with a similarity of 88.71 to 96.56% with CLCuGeV-Burkina Faso
(FN554528), CLCuGeV -Niger (FJ469627) ) and Okra yellow crinkle virus (OYCrV)
with a similarity of 86.52% to 97.70% with OYCrV from Côte d'Ivoire
(KX100572), Mali (EU024118) and Cameroon (FM164724). This study identified
Begomovirus species infecting okra in Togo. These viruses are a real
problem not only for of okra and cotton (Gossypium sp L.) production
in Togo as in other West African producing countries. In view of the negative
socio-economic impacts that these viruses could cause, the implementation of
effective control strategies is necessary.
Key words: Begomovirus, CLCuGeV, OYCrV,
PCR, okra, Togo
INTRODUCTION
1
Introduction
Introduction
Le gombo (Abelmoschus esculentus (L.) Moench) est un
légume cultivé dans la plupart des pays des régions
tropicales, subtropicales et méditerranéennes d'Afrique, d'Inde
et d'Amérique. Il appartient au genre Abelmoschus et à
la famille des Malvaceae (Spichiger et al., 2000). Le genre
Abelmoschus se répartit en 7 espèces dont A.
angulosus, A. callei, A. crinitus, A. esculentus, A. ficulneus, A. manihot et
A. moschatus (Hamon et Charrier, 1997). Parmi ces espèces de gombo,
4 sont cultivées (A. callei, A. esculentus, A. manihot et A.
moschatus) et les 3 autres (A. angulosus, A. crinitus et A.
ficulneus) sont strictement spontanées (Borssum-Waalkes, 1966).
C'est une plante exceptionnelle et originale, car toutes ses parties (racines,
tiges, feuilles, fruits, graines) sont valorisées sur les plans
alimentaire, médicinal, artisanal et même industriel (Marius
et al., 1997). Les jeunes fruits et feuilles du gombo sont couramment
utilisés comme condiment ou liant dans les sauces (Hamon et Charrier,
1997). Sur le plan nutritionnel, le gombo est riche en calcium, fer,
protéines, glucides, lipides, vitamines A, C et en magnésium
(Charrier, 1983 ; Siemonsma et Hamon, 2004 ; Doumbia, 2010) qui sont des
compléments alimentaires utiles pour des repas à base de
céréales.
En Afrique et particulièrement au Togo, le gombo est
hautement prisé et est le plus souvent utilisé comme condiment ou
liant dans les sauces (Hamon et Charrier, 1997). Il procure également
des revenus très importants aux producteurs en général les
femmes en améliorant leurs conditions de vie. Ainsi, le gombo pourrait
jouer un rôle essentiel dans les programmes d'amélioration de
l'alimentation des populations ainsi que dans les programmes de lutte contre la
pauvreté.
Cependant, la production de cette spéculation fait face
à d'énormes contraintes entrainant des pertes
considérables de rendement dans les pays tropicaux en
général et particulièrement au Togo. Parmi ces
contraintes, figure en bonne place la maladie de l'enroulement foliaire du
gombo encore appelée Okra leaf curl disease (OLCD). Elle est
causée par des virus appartenant au genre Begomovirus (famille
des Geminiviridae) qui sont transmis par la mouche blanche
(Bemisia tabaci Genn.). La maladie induit chez des plants de gombo
infectés des symptômes très caractéristiques qui
vont d'une déformation des feuilles en forme de cuillère à
un enroulement des feuilles sur elles-mêmes (vers le bas ou vers le
haut).
Face à cette situation, d'innombrables travaux de
caractérisation moléculaire ont été
effectués en Afrique dans le but d'identifier les espèces virales
responsables de cette maladie. C'est ainsi que la maladie de l'enroulement
foliaire du gombo a été décrite dans de
2
Introduction
nombreux pays d'Afrique sahélienne et ses environs,
notamment au Burkina Faso (Barro et al., 1994), au Cameroun (Leke
et al., 2013), en Côte d'Ivoire (N'Guessan et al.,
1992), au Ghana (Swanson et Harrison, 1993), au Mali (Kon et al.,
2009), au Niger (Shih et al., 2009), au Nigéria et au Tchad
(Swanson et Harrison, 1993) et en Égypte (Bigarré et
al., 2001). Mais, force est de constater qu'au Togo, la littérature
demeure déficitaire, voire quasi inexistante en informations
nécessaires à un contrôle satisfaisant de ces virus.
Pourtant, des symptômes de repliements foliaires retardant la croissance
et affectant l'appareil végétatif des plants de gombo ont
été observés sur toute l'étendue du territoire. Au
regard de la situation actuelle du Togo face à cette pathologie
dévastatrice, on se pose des questions sur la situation réelle de
cette maladie à Begomovirus au Togo : (i) quelle est
l'identité moléculaire des Begomovirus qui infectent le
gombo au Togo ? (ii) Quels liens existent entre les caractéristiques
moléculaires des Begomovirus du Togo et ceux d'Afrique ?
D'où l'initiation de cette étude dont l'objectif
général est d'évaluer la diversité des
Begomovirus infectant le gombo au Togo. Plus spécifiquement
cette étude vise à (i) déterminer l'identité
moléculaire des principaux Begomovirus infectant le gombo au
Togo et (ii) d'établir la taxonomie de ces Begomovirus
du Togo.
CHAPITRE I : SYNTHÈSE BIBLIOGRAPHIQUE
3
Chapitre I : synthèse bibliographique
Chapitre I: Synthèse bibliographique
I. Généralité sur le gombo
1. Le genre Abelmoschus 1.1.
Historique
Les espèces de gombo cultivées et celles
sauvages apparentées ont été initialement classées
dans le genre Hibiscus, section Abelmoschus par Linné
(1737). Medikus (1787) a proposé d'élever cette section au rang
d'un genre distinct, mais la référence au genre
Hibiscus
est restée jusqu'au milieu du 20e
siècle. Il a fallu attendre la réhabilitation du genre
Abelmoschus par Hochreutiner (1924) pour que son emploi soit admis
dans les flores et la littérature contemporaine. Ce genre se distingue
du genre Hibiscus d'après les caractéristiques du
calice: calice spatiforme à cinq dents courtes, soudées à
la corolle et caduques après la floraison (Terrell et Winters, 1974).
1.2. Données taxonomiques
Le genre Abelmoschus appartient à la famille
des Malvacées. Il comprend environ 1500 espèces. C'est une
famille très facile à reconnaître par sa fleur qui a un
aspect typique dû à ses pétales à
préfloraison tordue et aux nombreuses étamines soudées en
un tube (Guignard, 1993). Il se distingue par les caractéristiques du
calice. Le genre Abelmoschus est constitué d'une série
polyploïde dont l'organisation n'est pas aisée à saisir. On
peut cependant distinguer trois niveaux de ploïdie. Un premier ensemble
d'espèces possède des nombres chromosomiques de bases compris
entre 2n=58 et 2n=78 chromosomes. Il s'agit d'Abelmoschus tubernaculatus,
Abelmoschus manihot, Abelmoschus moschatus, Hibiscus coccineus, Hibiscus
grandiflorus et Abelmoschus ficulneus. Le deuxième niveau
comprend les polyploïdes issus directement des génomes de base (2n
= 120 à 140) : ce sont Abelmoschus esculentus, Abelmoschus
tetraphyllus et Abelmoschus pungens. Le dernier niveau comprend
les gombos de type "Guinéen" d'Afrique occidentale à 2n=192 ou
194 chromosomes (Charrier, 1983).
2. Description de l'espèce Abelmoschus
esculentus (L.) Moench 2.1. Origine géographique
Le gombo (Abelmoschus esculentus (L). Moench) est une
plante cultivée d'origine controversée. En effet, si l'origine du
genre Abelmoschus ne souffre d'aucun débat, deux
hypothèses s'affrontent quant à l'origine géographique
d'Abelmoschus esculentus (L.) Moench
Chapitre I : synthèse bibliographique
(Ouedraogo, 2009). Certains auteurs soutenant que l'un de ses
ancêtres (Abelmoschus tuberculatus L.) est natif de « Uttar
» Pradesh (Nord de l'Inde) suggèrent que l'espèce
Abelmoschus esculentus (L.) Moench est originaire de cette aire
géographique (Hamon et Sloten, 1995). Alors que d'autres, sur la base de
sa culture antique en Afrique orientale et la présence de l'autre
ancêtre (Abelmoschus ficulneus), suggèrent que l'aire de
domestication d'Abelmoschus esculentus (L.) Moench est le nord de
l'Égypte ou l'Éthiopie. Cependant, aucune preuve
définitive n'est disponible aujourd'hui.
2.2. Systématiques
L'espèce Abelmoschus esculentus (L.) Moench
appartient à la famille des Malvacées (Hamon et Charrier, 1983)
qui comporte environ 1500 espèces surtout intertropicales. La
systématique de l'espèce est consignée dans le tableau
I
Tableau I : Classification de Abelmoschus
esculentus (L.) Moench

Règne Plantae
Sous-règne Tracheobionta Division Magnoliophyta
Classe Magnoliopsida
Sous-classe Dilleniidae
Ordre Malvales
Famille Malvaceae
Genre Abelmoschus
Espèce Abelmoschus esculentus (L.) Moench
4
Source : Classification
phylogénétique APG II, 2003 (
https://www.tela-botanica.org/2009/11/article3354/)
|
3. Description botanique
|
Figure 1 : Plant de gombo (Photo Akakpo,
2018)
Figure 1: Pant de gombo (Photo AKAKPO 2018
|
Le gombo (Abelmoschus esculentus (L.) Moench) est une
plante herbacée annuelle, à port érigé, de 1
à 4 m de haut. Il est généralement peu ramifié,
à tige cylindrique, avec des poils raides disséminés,
souvent tachetés de rouge (Rohwer, 2002 ; Grubben, 2004).
Les feuilles (Figure 1a) du gombo sont simples,
alternées, de forme et de taille variables. Ses stipules sont filiformes
et mesurent jusqu'à 2 cm de long. Ils sont souvent fendus jusqu'à
la base et couverts de poils raides. Le pétiole mesure 5 à 50 cm
de long (Rohwer, 2002).
5
Chapitre I : synthèse bibliographique
Les fleurs (Figure 1b) sont axillaires, solitaires ou en
grappe. Le pédicelle mesure jusqu'à 3 cm de long sur la fleur et
7 cm sur le fruit, avec des poils raides disséminés ; le calice
mesure 2-6 cm de long, avec 5 dents à l'apex et se fendant
généralement sur un côté lors de l'expansion de la
corolle ; la fleur est formée de 5 pétales libres de 3-7 cm de
long. (Siemonsma, 1982).
Le fruit (Figure 1c) est une capsule érigée en
section ronde de 5 à 10 angles, concave entre les côtes, variant
quand il est jeune d'une couleur rouge-violet et vert rougeâtre à
vert foncé. Il peut, contenir jusqu'à 100 graines à la
maturité (Siemonsma, 1982).
Les graines (figure 1d) de forme globuleuse à
ovoïde mesurent 3-6 mm de diamètre (Siemonsma, 1982).

Figure 2 : Illustration des différentes
parties de la plante du gombo (Abelmoschus esculentus (L.) Moench):
feuilles (a), fleurs (b), fruits (c), et graines (d) (photo Akakpo, 2018)
6
Chapitre I : synthèse bibliographique
4. Croissance et développement
Le cycle du gombo varie de 3 mois pour les
variétés les plus précoces à un (1) an et parfois
plus pour les plus tardives (Koechlin, 1989). La multiplication se fait par
graine. La germination a généralement lieu au bout d'une semaine.
Selon la variété et les conditions climatiques, la floraison se
produit un à deux mois après semis. Elle est continue dans le
temps. C'est une plante auto compatible et susceptible de fécondation
croisée par des insectes pollinisateurs à un taux qui peut
atteindre 20% (Charrier, 1983). Après la fécondation, la
croissance du jeune fruit est rapide. L'ovaire de moins de 2 cm donne en trois
jours un fruit de plus de 5 cm de long. La croissance est ralentie par la suite
(Hamon, 1987). Pour l'utilisation en légumes, les jeunes fruits sont
cueillis environ une semaine après la floraison (Siemonsma et Hamon,
2004).
5. Exigences pédoclimatiques
Le gombo est une espèce bien adaptée aux
climats chauds et humides. Il est sensible à la sécheresse, mais
cette sensibilité varie suivant les phases du cycle. L'effet du stress
hydrique en phase de boutonnisation est très néfaste pour le
gombo et se manifeste par une baisse considérable des composantes du
rendement (Sawadogo, 2006). Le gombo ne supporte pas des températures
nocturnes trop basses. Il nécessite des températures
supérieures à 20°C pour avoir une croissance normale
(Grubben, 2004). L'initiation florale et la floraison sont retardées
à mesure que la température s'élève (De Lannoy,
2001). Par contre, les meilleurs rendements sont obtenus en période
chaude (Nana, 2005). Le taux de germination et la rapidité de
levée des semis sont optimaux à 25°C selon une étude
réalisée par Bendkhil et Denden (2010). En outre une basse
température de 10°C a entraîné une entrée en
dormance de la graine. L'optimum pour la croissance est de 26-28°C. Le
gombo tolère une grande diversité de sols. Cependant, il
préfère les sols profonds, limon sableux, bien drainés et
riches en matières organiques. Le pH optimal pour la culture du gombo
varie de 6,2 à 6,5 (De Lannoy, 2001 ; Siemonsma et Hamon, 2004 ; Lim et
Chai, 2007).
6. Composition biochimique, usages et valorisations du
gombo
6.1. Composition biochimique du gombo
6.1.1. Composition en éléments organiques
et minéraux
Le gombo est un légume qui procure un apport
intéressant d'antioxydants, de vitamines et de minéraux (Hamon,
1988). La composition biochimique des fruits et des feuilles de gombo pour 100
g de partie comestible est présentée dans le tableau II.
7
Chapitre I : synthèse bibliographique
Tableau II : Composition biochimique des fruits
et des feuilles de gombo pour 100 g de partie comestible (matière
fraîche)
Éléments chimiques
Quantité
Lutéine Zéaxanthine 5,16 mg
Bêta-carotène 2,25 mg
Phosphore 63 mg
Cuivre 0,094 mg
Calcium 81 mg
Manganèse 0,990 mg
Magnésium 57 mg
Zinc 0,6 mg
Sélénium 0,7 mg
Fer 0.8 mg
Potassium 303 mg
Sodium 8 mg
Vitamine K 5,3 mg
Vitamine E 375 UI
Vitamine C 21,1 mg
Thiamine (Vitamine B1) 0.2 mg
Riboflavine (Vitamine B2) 0.06 mg
Pyridoxine (Vitamine B6) 0,215 mg
Acide pantothénique (Vitamine B5) 0,245 mg
Niacine (Vitamine B3) 1 mg
Folates (Vitamine B9) 8,8 mg
Lipides 0,1 mg
Fibre diététique 9%
Protéines 2 g
Glucides 7,03 g
Énergie 1,50%
Source :
http://externe-vip.com/fr/article/voila-ce-qui-arrive-qd
vous mangez régulièrement du gombo
8
Chapitre I : synthèse bibliographique
6.1.2. Composition en mucilages
Les mucilages confèrent au gombo son aspect gluant qui
est une caractéristique de ce fruit-légume. Les mucilages
d'origine végétale sont un complexe polysaccharidique
composé de galactose, de rhamnose et d'acide galacturonique (Hirose
et al., 2004). Ils sont localisés dans les cellules
épidermiques, le xylème primaire et le cortex
périphérique (Clifford et al., 2001). Ils sont aussi
présents dans le style des fleurs et à l'extrémité
des racines (Macquet, 2007). Les fruits sont riches en glucides (7 à 8 %
de la matière sèche) présents sous forme de mucilages
(Sawadogo, 2006).
6.2. Usages et valorisation du gombo
Le gombo est une plante exceptionnelle et originale, car
presque toutes ses parties (racines, tige, feuilles, fruits et graines) sont
valorisées sur le plan alimentaire, médicinal, artisanal et
industriel (Marius et al., 1997).
6.2.1. Valorisations nutritionnelles et sociales
Les jeunes fruits immatures de gombo constituent un
légume important, consommé comme condiment ou liant dans les
sauces (Hamon et Charrier, 1997). Ils sont riches en glucides,
protéines, vitamines K, E, C, B9, fer, phosphore, potassium et
magnésium (Leung et al., 1968 ; Hamon, 1988, Koechlin, 1989 ;
Nzikou et al., 2006). Sa valeur nutritionnelle dépasse celle de
la tomate (Hamon et Charrier, 1997). Selon Kumar et al., (2010), les
graines de gombo pourraient servir à fabriquer une huile
végétale de bonne qualité ainsi que de la farine (en vue
de supplémenter une farine de céréales). Après le
pressage des graines, le tourteau contient 30% de protéines (Marius
et al., 1997). Les graines torréfiées sont
employées dans certaines régions du Nigeria comme substitut du
café (Siemonsma et Hamon, 2004). Les feuilles sont une excellente source
de calcium et contiennent plus de 25% de protéines à
l'état sec (Kennedy, 2012). Consommées comme salade, les feuilles
fraîches ou séchées sont très intéressantes
pour épaissir des soupes et ragoûts (Koechlin, 1989 ; Kennedy,
2012).
Sur le plan social, le gombo joue aussi un rôle
important dans la vie d'une grande proportion des familles pauvres en
améliorant leur repas quotidien, grâce à sa capacité
d'épaissir les soupes, sa diversité de préparation ainsi
qu'à sa valeur nutritive (Tshomba et al., 2015).
9
Chapitre I : synthèse bibliographique
6.2.2. Valorisations industrielles et artisanales
La tige, constituée de fibres (Igor et al.,
2009) sert à l'industrie textile, à la fabrication de papier, de
carton, des cordes, des sacs, des lignes de pêche et de piège
à gibier (Martin, 1982 ; Marius et al., 1997 ;Shamsul et
Arifuzzaman, 2007). Le gombo (Abelmoschus esculentus (L.) Moench)
présente donc un certain potentiel industriel. La possibilité
d'utiliser les graines pour fabriquer du biodiesel a été
prouvée (Anwar et al., 2010).
6.2.3. Valorisations thérapeutiques
Le gombo possède de nombreuses vertus
thérapeutiques. Il est utilisé pour le traitement de maux de
dents (Lapointe, 1987), de la constipation (Nacoulma, 1996), et de la
glycémie (Oyen et Lemmens, 2002). Pour Mugnier, (2008), toutes les
parties de la plante sont utilisées, principalement pour traiter les
maladies cardio-vasculaires ou digestives. Les feuilles sont parfois
utilisées comme base de cataplasmes, comme émollient, sudorifique
ou antiscorbutique et pour traiter la dysurie (Siemonsma et Hamon, 2004). Les
jeunes fruits contiennent un mucilage ayant des propriétés
variées de substitut de plasma sanguin, fluidifiant des systèmes
liquides et sanguins (Martin et al., 1981 ; Marius et al.,
1997). En plus les mucilages du gombo peuvent potentiellement être
utilisés en médecine, ou comme additif alimentaire (Kumar et
al., 2010). L'extrait végétal de gombo présente des
propriétés médicinales dont les molécules ont des
effets immuno modulateurs (Sheu et Lai, 2012). Les nanoparticules pourraient
avoir un grand potentiel pharmaceutique grâce à leur
activité antifongique (Jayaseelan et al., 2013).
7. Production et commercialisation du gombo
La production mondiale du gombo était estimée
à 9 641 284 de tonnes environ en 2017, répartie dans 43 pays sur
une superficie légèrement supérieure à 1,1 million
d'hectares (FAOSTAT, 2019). Les pays en développement d'Asie et
d'Afrique concentrent environ 99% de la production mondiale du gombo (FAOSTAT,
2019).
En Afrique, sa production est caractérisée par
une très faible productivité (2,7 tonnes/ha) par rapport à
la moyenne mondiale de 10 tonnes/ha (FAOSTAT, 2019). Les cinq premiers pays
producteurs en 2017 ont été: l'Inde (6,35 millions de tonnes), le
Nigeria (1,1 million de tonnes), l'Iraq (142 mille tonnes), la Côte
d'Ivoire (139 mille tonnes) et le Pakistan (108 mille tonnes). Au Togo, selon
la Direction des Statistiques Agricoles, de l'Informatique et de la
Documentation (DSID), la production annuelle du gombo s'élevait à
1279,66 tonnes en 2016.
10
Chapitre I : synthèse bibliographique
II. Maladies et insectes ravageurs du gombo 1. Maladies
causées par les champignons
Selon le Programme PIP (2008), les principales maladies
fongiques qui s'attaquent au gombo sont :
V' Le blanc
Les symptômes sont des taches blanches, poudreuses,
surtout sur la face inférieure des feuilles. La progression est rapide,
et entraine le dessèchement des vieilles feuilles. Les conditions
favorables à ce champignon sont : temps assez chaud (24 à
30°C), sans pluie avec des taux d'humidité relative
modérés entre 50 et 90% (PIP, 2008).
V' La cercosporiose noire
Elle se manifeste par la présence de taches foliaires
chlorotiques diffuses, de forme et dimension variables. À la face
inférieure des feuilles, on trouve un amas pulvérulent brun
noirâtre. La maladie progresse des feuilles du bas de la plante vers le
haut de la plante. Les taches peuvent envahir tout le limbe. Les feuilles se
dessèchent et tombent. La saison chaude et humide est la période
favorable (PIP, 2008).
V' La fusariose
Elle peut intervenir à tous les stades de croissance du
gombo, mais surtout au stade préfloraison. Elle provoque le
flétrissement de la plante qui peut finir par mourir (PIP, 2008).
2. Maladies bactériennes
La bactériose est la principale maladie
bactérienne du gombo. La plante est sensible à cette maladie
à n'importe quel stade de son développement. Cette maladie est
occasionnée par Xanthomonas axonopodis. Elle se manifeste par
des petites taches circulaires sur les cotylédons, des fontes de semis
et des pertes de jeunes plants et des taches vitreuses et circulaires sur le
fruit (PIP, 2008).
11
Chapitre I : synthèse bibliographique
3. Maladies virales
3.1. Virus responsables de la maladie de l'enroulement des
feuilles du gombo (Okra Mosaic Desease)
Les travaux de caractérisation moléculaire ont
permis de décrire plusieurs espèces de virus du genre
Begomovirus responsables de la maladie de l'enroulement foliaire du
gombo en Afrique. Ces virus sont résumés dans le tableau III.
Tableau III : Espèces de Begomovirus
responsables de l'OLCD en Afrique
Virus Pays Références
Okra leaf curl virus (OLCV) Cameroun Leke et
al., 2007
|
Cotton leaf curl Gezira virus (CLCuGV)
|
Soudan Idris et Brown, 2000
Niger Shih et al., 2009
Côte d'Ivoire Séka et al., 2016
Burkina Faso Tiendrébéogo et al., 2010
|
|
Okra yellow crinkle virus (OYCrV)
|
Shih et al., 2007 ; Kon et al.,
Mali 2009
|
Côte d'Ivoire Séka et al., 2016
Égypte Bigarré et al., 2001
Hollyhock leaf crumple virus (HoLCrV)
Soudan Idris et Brown, 2002
3.2. Maladie de l'enroulement foliaire du gombo
La maladie de l'enroulement foliaire du gombo ou Okra leaf
curl disease (OLCD) est une maladie virale du gombo occasionnée par
des virus du genre Begomovirus. Les plantes atteintes
présentent un retard de croissance avec un repliement des feuilles (vers
le haut ou vers le bas) et un épaississement de nervures associés
ou non à un jaunissement. Ce qui affecte significativement la
capacité de la plante à produire. Le Okra leaf curl disease
(OLCD) est répandue dans toute la région tropicale et
constitue une contrainte majeure pour la production du gombo parce qu'il
occasionne des pertes considérables de rendement (Barro, 1994 ;
Tiendrébéogo, 2010). Les études entreprises ces
dernières années ont permis de décrire cette maladie sur
le gombo dans de nombreux pays d'Afrique sahélienne et ses environs: en
Côte d'Ivoire (N'Guessan et al., 1992 ; Séka et
al., 2016), au Ghana, au Nigéria et au Tchad (Swanson et Harrison,
1993), en Égypte (Bigarré et al., 2001), au Mali (Kon
et al., 2009), au
12
Chapitre I : synthèse bibliographique
Niger (Shih et al., 2009), au Burkina Faso
(Tiendrébéogo et al., 2010), au Cameroun (Leke et
al., 2013).
4. Insectes ravageurs
Le gombo en comparaison avec la plupart des plantes de la
famille des Malvaceae est considéré comme une plante
entomologique par excellence compte tenu du nombre d'espèces d'insectes
qui l'attaquent (De Lannoy, 2001; PIP, 2008). On rencontre:
V' Les altises (Nisotra spp.)
Les altises sont de petits coléoptères d'environ
3 mm de long (figure 2, A). Les oeufs sont pondus aux pieds des plantes
à proximité du sol. Les larves se nourrissent des racines de la
plante. Les dommages sont négligeables sur les racines. Les
dégâts sont causés par les adultes qui rongent les feuilles
et laissent généralement des petits trous ronds donnant une
apparence criblée aux plantes. Les dégâts sont surtout
dommageables sur les jeunes plants. Les conditions favorables à cet
insecte sont: les sols légers, l'excès d'azote, les plantes
chétives, le sol pauvre en matière organique et un fort
ensoleillement (De Lannoy, 2001).
V' La punaise du cotonnier (Dysdercus
spp.)
Elle se reconnait par le dessèchement et la
tombée des jeunes fruits, des taches sur les fruits à
maturité avancée et parfois leur dessèchement (figure 2,
E). Elle apparait de la floraison jusqu'à la récolte (De Lannoy,
2001).
V' Le puceron du cotonnier (Aphis
gossypii)
Ce puceron a une coloration variable, jaune, vert ou brun. De
forme ailée, elle a la tête et le thorax noir, et l'abdomen vert
à jaune (figure 2, D). Ce puceron suceur affaiblit la plante et provoque
une déformation des feuilles quand ils sont en grand nombre. Ils
produisent un miellat sur lequel un champignon saprophyte peut se
développer. Ce champignon empêche une bonne photosynthèse
(De Lannoy, 2001).
V' Les cicadelles ou jassides
C'est une cicadelle dont l'adulte fait 2,5 à 3 mm de
long, de couleur vert clair à vert jaunâtre (figure 2, C). Pendant
la journée, elles sont sous les feuilles, mais passent sur le dessus des
feuilles le soir. Les nymphes et les adultes sucent la sève des feuilles
en sécrétant une salive toxique. Cela entraîne une
décoloration des feuilles, d'abord aux bords puis aux
13
Chapitre I : synthèse bibliographique
tissus inter-nervaires. Une couleur brunâtre et
rougeâtre peut ensuite apparaître. En cas de forte infestation, la
croissance de la plante peut être bloquée (De Lannoy, 2001).
? La mouche blanche (Bemisia tabaci
Genn.)
C'est un aleurode extrêmement polyphage rencontré
dans les zones de production agricole des cultures maraîchères en
général et particulièrement celles du gombo. Les adultes
de l'insecte mesurent entre 0,8 et 1 mm de long (figure 2, B). Tous les stades
du développement du gombo sont attaqués par les mouches blanches.
Par ailleurs, les attaques précoces par des individus porteurs de virus
constituent de véritables dangers pour les plantules qui cessent de
croître, compromettant ainsi la culture toute entière (PIP,
2008).
? Les chenilles enrouleuses (Syllepte
derogata)
Les chenilles sont vert translucide à pattes et
tête noires (figure 2, F). Les feuilles attaquées sont
découpées et enroulées. Ces chenilles sont le plus souvent
cachées dans les feuilles enroulées. Si les attaques sont fortes,
la croissance de la plante peut être freinée. Les
dégâts sont généralement limités à
quelques plantes dans un champ (De Lannoy, 2001).

Figure 3: Principaux insectes ravageurs
observés sur le gombo. (A) Altise (Podagrica decolorata); (B)
Mouche blanche (Bemisia tabaci Genn.); (C) Jasside (Jacobiasca
sp); (D) Puceron (Aphis gossypii); (E) Dysdercus sp ;
(F) Syllepte derogata ; (G) Anomis flava (Ekra, 2010)
14
Chapitre I : synthèse bibliographique
III. Famille des Geminiviridae 1.
Généralité et classification
Les Begomovirus appartiennent à la famille des
Geminiviridae. Du latin « geminus » qui signifie
« jumeau ». Cette famille comprend des virus dont le
génome est constitué d'une molécule (monopartite) ou de
deux molécules (bipartites) d'ADN simple brin (ADNsb) de tailles
identique (environ 2,8 kilobases). Les molécules d'ADNsb sont
protégées par une capside de forme icosaédrique, en
doublet d'une taille d'environ 18-20 nm de diamètre et 30 nm de long
(figure 3, Zhang et al., 2001; Jeske, 2009).
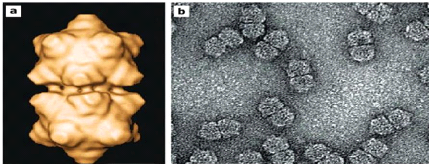
Figure 4 : (a) Reconstruction
tridimensionnelle d'une particule géminivirale. L'échelle
représente 10 nm (Zhang et al., 2001). (b) Particules
géminivirales vues au microscope électronique montrant leur forme
icosaédrique associée en doublets. L'échelle
représente 50 nm (Brown et al., 2011).
Les virus appartenant à cette famille avaient
été classés en quatre genres en se basant sur leur gamme
d'hôtes, leur insecte vecteur et leur organisation génomique
(Brown et al., 2011). Il s'agissait : des virus du genre
Begomovirus qui infectent principalement les plantes
dicotylédones et transmis exclusivement par l'aleurode Bemisia
tabaci Genn., ceux du genre Curtovirus infectant des
plantes dicotylédones et transmis par des cicadelles, ceux du genre
Mastrevirus, infectant principalement des plantes
monocotylédones et transmis par des cicadelles, et enfin ceux du genre
Topocuvirus infectant des plantes dicotylédones et transmis par
un membracide (Rojas et al., 2005). Le genre Begomovirus tire
son nom du Bean golden
mosaic virus responsable de la
mosaïque dorée du haricot (Regenmortel et al., 2000). Ce
genre comporte le plus grand nombre d'espèces décrites, avec plus
de 388 espèces reconnues
15
Chapitre I : synthèse bibliographique
par l'ICTV (Brown et al., 2015). Il est suivi du
genre Mastrevirus avec plus de 30 espèces reconnues
(Zerbiniet al., 2017). Selon leur répartition
géographique, il faut distinguer les Begomovirus de l'Ancien
Monde regroupant des Begomovirus originaires d'Europe, d'Inde,
d'Afrique et d'Asie et les Begomovirus du Nouveau Monde originaires
d'Amérique. Grâce aux nouvelles techniques de
caractérisation moléculaire développées ces
dernières années, 5 nouveaux genres ont vu le jour. Il s'agit:
des Becurtovirus, des Eragrosvirus, des Turncurtovirus
(Varsani et al., 2014) puis des Capulavirus, et des
Grablovirus (Varsani et al., 2017). En outre, quatre autres
espèces de virus étroitement liés aux géminivirus
ont été découvertes et acceptées par le
Comité exécutif de l'ICTV pour inclusion dans la famille des
Geminiviridae. Mais ces espèces resteront non attribuées
à un genre, en attendant l'identification de leurs espèces
vectrices et la confirmation de la morphologie de la particule virale (Varsani
et al., 2017). Il s'agit des espèces Apple geminivirus
(Liang et al., 2015), Citrus chlorotic dwarf associated virus
(Loconsoleet al., 2012; Guo et al., 2015), Grapevine
geminivirus A (Al Rwahnih et al., 2017), Mulberry mosaic
dwarf associated virus (Lu et al., 2015; Ma et al.,
2015) et Tomato associated geminivirus 1 (Fontenele et al.,
2017).
2. Origine des géminivirus
Les géminivirus actuels auraient évolué
à partir de réplicons d'ADN extra-chromosomiques présents
chez les ancêtres procaryotes ou eucaryotes primitifs des plantes
modernes (Koonin et Ilyina, 1992; Rojas et al., 2005). Dans ce
scénario, le géminivirus ancestral est envisagé sous la
forme d'un plasmide extra-chromosomique circulaire d'ADN sb qui se serait
répliqué par un mécanisme de cercle roulant impliquant une
forme réplicative d'ADN db. Les relations phylogénétiques
étroites des protéines Rep des géminivirus avec celles des
protéines Rep des plasmides des phytoplasmes, ainsi que la ressemblance
structurelle forte entre la CP des géminivirus et celle des virus
icosaédriques à ARNsb ont permis de proposer un deuxième
scénario évolutif dans lequel l'acquisition du gène codant
pour la protéine de capside d'un virus de plante à ARNsb par un
plasmide de phytoplasme a donné naissance à l'ancêtre des
géminivirus (Krupovic et al., 2009).
3. Le genre Begomovirus
3.1. Organisation génomique du genre Begomovirus
Selon l'organisation génomique, les Begomovirus
ont été scindés en deux groupes: les Begomovirus
monopartites ayant un ADN simple brin circulaire d'environ 2,8 kilobases
(ADN-A) et les Begomovirus bipartites ayant deux ADN simples brins
circulaires d'environ 2,8 kilobases chacun (ADN-A et ADN-B ; Jeske, 2009 ;
ICTV, 2012). Chacun des deux
16
Chapitre I : synthèse bibliographique
composants (ADN-A et ADN-B) possède des gènes
qui codent pour plusieurs protéines et une région
intergénique (Intergenic Region, IR) d'environ 200 paires de bases
comprenant l'origine de réplication (ori), les itérons (courtes
séquences répétées), la région commune
(Common Region, CR) et la tige boucle (5' TAATATT?AC 3') (Brown et
al., 2011; Zerbini et al., 2017) (figure 5).
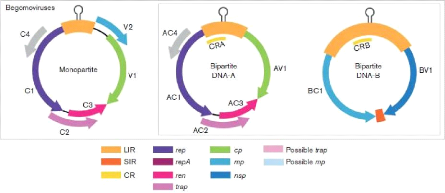
Figure 5: Représentation de
l'organisation génomique des Begomovirus (monopartites et
bipartites). LIR (Long Intergenic Region); SIR (Short Intergenic
Region); CR (Common Region) (Zerbini et al., 2017).
L'ADN-A porte une région intergénique (IR) et 6
cadres ouverts de lecture ou ORFs (Open Reading Frames) dont certains
sont chevauchants (Brown et al., 2011; Fondong, 2013). Sur le brin
viral, l'ORF V1 (AV1 chez les bipartites) code pour la Protéine de
Capside CP qui est l'unité de base dans la constitution de la particule
virale en doublet des Begomovirus. L'ORF V2 (AV2 chez les bipartites)
code pour la Protéine de Mouvement MP qui associé à la CP
permet de diffusion du virus dans la plante hôte (Brown et al.,
2011; Fondong, 2013). Par ailleurs, des études ont montré que les
Begomovirus bipartites du Nouveau Monde ne possèdent pas l'ORF
AV2 (Rojas et al., 2001; Bull et al., 2007; Fondong, 2013;
Hanley-Bowdoin et al., 2013). Les ORFs portés par le brin
complémentaire codent pour la protéine associée à
la réplication (Rep) du virus au sein de la plante (ORF C1), la
protéine activatrice de la transcription (TrAP) (ORF ) (Wezel et
al., 2001), la protéine activatrice de la réplication (REn)
(ORF C3) et la protéine C4 (ORF C4). La protéine C4 semble
intervenir dans l'expression de la sévérité des
symptômes et dans le déplacement du virus dans la gamme
d'hôtes (Krake et al., 1998; Vanitharani et al., 2005).
Associé à la REn, la Rep permet
17
Chapitre I : synthèse bibliographique
l'accumulation de la charge virale dans les cellules de la
plante (Hanley-bowdoin et al., 1989).
L'ADN-B des Begomovirus bipartites porte deux ORFs
non chevauchants qui codent pour deux protéines. La protéine MP
(ORF BC1) qui est impliquée dans le mouvement de cellule à
cellule de l'ADN viral et la protéine NSP (Nuclear Shuttle
Protein, ORF BV1) qui est impliquée dans le transport de l'ADN
viral du noyau vers le cytoplasme à travers la membrane nucléaire
(Sanderfoot et al., 1996; Gafni et Epel, 2002).
3.2. Association des Begomovirus avec des satellites
Les Begomovirus monopartites de l'Ancien Monde ont
été associés à la description de trois types de
molécules d'ADNsb circulaires. Il s'agit des á satellites et des
f3 satellites d'une taille d'environ 1350 nucléotides chacun (Briddon et
Stanley, 2006; Zhou, 2013) et les satellites défectifs d'une taille
d'environ 682 nucléotides (Lozano et al., 2016 ; Zhou,
2013).
Les á satellites sont capables d'autoréplication
dans les cellules végétales, mais dépendent de leurs
Begomovirus assistants, également nommés «
helper », pour le mouvement au sein de la plante hôte et la
transmission inter-plantes via l'insecte vecteur (Mansoor et al.,
1999; Saunders et Stanley, 1999). Ils codent pour une seule protéine
associée à la réplication (Zhou, 2013). Ils semblent jouer
un rôle de modulation de la virulence du complexe Begomovirus-f3
satellite (Idris et al., 2011) en diminuant, voire en supprimant
l'expression des symptômes de certaines maladies à
Begomovirus.
Les f3 satellites quant à eux sont entièrement
dépendants de leur association avec les Begomovirus pour leur
réplication, leur encapsidation et leur dissémination. Ils
contiennent trois régions conservées : une séquence riche
en Adénine (A-richsequence), une séquence
conservée connue sous le nom de région conservée des
satellites (satellite conserved region, SCR) et un seul gène
dans le sens complémentaire qui code pour une petite protéine
(~118 acides aminés) connue sous le nom de f3C1. La protéine f3C1
est une protéine multifonctionnelle intervenant dans la
sévérité de la maladie et la suppression de certains
mécanismes de résistance de la plante.
Les satellites défectifs (deltasatellites),
troisième classe d'ADN satellites en association avec des
Begomovirus, ont récemment été définis
(Fiallo-Olivé et al., 2012; Zhou, 2013; Lozano et al.,
2016). Ils sont distincts des á satellites et des f3 satellites en
raison de la petite taille de leur génome (~ 682 nucléotides) et
de l'absence d'ORF.
18
Chapitre I : synthèse bibliographique
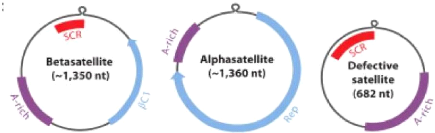
Figure6: Représentation des ADN
â satellites et á satellites codant respectivement pour les
protéines ßC1 et Rep, et des molécules défectives
sans ORFs. A-rich (Adenine-rich region); CR (Common Region); IR (Intergenic
Region); SCR (Satellite Conserved Region) (Zhou, 2013).
3.3. Réplication des Begomovirus
Les Begomovirus utilisent deux mécanismes pour
la réplication de leur génome. Une fois dans le cytoplasme
cellulaire de la plante hôte, l'ADN simple brin viral,
décapsidé, rejoint le noyau pour être converti en forme
réplicative ADN double brin (ADNdb) par la polymérase cellulaire
de l'hôte. L'ADN double brin est capable de se répliquer par un
mécanisme de cercle roulant (RCR), de manière analogue aux
plasmides bactériens et phages à ADN sb circulaire, et par un
mécanisme de réplication dépendante de la recombinaison
(RDR) (Hanley-Bowdoin et al., 1999; Jeske et al., 2001;
Preiss et Jeske, 2003 ; figure 6). La protéine Rep seule est suffisante
à l'initiation de la réplication RCR en introduisant une
ouverture au sein du motif conservé de la tige boucle (TAATATT?AC)
contenant le site d'initiation (?) de la RCR (Hanley-Bowdoinet al.,
2000). Ainsi, elle dérégule le cycle cellulaire et fournit les
facteurs cellulaires compatibles avec la réplication de l'ADN. Une
dernière étape permet le transport des génomes filles
d'ADNsb vers les cellules adjacentes via les plasmodesmes, ou leur
encapsidation pour former de nouvelles particules virales (Gutierrez, 1999).
Chapitre I : synthèse bibliographique

19
Figure7: Schématisation des
modèles de réplication en cercle roulant (RCR) et de la
réplication dépendante de la recombinaison (RDR) des
Begomovirus (Bernardi et Timchenko, 2008 ; Jeske et al.,
2001).
Synthèse de la forme réplicative ADN db
: Après décapsidation, il y a la
synthèse d'une amorce complémentaire à la région
intergénique JR. La synthèse du brin complémentaire est
initiée à partir de cette amorce. L'ADNdb circulaire servira pour
la réplication.
Réplication en cercle roulant (RCR)
:
Etape a : accrochage de la
protéine associée à la réplication (Rep) à
l'origine de réplication (ori).
Etape b : ouverture de l'ADN
et liaison covalente de la Rep à l'extrémité 5'.
Etape c : déplacement
et réplication.
Etape d : nouvelle ouverture
de l'ADN, fermeture des ADN simple brin et relargage de la Rep.
Réplication dépendante de la
recombinaison (RDR) :
Etape e : interaction entre
un ADN simple brin incomplet et la forme super-enroulée de l'ADN viral
(cccDNA) à
des sites homologues.
Etape f : recombinaison
homologue.
Etape g : élongation
de l'ADN simple brin.
Etape h:
synthèse de l'ADN complémentaire et obtention d'un ADN double
brin.
20
Chapitre I : synthèse bibliographique
3.4. Variabilité génétique des
Begomovirus
3.4.1. Mutations
Les mutations sont des erreurs de copies
générées lors de la réplication du génome
par les polymérases ARN ou ADN. Le taux de ces erreurs varie grandement
entre les ADN polymérases, présentant divers mécanismes de
correction et des taux d'erreurs relativement faibles (entre 10-6 et
10-8 erreurs par nucléotide et par réplication;
Garcia-Diaz et Bebenek, 2007) et les ARN polymérases aux taux d'erreurs
nettement plus élevés (entre 10-3et
10-5erreurs par nucléotide et par réplication;
Jenkins et al., 2002). Chez les géminivirus, plusieurs
études ont estimé des taux de substitution de l'ordre de
10-3 à 10-4substitutions par position
nucléotidique et par année (Isnard et al., 1998; Ge
et al., 2007; van der Walt et al., 2008; Duffy et Holmes,
2009). Plusieurs hypothèses ont été avancées pour
expliquer ces taux élevés, tels que l'incompatibilité
entre les enzymes de correction et le génome viral, la présence
de structures secondaires favorisant les erreurs, ou bien le rôle
potentiel de protéines virales dans l'altération de la
fidélité de la polymérase (Gutierrez, 1999;
Arguello-Astorga et al., 2007). Le taux de substitution
élevé des Begomovirus en tant que moteur de leur
diversification est associé à un deuxième processus
évolutif majeur chez les géminivirus: la recombinaison.
3.4.2. Survenue des phénomènes de
recombinaisons
La recombinaison virale est un processus permettant la
genèse d'un nouveau virus issu de nouvelles combinaisons
génomiques à partir de l'information génétique de
deux virus parentaux et de la création de nouveaux types d'arrangement
génomiques. Elle permet l'acquisition en une seule étape d'une
grande variabilité génétique et donc la création de
nouveaux types d'arrangements dans le génome. La recombinaison a
été largement décrite chez les géminivirus. Les
phénomènes de recombinaison ont été décrits
à la fois entre des virus de la même espèce (Zhou et
al., 1997), d'espèces différentes (Monci et al.,
2002) et aussi entre virus appartenant à différents genres
(Saunders et Stanley, 1999). En ce qui concerne le genre Begomovirus,
une multiplicité et une variété de description
d'évènements de recombinaison conduisant à une
diversité moléculaire élevée ont été
décrites ces dernières années (Lefeuvre et al.,
2007 ; Tiendrébéogo et al., 2010).
21
Chapitre I : synthèse bibliographique
3.5. Dissémination spatiale et temporelle des
Begomovirus
L'émergence des maladies à Begomovirus
ces dernières années est due non seulement à la
capacité de recombinaison et d'adaptation de ces virus, mais
également et surtout à la dissémination vectorielle et aux
activités humaines.
3.5.1. Insecte vecteur
Les Begomovirus sont transmis des plantes malades aux
plantes saines par l'intermédiaire d'une mouche blanche, Bemisia
tabaci Genn. (ICTV, 2012).C'est une mouche d'origine tropicale faisant
partie de l'ordre des Hemiptera, de la famille des Aleyrodidae et de la sous-
famille des Aleyrodinae (De Barro et al., 2011). Bemisia tabaci
Genn. est un aleurode mesurant 1 à 1,5 millimètre de long,
de couleur blanc-jaunâtre et disposant de 2 paires d'ailes blanches
(Figure 7). C'est un polyphage piqueur-suceur qui se nourrit de plusieurs
espèces végétales (Osborne, 1988).
Considérée comme l'une des espèces les plus envahissantes
au monde, la mouche blanche se nourrit des liquides intracellulaires des
plantes hôtes. Présente sur la majorité des continents,
elle prolifère surtout dans l'hémisphère sud et aurait
favorisé l'émergence et l'évolution des populations
bégomovirales dans les régions tropicales et subtropicales
(Jeske, 2009).

Figure 8: Bemisia tabaci adulte
(Photo Akakpo, 2019)
Des études récentes ont permis de décrire
plusieurs biotypes à travers les différentes régions du
monde. Parmi ces biotypes, le biotype B a beaucoup contribué à
l'expression des Begomovirus sur de nouveaux hôtes, car il est
très fécond, plus répandu et est
22
Chapitre I : synthèse bibliographique
le plus redoutable ravageur des plantes (Perring et
al., 1991; Delatte et al., 2007). La transmission des
Begomovirus aux plantes se fait selon le mode circulant et persistant
(Czosnek, 2007). Le virus est acquis passivement lors de prise d'aliment de
Bemisia tabaci Genn. et se retrouve ensuite dans les glandes
salivaires de l'insecte. Le virus sera donc transmis à une nouvelle
plante par salivation lors d'une nouvelle alimentation de l'insecte sur une
plante saine.
3.5.2. Activités humaines
L'activité humaine a également été
reconnue dans l'émergence des Begomovirus. Le mode de culture
basé sur la monoculture et le transfert d'espèces
végétales hors de leurs zones géographiques ont
contribué énormément à la mise en place de
conditions favorables à la transmission par Bemisia tabaci
Genn. de virus indigènes vers les plantes exotiques introduites
(Jones, 2009). Ainsi, les échanges de matériel
végétal contaminé et les pratiques culturales
inappropriées favorisent l'élargissement des aires de
distributions des virus.
3.6. Hôtes et symptômes causés par les
Begomovirus
Les Begomovirus sont rencontrés sur une large
gamme d'espèces de plante. Les plantes hôtes les plus
répertoriées dans la littérature appartiennent à la
famille des dicotylédones : Amaranthaceae, Asteraceae,
Brassicaceae, Convolvulaceae, Cucurbitaceae,
Euphorbiaceae, Labiaceae, Fabaceae, Malvaceae,
Solanaceae et Verbenaceae (Silva et al., 2017).
Les principaux symptômes observés (Figure 8) chez les plantes
atteintes de maladies associés aux Begomovirus sont : (1) des
déformations foliaires associées ou non à des
mosaïques, comme la mosaïque du manioc (Cassava Mosaic
Disease, CMD) (Legg et al., 2014a,b),(2) des repliements
foliaires en forme de cuillère associés ou non à du
jaunissement et du nanisme, comme la maladie de l'enroulement et/ou du
jaunissement foliaire de la tomate (tomato (yellow) leaf curl disease,
ToLCD-TYLCD) (Tiendrébéogo et al., 2010 ;
Navas-Castillo et al., 2011; Péréfarres et al.,
2014 ; Mivedor et al., 2017) et la maladie de l'enroulement foliaire
du gombo (okra leaf curl disease, OLCD ; Tiendrébéogo
et al., 2010), et (3) des jaunissements des nervures, comme la maladie
du jaunissement des nervures du piment (pepper yellow vein disease,
PYVD, Tiendrébéogo et al., 2008).
23
Chapitre I : synthèse bibliographique

Figure 9: Symptôme (a) de la maladie de
l'enroulement foliaire du gombo (Okra leaf curl disease, OLCD)
observé au Burkina Faso, (b) de la maladie du jaunissement des nervures
du piment (Pepper yellow vein disease, PYVD ; Tiendrebeogo et al.,
2008) et (c) de la maladie de l'enroulement (et du jaunissement) foliaire
de la tomate (Tomato (yellow) leaf curl disease, ToLCD-TYLCD)
observé au Togo (Mivedor et al., 2017)
4. Principales méthodes de diagnostics des
Begomovirus
Les symptômes caractéristiques d'une infection
à Begomovirus précédemment décrits sont
généralement utilisés pour diagnostiquer les plantes
atteintes de la maladie. Cependant, l'état de ces symptômes et
même l'état général de la plante peuvent être
influencés par d'autres facteurs. Ces facteurs sont entre autres
l'action des insectes ravageurs des feuilles, des insectes piqueurs-suceurs et
aussi du stress hydrique (Jones, 2003). Face à cette difficulté
rencontrée par cette méthode de diagnostic, plusieurs
méthodes de laboratoire ont été développées
(Matthews, 1991). Il s'agit des tests de diagnostic sérologique (Al
Bitar et Luisoni, 1995; Wu et al., 2012) et moléculaire
(Patel et al., 1993; Inoue-Nagata et al., 2004).
4.1. Tests sérologiques
Le test sérologique est basé sur l'utilisation
des techniques de précipitation en immuno-diffusion (ID) et des tests
immuno-enzymatiques (ELISA). Les anticorps utilisant les tests ELISA ont permis
la détection des Begomovirus dans différents extraits
végétaux (Konaté et al., 1995; Harrison et
Robinson,1999). La variante TAS-ELISA utilisant les anticorps monoclonaux a
été utilisée pour la détection et la classification
de ce groupe de virus (Barro, 1994; Konaté et al., 1995;
Tiendrébéogo et al., 2006). Par ailleurs, d'autres tests
sérologiques utilisant des sérums polyclonaux sont
utilisés avec succès. Ce sont: "Western
24
Chapitre I : synthèse bibliographique
blots", "Leafimprint blots", "Tissue blots", "Transmission
electronmicroscopy and immunolabeling" (Abouzid et al., 2002).
4.2. Tests moléculaires
Les techniques moléculaires de détection et
d'identification des virus sont basées sur la réaction de
polymérisation en chaîne (PCR). Des couples d'amorces
spécifiques ou universelles ont été
développés pour la détection respectivement de certaines
espèces de Begomovirus ou de certains groupes de
Begomovirus, notamment à partir d'informations sur la
séquence des régions conservées de l'ADN-A (Wyatt et
Brown, 1996; Atzmon et al., 1998; Brown et al., 2001). De nos
jours, les comparaisons de séquences nucléotidiques et la
phylogénie permettent de distinguer avec précision les
espèces virales (Padidam et al., 1995; Brown, 1997; Fauquet
et al., 2003). Ces comparaisons sont généralement
basées sur la séquence de la région intergénique
(IR) et celle du gène de la protéine de capside (CP) en l'absence
de séquence complète de l'ADN-A (Brown, 1997; Brown et
al., 2001). Mais pour une taxonomie définitive, la séquence
complète de l'ADN-A est nécessaire. Le seuil de distinction
taxonomique des Begomovirus actuellement admis par le comité
international de taxonomie virale (CITV) est de 89% pour l'ADN-A (Fauquet
et al., 2008 ; Brown et al., 2015).
5. Méthodes de lutte contre les
Begomovirus
Les maladies virales des plantes occasionnent de pertes
importantes à la fois sur le rendement et la qualité des produits
agricoles. De nos jours, peu de méthodes de lutte sont disponibles dans
le cas d'une infection virale et surtout, aucun traitement curatif ne peut
être appliqué. Les principales mesures de lutte contre les
Begomovirus visent surtout à réduire les sources de
l'inoculum viral et la population du vecteur. Ces moyens de lutte sont
essentiellement biologiques, chimiques, culturaux et physiques. La lutte peut
être également génétique et basée sur
l'utilisation de variétés résistantes ou tolérantes
au virus et/ou au vecteur (Brown et Bird, 1992; Brown, 1994; Thresh et
Otim-Nape, 1994; Pico et al., 1996; Polston et Anderson, 1997; Varma
et Malathi, 2003).
5.1. Pratiques culturales, prophylaxie
Ce sont des pratiques qui consistent à empêcher
ou réduire considérablement l'arrivée du virus sur les
parcelles de production. Ils consistent donc à empêcher les
introductions et les mouvements de plants infectés par le virus vers les
zones encore indemnes (saines). Ces pratiques constituent une priorité
dans la lutte contre les Begomovirus (Anderson
25
Chapitre I : synthèse bibliographique
et al., 2004). Dans les zones déjà
contaminées, l'emploi de plants sains et certifiés ainsi que
l'élimination des plantes adventices hôtes de Begomovirus
à proximité des parcelles de production constituent les
premières mesures qui tendent à repousser l'infestation
(Sánchez-Campos et al., 1999; Papayiannis et al.,
2011). Également, la rotation des cultures d'une saison à une
autre et l'élimination des résidus de culture sont
également recommandées (Morilla et al., 2005).
Le contrôle de l'insecte vecteur constitue aussi un
élément très important de la lutte contre les
Begomovirus. Ce moyen de lutte peut se baser sur l'utilisation de
barrières physiques (Cohen et Antignus, 1994), de méthodes qui
perturbent le comportement de l'insecte (modification du rayonnement UV dans
les serres, Antignus et al., 2001), de la lutte biologique avec des
insectes auxiliaires des cultures (insectes des genres Eretmocerus et
Encarsia par exemple pour la lutte contre B. tabaci;
Picó et al., 1996) et bien sûr de la lutte chimique
à travers des pulvérisations d'insecticides. Toutefois, les
emplois répétés d'insecticide favorisent le
développement de résistances chez les insectes vecteurs.
5.2. Résistance variétale
Les pratiques culturales et la prophylaxie peuvent aider
à contrôler l'apparition ou le développement de la maladie,
mais sont en général insuffisantes (Antignus, 2007; Polston et
Lapidot, 2007). Une autre méthode plus "simple" à mettre en
oeuvre est l'utilisation de variétés résistantes aux
virus. Cette méthode est la plus commode et la plus rentable pour le
contrôle des maladies virales (Hall, 1980; Yang et al., 2004 ;
De Castro, 2005). Elle reste la meilleure stratégie pour assurer une
production acceptable en quantité et en qualité. La
sélection de plantes naturellement résistantes à la
maladie s'effectue par des croisements. Le plus souvent des successions de
rétrocroisements sont effectuées entre une variété
résistante et une variété sensible présentant des
caractères agronomiques intéressants. Les plantes
résistantes ayant les meilleures qualités agronomiques issues de
ces croisements sont conservées pour être de nouveau
croisées avec la variété sensible. Ce processus est
répété plusieurs fois jusqu'à l'obtention d'une
plante résistante et ayant des qualités agronomiques optimales.
La lutte génétique contre les virus en général
demeure longue à mettre en oeuvre. Il s'écoule parfois plus de 10
ans entre le moment où l'on découvre une résistance et le
moment où cette résistance sera intégrée dans une
variété intéressante. Dans certains cas, la
résistance est spécifique et sera surmontée par des
souches particulières du virus. Il faudra donc créer des
variétés multi résistantes (possédant des
résistances à plusieurs virus à la fois) pour obtenir une
protection optimale des cultures.
CHAPITRE II : MATERIEL ET METHODES
26
Chapitre II : Matériel et méthodes
Chapitre II: Matériel et méthodes
I. Matériel d'étude
1. Cadre et période de l'étude
La présente étude s'est déroulée
au Burkina Faso et plus précisément au Centre de Recherches
Environnementales, Agricoles et de Formation (CREAF) de Kamboinsé. C'est
un centre de l'Institut de l'Environnement et de Recherches Agricoles du Centre
National de la Recherche Scientifique et Technologique (INERA/CNRST). Il est
situé à l'ouest de Ouagadougou sur l'axe Ouagadougou-Kongoussi,
à proximité du barrage de Kamboinsé. Les
coordonnées géographiques sont les suivantes : latitude
12°28N, longitude 1°32W avec une altitude de 296 m du niveau de la
mer. Les travaux de laboratoire ont été effectués au
Laboratoire de Virologie et Biotechnologies Végétales (LVBV)
dudit centre durant 11 mois (3 septembre 2018 au 3 août 2019). Les 4
zones agroécologiques du Togo, notamment (1) la zone
agroécologique de la savane sèche, (2) la zone
agroécologique de la savane humide, (3) la zone agroécologique de
la forêt et (4) la zone agroécologique du littoral ont servi de
sites de prospections et de collectes des échantillons. La prospection
et la collecte des échantillons a été faite en juillet
2018 avec l'aide des Techniciens Spécialisés en Production
Végétale (TSPV) des Instituts de Conseil et d'Appui Technique
(ICAT). Ces techniciens agricoles travaillent pour le compte de l'état
et ont pour rôle d'appuyer les producteurs dans leurs activités
agricoles. Au total 12 localités (figure 9) à grande production
du gombo ont été prospectées.
27
Chapitre II : Matériel et méthodes
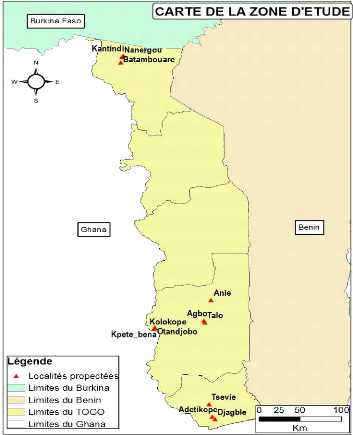
Figure 10: Carte de la zone d'étude
montrant les douze (12) localités prospectées (Photo Akakpo,
2019)
2. Matériel végétal
L'étude a été réalisée
essentiellement sur le gombo. Une jeune feuille de gombo présentant des
symptômes caractéristiques d'une infection à
Begomovirus et une jeune feuille asymptomatique ont été
collectées par plant sur les parcelles prospectées.
3. Matériel de laboratoire et de
prospection
Le matériel de laboratoire est constitué
principalement des outils du plateau technique du Laboratoire de Virologie et
de Biotechnologie Végétale (LVBV) et du Laboratoire Mixte
International (LMI) pour les analyses moléculaires. Le matériel
de prospection a été composé de fiches
d'échantillonnage et de collecte des données, un appareil
28
Chapitre II : Matériel et méthodes
photo numérique, un GPS (GARMIN 62s), des enveloppes,
un stylo à bille, un marqueur et un sac en jute pour le transport des
échantillons.
II. Méthodologie
1. Collecte des échantillons
Au total 118 échantillons (tableau IV) de jeunes
feuilles (une feuille par échantillon) de gombo (Abelmoschus
esculentus (L.) Moench) présentant des symptômes
caractéristiques d'une infection à Begomovirus
(repliement foliaire, enroulement et/ou jaunissement) et des feuilles
asymptomatiques ont été collectées. La collecte a
été effectuée selon la méthode
d'échantillonnage basant sur les diagonales `X' décrite par
Sseruwagi et al (2004). Les feuilles échantillonnées ont
été photographiées, mises dans des enveloppes et les
coordonnées géographiques des localités prospectées
ont été notées à l'aide d'un GPS (GARMIN V.62s).
Sur chaque enveloppe a été inscrit le nom de la localité,
numéro de l'échantillon et les coordonnées
géographiques. Les échantillons prélevés ont
été convoyés puis séchés à
l'étuve à 37°C pendant 5 jours.
Tableau IV : Répartition des
échantillons collectés suivant les zones agroécologiques
et les localités
|
Zones agro
écologiques
|
Localités
|
Nombre d'échantillons
|
Date de
collecte
|
Référence GPS
|
|
Infectés
|
Saines
|
Total
|
|
RH
|
RB
|
JN
|
|
Savane sèche
|
Kantindi
|
3
|
3
|
3
|
1
|
10
|
06/07/2018
|
N10°54.625/E000°16.353/276m
|
|
Batambouaré
|
3
|
2
|
2
|
1
|
8
|
23/07/2018
|
N10°49,129/E000°14,359/274m
|
|
Nanergou
|
3
|
3
|
3
|
1
|
10
|
23/02/2018
|
N10°53.377/E000°15.945/282m
|
|
Savane humide
|
Agbo
|
3
|
3
|
3
|
1
|
10
|
13/07/2018
|
N07,51363°/E001,14212°/893
|
|
Talo
|
3
|
3
|
3
|
1
|
10
|
13/07/2018
|
N07,48825°/E001,15353°/822
|
|
Kolokopé
|
3
|
3
|
3
|
1
|
10
|
20/07/2018
|
N07,40802°/E000,60630°/554
|
|
Anie
|
3
|
3
|
3
|
1
|
10
|
20/07/2018
|
N07,77417°/E001,22226°/584
|
|
Forêt
|
Kpété-béna
|
3
|
3
|
3
|
1
|
10
|
14/07/2018
|
N07,42464°/E000,60711°/832
|
|
Otandjobo
|
3
|
3
|
3
|
1
|
10
|
16/07/2018
|
N07,42397°/E000,60704°/1492
|
|
Littorale
|
Tsévié
|
3
|
3
|
3
|
1
|
10
|
10/07/2018
|
N06°26,521/E001°12,179/104m
|
|
Adetikopé
|
3
|
3
|
3
|
1
|
10
|
10/07/2018
|
N06°18,756/E001°13,512/53m
|
|
Djagble
|
3
|
3
|
3
|
1
|
10
|
10/07/2018
|
N06°15,046/E001°16,613/23m
|
RH=Repliement vers le haut ; RB= Repliement vers le bas ;
JN=Jaunissement 2. Extraction des ADNs totaux
L'extraction de l'ADN total des échantillons de
feuilles de gombo collectées a été réalisée
selon le protocole de Doyle et Doyle (1987) modifié pour l'extraction
d'ADN au
29
Chapitre II : Matériel et méthodes
Cétyl Trimétyl Ammonium Bromide (CTAB). Pour ce
faire, 20 à 30 mg de feuilles sèches a été
pesé à l'aide d'une balance (figure 10 B) et mis dans un tube
eppendorf de 2 ml auquel ont été ajoutées 3 billes
stériles. Le tube a été ensuite placé sur le
portoir du broyeur à bille (figure10, A) pour le broyage pendant 30 s.
Cela nous a permis d'obtenir une poudre bien fine. Ensuite, nous avons
retiré les billes et y ajouté 1,5 ml du tampon d'extraction CTAB
(2% CetylTrimetyl Ammonium Bromide (CTAB), 1,4 M NaCl, 0,2 % beta mercapto
éthanol, 20 mM Etylene Diamine Tetra acetic Acid (EDTA) et 100 mM
Tris-HCl ou Trizma base, pH8) préchauffé à 60°C au
bain-marie. Après vortexage, nous avons incubé
l'échantillon à 60°C au bain-marie sous agitation douce
(toutes les 10 min) pendant 60 min. Après une centrifugation à
12000 rpm pendant 15 min, 1 ml du surnageant a été
prélevé et transféré dans un nouveau tube eppendorf
de 2 ml. Un égal volume de mélange de chloroforme et d'alcool
isoamylique (CIA ; 24:1) a été ajouté au surnageant suivi
d'une agitation douce pendant 5 min. Une deuxième centrifugation
à 12000 rpm pendant 15 min a été réalisée en
vue de concentrer les phases. Cela pour se débarrasser des débris
en suspension dans la phase aqueuse. Après cette opération, 800
tl du surnageant ont été transféré dans un tube
eppendorf de 1.5 ml auquel nous avons ajouté 0.8 de volume d'isopropanol
(640 tl) pour permettre la précipitation des acides nucléiques.
L'échantillon a été ensuite incubé à
-20°C pendant 30 min et centrifugé à 12000 rpm pendant 15
min. Le surnageant a été jeté et 500 tl d'éthanol
70% ont été ajouté au culot pour le lavage. Après
vortexage, l'échantillon a été laissé à
température ambiante pendant au moins 20 min. L'échantillon a
été centrifugé une nouvelle fois à 12000 rpm
pendant 15 min et l'éthanol a été vidé
soigneusement. Nous avons ensuite procédé à un
séchage à température ambiante pour éliminer
l'éthanol. Les ADNs obtenus ont été suspendus dans 100
ìl d'eau distillée stérile puis gardés à
4°C pour dissoudre les ADNs. Cet ADN total a été dosé
au Nanodrop pour vérifier sa qualité (bonne qualité avec
un ratio DO260/DO280 ~1,8-2,0) et sa concentration (en ng/tl) puis
conservés à -20°C pour les analyses.
Chapitre II : Matériel et méthodes

Figure 11: (A) broyeur à bille
(Tissuelyser II) utilisé pour le broyage des échantillons ; (B)
balance électronique utilisée pour peser les échantillons
(Photo Akakpo, 2019)
3. Détection des Begomovirus par la
PCR
Pour l'identification des Begomovirus et des
satellites (Betasatellites), nous avons utilisé la PCR conventionnelle
avec des amorces dégénérées. L'ADN total extrait a
été dilué à 100 ng/ul et a servi de matrice pour
l'amplification.
3.1. Amorces utilisées
Les amorces consignées dans le tableau V ont
été utilisées pour la détection des Begomovirus
et des éventuels ADN Betasatellites dans les échantillons.
Le couple d'amorces VD360/CD1266 est spécifique aux gènes codant
pour la protéine de la capside (CP) localisés sur l'ADN-A des
Begomovirus. Le couple d'amorces Beta01/Beta02 est spécifique
d'une région conservée de l'ADN Betasatellite.
Tableau V: Liste des amorces utilisées
pour les différentes réactions PCR
Détection de l'ADN A Séquences des amorces
Tailles
attendues (pb)
VD360/CD1266 (Dellate et al., 5'-AGRCTGAACTTCGACAGC-3'
906
2005) 5'-TCTCAACTTCARGGTCTG-3'
Détection de l'ADN Beta
Beta01/Beta02 (Briddon et al.,
5'-GGTACCACTACGCTACGCAGCAGCC-3' ~1400
2002) 5'-GGTACCTACCCTCCCAGGGGTACAC-3'
30
31
Chapitre II : Matériel et méthodes
3.2. Composition des mix et programmes PCR
Les réactions PCR ont été conduites selon
les protocoles de Briddon et al. (2002), Bull et al. (2003)
et Delatte et al. (2005). Pour les deux couples d'amorcesVD360/CD1266
et Beta01/Beta02, la composition des mix PCR a été conforme aux
indications des auteurs ci-dessus et est consignée dans les tableaux VI
et VII ci-dessous :
Tableau VI: Composition du Mix PCR pour la
détection des Begomovirus
|
Réactifs
|
Volume pour une réaction (pd)
|
Conc.
Initiale
|
Conc. finale
|
|
ADN
|
2
|
|
|
|
VD360
|
1
|
10 uM
|
0,5 uM
|
|
CD1266
|
1
|
10 uM
|
0,5 uM
|
|
H2O qsp 20
|
13
|
|
|
|
Taq Polymerase (Hot firepol)
|
3
|
5U/ul
|
1U
|
|
Vol total mix
|
20
|
|
|
Tableau VII: Composition du Mix PCR pour la
détection des Betasatellites
|
Réactifs
|
Volume pour une réaction (pd)
|
Conc.
Initiale
|
Conc.
Finale
|
|
ADN
|
2
|
|
|
|
Beta 01
|
1
|
10 uM
|
0,5 uM
|
|
Beta 02
|
1
|
10 uM
|
0,5 uM
|
|
H2O qsp 20
|
13
|
|
|
|
Taq Polymerase (Hot firepol)
|
3
|
5U/ul
|
1U
|
|
Vol total mix
|
20
|
|
|
Pour la réalisation de la PCR proprement dite, nous
avons utilisé des tubes de 0,2 ml dans lesquels nous avons
préparé nos mix PCR composés des éléments
listés dans le tableau VI et VII et 5. L'amplification a
été effectuée à l'aide d'un thermocycleur (figure
12). Le programme d'amplification était le suivant :
Le programme PCR du couple d'amorces VD360/CD1266 est le suivant
:
32
Chapitre II : Matériel et méthodes
35 cycles
95°C pendant 12 min pour la dénaturation initiale
94°C pendant 30s pour la dénaturation 55°C
pendant 30s pour l'hybridation 72°C pendant 1min pour
l'élongation
72°C pendant 10min pour l'élongation finale
Le programme PCR du couple d'amorces Beta01/Beta02 est le suivant
:
|
95°C pendant 12 min pour la dénaturation initiale
94°C pendant 1 min pour la dénaturation 54°C pendant 1 min
pour l'hybridation 72°C pendant 1 min 30s pour l'élongation
|
35 cycles
|
72°C pendant 10 min pour l'élongation finale

Figure 12: Thermocycleur 2720 (Applied
Bioystems) utilisé pour l'amplification (Photo Akakpo, 2019)
4. Analyse des produits de la PCR 4.1. Préparation
du gel
Afin de vérifier l'efficacité des
amplifications, les produits de la PCR ont été analysés.
À cet effet, un gel d'agarose (sigma grade biologie moléculaire)
à 1% (p/v) a été préparé dans du tampon TAE
0,5X (Tris base, acide acétique glacial, EDTA 0,5M pH 8). La
33
Chapitre II : Matériel et méthodes
préparation a été faite comme suit : 1 g
d'agarose a été additionné à 100 ml de tampon TAE
0,5X (Tris base, acide acétique glacial, EDTA 0,5M pH 8). Le
mélange a été porté au micro-onde afin de
solubiliser la solution. 4ul de bromure d'éthydium (BET) ont
été incorporées au gel. Le mélange a
été coulé sur un support de gel pour
l'électrophorèse. Un peigne formé de 13 dents a
été préalablement placé sur le support afin de
former 13 puits. Le gel a été laissé à
température ambiante pour gélification.
4.2. Électrophorèse et visualisation
Après gélification, le gel a été
plongé dans une cuve d'électrophorèse contenant du tampon
TAE 0,5X (Tris base, acide acétique glacial, EDTA 0,5M pH 8) de sorte
à immerger totalement le gel dans la cuve. Une quantité de 12,5
ìl des produits d'amplification a été
prélevée à l'aide d'une micropipette et
déposée dans les puits du gel d'agarose à 1%. Dans le
premier puits, a été déposé le marqueur de poids
moléculaire 100 paires de bases DNA ladder (Solis BioDyne) afin de
servir de référentiel pour mesurer la taille de nos bandes. L'eau
(H2O), un témoin positif constitué de l'ADN du manioc
infecté par le ACMV (African cassava mosaic virus) et un
témoin négatif constitué de l'ADN du manioc non
infecté par un Begomovirus ont été également
déposés dans les trois derniers puits. La migration
électrophorétique a été faite à 100 volts
pendant 35 minutes. La visualisation a été faite à l'aide
d'un transilluminateur UV (figure 13).

Figure 13: GEL DOC Major Science (ms) UVDI
utilisé pour la visualisation (Photo Akakpo, 2019)
34
Chapitre II : Matériel et méthodes
5. Séquençage de certains produits
PCR
En vue de séquencer les produits PCR après les
tests de diagnostic, 12 échantillons ont été
sélectionnés sur la base de la diversité puis
amplifiés. Les réactions d'amplification ont été
conduites dans un volume final de 40 ul. Après l'amplification, 10 ul
des produits PCR ont été mélangés avec 1ìl
de tampon bleu de charge (0,040 % bromophénol, 7% glycérol, 6 mM
EDTA) puis déposés sur le gel d'agarose (1 %) afin de
vérifier la taille des amplicons. Les 30 ul restants ont
été dosés à l'aide d'un Nanodrop pour s'assurer que
la concentration finale est supérieure ou égale à de 50
ng/ul (concentration indiquée par le compagnie de
séquençage). Les produits ont été envoyés
à la compagnie Macrogen Europe (Pays Bas) pour le
séquençage.
6. Analyses statistiques et
phylogénétiques 6.1. Analyses statistiques
Le logiciel Microsoft Excel version 2013 a été
utilisé pour les calculs de moyennes, de la prévalence de la
maladie de l'enroulement foliaire du gombo et pour la construction des
graphiques. Les pourcentages sont calculés en appliquant la formule
suivante :
6.2. Analyses phylogénétiques
Pour mieux comprendre les informations obtenues,
différents programmes bioinformatiques ont été
utilisés. Les fragments de séquençage appelés
"contigs" issus du séquençage, ont été
édités et assemblés à l'aide du logiciel Geneious
10.2.3. Après obtention des séquences consensus, l'algorithme
BLASTn du site NCBI (
https://www.ncbi.nlm.nih.gov/)
a été utilisé pour la recherche de l'identité des
virus par comparaison à des séquences existantes dans des banques
de données nucléotidiques (GenBank-EMBL-DDBJ). À travers
le même site, nous avions procédé à la recherche de
séquences proches des nôtres pour la construction de l'arbre
phylogénétique. Le RDP 5.3 a été utilisé
pour sélectionner les séquences d'intérêt. Le
logiciel MEGA X 10.0.5 a servi à l'alignement des séquences
obtenues et à la construction de l'arbre phylogénétique
selon la méthode du maximum de vraisemblance (Maximum Likelihood
method). Les arbres phylogénétiques ont été
construits par le logiciel avec 1000 répétitions de bootstrap. Le
logiciel FigTreev1.4.3 a été utilisé pour la visualisation
de l'arbre et le logiciel Inkscape 0.92.4 pour l'édition des noms des
espèces virales sur l'arbre.
CHAPITRE III : RÉSULTATS ET DISCUSSION
35
Chapitre III : Résultats et discussion
Chapitre III: Résultats et discussion
I. Résultats
1. Symptômes observés dans les parcelles de
production de gombo au Togo
Les prospections des champs de gombo dans les 4 zones agro
écologiques du Togo ont permis d'observer divers types de
symptômes caractéristiques d'une infection à
Begomovirus chez le gombo. Toutes les variétés de gombo
rencontrées présentaient des symptômes.
La Figure 14 illustre la diversité des symptômes
observés sur le gombo lors des prospections. Les symptômes
observés sont des déformations, jaunissements et des enroulements
foliaires en forme de cuillères ou des enroulements des feuilles sur
elle-même. Dans certains cas les feuilles de gombo sont repliées
vers le bas. D'autres plants infectés présentent des nervures
épaisses, subissent un rabougrissement avec des feuilles de très
petite taille.
Dans l'ensemble, la majorité des champs
prospectés étaient infectés à l'exception de
quelques champs dont les plants étaient au stade levé.
36
Chapitre III : Résultats et discussion

A
E
C
D
B
F
Figure14: (B) et (D) plants de gombo avec des
feuilles déformées (les feuilles présentent des
déformations du limbe avec un épaississement des nervures); (C)
enroulement des feuilles de gombo sur elles même (repliement des feuilles
sur elle-même vers le haut ou vers le bas avec rabougrissement du plant);
(A) et (F) feuilles de gombo en formes de cuillères (une forme de
repliement des feuilles vers le haut en forme d'une cuillère
accompagné parfois de petites déformations du limbe); (E)
feuilles
37
Chapitre III : Résultats et discussion
de gombo présentant des jaunissements (feuilles
présentant de petites tâches chlorotiques (jaune) en mosaïque
sur le limbe) . (Photo Akakpo, 2018)
2. Évaluation de la prévalence de la maladie
de l'enroulement foliaire du gombo au Togo
L'évaluation de la prévalence de la maladie de
l'enroulement foliaire du gombo au Togo a été faite en
considérant les 4 zones agro- écologiques du Togo. Pour se faire,
un total de 118 échantillons de feuilles de gombo (symptomatiques et
asymptomatiques) a été collecté dans 12 localités
(Kantindi, Batambouaré, Nanergou, Agbo, Talo, Kolokopé,
Anié, Kpété-béna, Otandjobo, Tsévié,
Adetikopé, Djagblé) appartenant à ces 4 zones
agroécologiques qui sont : la zone littorale, la zone forestière,
la savane humide et la savane sèche. La maladie a été
identifiée dans toutes les localités. Les analyses statistiques
des résultats obtenus après analyses révèlent que
la prévalence de cette maladie au Togo varie d'une localité
à une autre ainsi que d'une zone agroécologique à une
autre. Elle est de l'ordre de 60% dans la zone forestière, 53,33% dans
la zone littorale, 50% dans la savane humide et 64,29% dans la savane
sèche (tableau VIII). Sur le plan national, la prévalence moyenne
de la maladie était de 56,91%.
Tableau VIII: Prévalences de la maladie
dans les quatre zones agroécologiques
|
Zone
agroécologique
|
Nombre
d'échantillons
|
PCR (+)
|
Prévalence (%)
|
|
Forêt
|
20
|
12
|
60,00
|
|
Littorale
|
30
|
16
|
53,33
|
|
Savane humide
|
40
|
20
|
50,00
|
|
Savane sèche
|
28
|
18
|
64,29
|
3. Caractérisation moléculaire des
espèces virales associées à la maladie de l'enroulement
foliaire du gombo
3.1. Amplification du gène de la protéine
capsidiale par la PCR
L'analyse moléculaire a consisté à faire
une amplification du gène de la protéine capsidiale (CP),
utilisé comme le marqueur de diversité chez les
Begomovirus. La Figure 15 illustre les résultats
d'amplification du gène de la CP (protéine capsidiale) en
utilisant les couples d'amorces VD 360/CD 1266. L'amplification a permis
d'obtenir une bande d'environ 906 paires de bases pour les différents
échantillons suspectés d'infection à Begomovirus.
Ce test a permis de détecter 66 échantillons positifs sur 118
échantillons collectés (tableau IX).
38
Chapitre III : Résultats et discussion
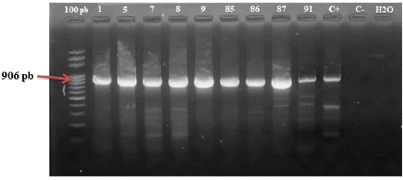
Figure15 : Gel d'électrophorèse
présentant les produits d'amplification du gène de la CP
(fragments attendus de 906 paires de bases) sur gel d'agarose à 1%.
100pb = marqueur de taille DNA ladder, 1 ; 5 ; 7 ; 8 ;
9 ; 85 ; 86 ; 87 ; 91 = échantillons positifs,
C+= témoin positif (échantillon positif à
l'ACMV,African cassava mosaic virus), C-=témoin négatif
(échantillon Begomovirus négatif), H2O=Contrôle
eau.
Tableau IX: Récapitulatif des
résultats PCR par zone agro-écologique et par localité
Zone agro-écologique Ville/village
Coordonnées_GPS Code_éch symptômes
Résultats_PCR
|
Littorale
|
Adetikope N06°18,447/E001°14,172 61AK Saine -
Adetikope 62AK JN +
Adetikope 63AK JN +
Adetikope 64AK JN +
Adetikope 65AK RH +
Adetikope 66AK RH -
Adetikope 67AK RH +
Adetikope 68AK RB -
Adetikope 69AK RB +
Adetikope 70AK RB -
Tsevie N06°26,521/E001°12,179 91AK RB +
Tsevie 92AK RB -
Tsevie 93AK RH +
Tsevie 94AK RH +
Tsevie 95AK RH +
Tsevie 96AK JN +
Tsevie 97AK JN +
Tsevie 98AK JN +
Tsevie 99AK saine -
Tsevie 100AK RB -
Djagble N06°15,046/E001°16,613 101AK saine -
Djagble 102AK RH +
Djagble 103AK RH -
|
39
|
|
|
Chapitre III : Résultats et
discussion
|
|
|
|
|
|
|
|
Zone agro-écologique
|
Ville/village
|
Coordonnées_GPS
|
Code_éch
|
symptômes
|
Résultats_PCR
|
|
Djagble Djagble Djagble Djagble Djagble Djagble Djagble
|
|
104AK 105AK 106AK 107AK 108AK 109AK 110AK
|
RH JN JN JN RB RB RB
|
+ + - - - - -
|
|
Forêt
|
Kpété-béna Kpété-béna
Kpété-béna Kpété-béna
Kpété-béna Kpété-béna
Kpété-béna Kpété-béna
Kpété-béna Kpété-béna Otandjobo
Otandjobo Otandjobo Otandjobo Otandjobo Otandjobo Otandjobo Otandjobo Otandjobo
Otandjobo
|
N07,42464°/E000,60711°
N07,42397°/E000,60704°
|
1AK 2AK 3AK 4AK 5AK 6AK 7AK 8AK 9AK 10AK 41AK 42AK 43AK 44AK 45AK
46AK 47AK 48AK 49AK 50AK
|
RH Saine RB RB RB JN JN JN RH RH Saine RH RH RH JN JN JN RB RB
RB
|
+ - + + + + + + + - + - - - + - + + - -
|
|
Savane humide
|
Agbo Agbo Agbo Agbo Agbo Agbo Agbo Agbo Agbo Agbo Talo Talo Talo
Talo Talo Talo Talo Talo Talo
|
N07,51363°/E001,14212°
N07,48825°/E001,15353°
|
71AK 72AK 73AK 74AK 75AK 76AK 77AK 78AK 79AK 80AK 51AK 52AK 53AK
54AK 55AK 56AK 57AK 58AK 59AK
|
Saine RB RB RB JN JN JN RH RH RH Saine RB RB RB RH RH RH JN JN
|
- - - + - - - + - - + - + + + - - -
-
|
40
Chapitre III : Résultats et discussion
Zone agro-écologique Ville/village
Coordonnées_GPS Code_éch symptômes
Résultats_PCR
Talo 60AK JN +
Anie N07,77417°/E001,22226° 81AK Saine -
Anie 82AK RH -
Anie 83AK RH -
Anie 84AK RH +
Anie 85AK RB +
Anie 86AK RB +
Anie 87AK RB +
Anie 88AK JN -
Anie 89AK JN +
Anie 90AK JN -
Kolokope N07,40802°/E000,60630° 21AK RB +
Kolokope 22AK RB +
Kolokope 23AK RB -
Kolokope 24AK RH +
Kolokope 25AK RH -
Kolokope 26AK RH +
Kolokope 27AK JN +
Kolokope 28AK JN +
Kolokope 29AK JN +
Kolokope 30AK Saine +
|
Savane sèche
|
Batambouare N10°49,129/E000°14,359 111AK RH -
Batambouare 112AK RH -
Batambouare 113AK RH +
Batambouare 114AK RB -
Batambouare 115AK RB +
Batambouare 116AK JN +
Batambouare 117AK Saine +
Batambouare 118AK JN +
Kantindi N10°54,176/E000°19,453 11AK RH -
Kantindi 12AK RH -
Kantindi 13AK RH +
Kantindi 14AK RB +
Kantindi 15AK RB +
Kantindi 16AK RB +
Kantindi 17AK JN -
Kantindi 18AK JN +
Kantindi 19AK JN +
Kantindi 20AK Saine +
Nanergou N10°53.377/E000°15.945 31AK RB -
Nanergou 32AK RB -
Nanergou 33AK RB +
Nanergou 34AK RH -
Nanergou 35AK RH +
Nanergou 36AK RH +
Nanergou 37AK Saine +
|
41
Chapitre III : Résultats et discussion
|
Zone agro-écologique Ville/village
Coordonnées_GPS
|
Code_éch
|
symptômes
|
Résultats_PCR
|
|
Nanergou
|
38AK
|
JN
|
-
|
|
Nanergou
|
39AK
|
JN
|
+
|
|
Nanergou
|
40AK
|
JN
|
+
|
RH : repliement vers le haut ;
RB : repliement vers le bas ; JN :
jaunissement
3.2. Amplification des â satellites
Afin de vérifier une éventuelle association des
Begomovirus détectés avec le â satellite, une
amplification a été conduite sur les 66 isolats positifs. Cette
amplification a permis de détecter la présence de â
satellite dans 25 isolats sur 66 isolats soit 37,88%. La Figure 16 illustre les
résultats d'amplification des â satellites en utilisant les
couples d'amorces Beta01/Beta02. La bande attendue d'environ 1400
nucléotides a été obtenue dans certains
échantillons avec succès.

Figure 15 : Electrophorèse des
produits d'amplification des â satellites (fragments attendus de 1400
paires de bases) sur gel d'agarose à 1%. 100 paires de bases = marqueur
de taille DNA ladder. 36 ; 37 ; 39 ; 40 ; 41 ; 54 sont positifs.
3.3. Séquençage des produits PCR du
gène de la protéine capsidiale
Le séquençage des produits PCR a permis d'avoir
l'identité des différentes espèces de Begomovirus
infectant le gombo au Togo. En effet, les produits du
séquençage ont permis d'obtenir des séquences dites
partielles, car concernant seulement le gène de la protéine de
capside (CP) des virus détectés. Au total neuf (9)
séquences ont été obtenues avec succès après
éditions et assemblages des contigs issus des produits de
séquençage. Cela a permis de générer neuf (9)
séquences virales que sont : 5AK, 6AK, 28AK, 39AK, 47AK, 62AK, 85AK,
93AK, 118AK. Ces séquences partielles soumises aux bases de
données génomiques (NCBI, EMBL et DDBJ) à l'aide de
l'algorithme BLAST ont révélé l'identité des
différents Begomovirus infectant le gombo au Togo. Les «
Blast search » ont révélé que les séquences
42
Chapitre III : Résultats et discussion
du Togo sont très proches de deux espèces de
Begomovirus. Il s'agit du Cotton leaf curl Gezira virus
(CLCuGeV) et du Okra yellow crinkle virus (OYCrV). Les
pourcentages d'homologie entre les isolats de Cotton leaf curl Gezira virus
(CLCuGeV) du Togo sont compris entre 88,71% et 96,56% avec CLCuGeV-Burkina
Faso (FN554528), CLCuGeV - Niger (FJ469627). Les isolats de Okra yellow
crinkle virus (OYCrV) du Togo quant à eux ont entre 86,52% et
97,70% d'identité nucléotique avec des isolats de Okra yellow
crinkle virus (OYCrV) de la Côte d'Ivoire (KX100572), du Mali
(EU024118) et du Cameroun (FM164724).
4. Diversité des virus responsables de la maladie de
l'enroulement foliaire du gombo au Togo
Afin d'apprécier la diversité des espèces
de Begomovirus obtenues après identification, une analyse
phylogénétique a été conduite. L'arbre
phylogénétique a été construit selon la
méthode dite du «Maximum likelihood» avec 1000
répétitions de bootstrap. L'isolat MaLCV-[CN:Fuj:06] (FJ712189) a
été utilisé comme racine de l'arbre. Ainsi, la topologie
de l'arbre phylogénique construite à partir des 9 isolats du Togo
a permis de confirmer cette proximité des isolats du Togo avec les deux
espèces virales susmentionnées. Parmi les neuf (9) isolats, 7 ont
formé un groupe homogène (bootstrap égale à 100)
avec des souches de Cotton leaf curl Gezira virus (CLCuGeV) du Burkina
Faso, du Niger et du Nigeria. Les 2 autres souches forment un groupe
homogène (bootstrap égale à 100) avec des souches de
Okra yellow crinkle virus (OYCrV) du Mali, de la Côte d'Ivoire et du
Cameroun (figure 16 A).
L'alignement des séquences réalisé par la
méthode des distances a permis de comparer les séquences deux
à deux et de faire ressortir les similitudes entre elles. La figure 16 B
présente les similitudes entre les séquences de la CP des isolats
caractérisés et ceux existant dans la banque de données
génomiques GenBank. Ces résultats mettent en évidence une
divergence nucléotidique assez variable entre les différentes
séquences.
43
Chapitre III : Résultats et discussion
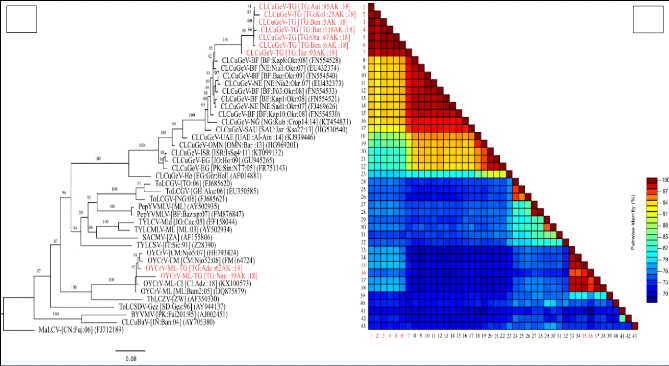
A
B
Figure16: (A) : Arbre phylogénique
généré à partir des séquences de CP des
CLCuGeV, OYCrV du Togo et d'autres Begomovirus existant dans le GenBank.
L'arbre a été construit selon la méthode dite du
«Maximum likelihood» avec 1000 répétitions de bootstrap
dont les pourcentages sont indiqués aux différents noeuds.
(B) : Matrice d'identité des 9 isolats par rapport aux
autres isolats recueillis sur GenBank.
44
Chapitre III : Résultats et discussion
II. Discussion
Le gombo (Abelmoschus esculentus (L.) Moench), de par
son importance nutritionnelle et économique constitue l'un des
légumes les plus cultivés en Afrique. Au Togo il est
cultivé durant toute l'année sur presque toute l'étendue
du territoire. Cependant, force est de constater que la production de ce
légume en Afrique de façon générale et
particulièrement au Togo est confrontée à un certain
nombre de contraintes occasionnant des pertes importantes de rendement. Parmi
ces contraintes, figure en bonne place la maladie de l'enroulement foliaire du
gombo (OLCD). Cette maladie induisant des symptômes de repliements
foliaires est fréquemment observée sur le gombo ces
dernières années au Togo tout comme dans plusieurs pays de
l'Afrique de l'Ouest. Aussi c'est un légume longtemps
délaissé par la recherche et considéré comme «
culture de femme » dans la plupart des pays dans lesquels il est
cultivé.
Dans le but d'identifier les espèces virales
associées à cette maladie et leur diversité au Togo, des
travaux de caractérisation moléculaire ont été
menés sur 118 échantillons de feuilles de gombo (symptomatiques
et asymptomatiques) collectés dans les 4 zones agro écologiques
(Foret, littoral, savane humide et savane sèche) du Togo. Au total 12
localités à grande production ont été
prospectées. Divers symptômes caractéristiques d'une
infection à Begomovirus ont été observés
sur la plupart des parcelles prospectées. Bien que l'observation des
symptômes soit utile pour l'identification de la maladie de l'enroulement
foliaire du gombo, elle n'est pas appropriée pour déterminer des
espèces virales responsables de l'OLCD. Ainsi les ADNs totaux des
échantillons ont été extraits et soumis à un
diagnostic moléculaire par PCR (Polymerase Chain Reaction)
conventionnelle à l'aide d'une amorce
dégénérée (VD 360/CD 1266) (Dellate et
al., 2005) spécifique au gène codant pour la protéine
de capside (CP) des Begomovirus. La CP a été
amplifié avec succès à partir de certains
échantillons (65/118). Ce couple d'amorces semblerait le mieux
adapté à la détection moléculaire des
Begomovirus en Afrique de l'Ouest. Les résultats obtenus ont
permis d'associer les divers symptômes observés au groupe des
Begomovirus, corroborant ainsi les travaux antérieurs portant
sur le OLCD dans certains pays de l'Afrique de l'Ouest dont le Burkina Faso
(Barro et al., 2007 ; Konaté et al., 1995 ;
Tiendrébéogo et al., 2010), la Côte d'Ivoire
(Séka et al., 2016 ), le Niger (Shih et al., 2009 ),
le Mali (Kon et al., 2009), le Ghana (Swanson et Harrison, 1993), le
Nigéria (Swanson et Harrison, 1993) et du centre comme le Cameroun (Leke
et al., 2007) et le Tchad (Swanson et Harrison, 1993). Afin de
déterminer les espèces de Begomovirus responsables, un
séquençage s'avérait nécessaire. À cet
effet, les
45
Chapitre III : Résultats et discussion
produits PCR de 12 échantillons (3 par zone agro
écologique) ont été séquencés (par Macrogen
Europe). Après séquençage, 9 produits de
séquençage étaient exploitables. L'analyse des neuf (9)
séquences obtenues a révélé une identité
élevée avec deux espèces virales ; le Cotton leaf curl
Gezira virus (CLCuGeV) et le Okra yellow crinkle virus (OYCrV).
C'est la première fois que ces 2 espèces virales ont
été identifiées sur le gombo au Togo. Le Okra yellow
crinkle virus (OYCrV) a été décrit pour la
première fois en Afrique de l'Ouest au Mali (Shih et al., 2007;
Kon et al., 2009), puis quelques années après en
Côte d'Ivoire (Séka et al., 2016). Le Cotton leaf
curl Gezira virus (CLCuGeV) quant à lui, il a été
identifié à la base au Soudan (Idris et Brown, 2002, 2005) puis
par la suite au Niger (Shih et al., 2009), au Burkina Faso
(Tiendrébéogo et al., 2010) et en Côte d'Ivoire
(Séka et al., 2016) sur le gombo. Il a été
également associé à la maladie de l'enroulement foliaire
du cotonnier (Gossypium sp. L.) au Soudan (Idris et al.,
2000), en Inde (Sohrab et al., 2014) et au Pakistan (Iqbal et
al., 2014 ). Au regard de la dynamique de ces deux espèces virales
dans la sous-région ouest-africaine ces dernières années,
leur découverte au Togo n'est pas surprenante. La propagation de ces
virus pourrait s'expliquer par la dissémination des populations de
mouche blanche qui constituent les principaux vecteurs. Notre étude a
montré que le CLCuGeV est majoritaire et se retrouve dans les 4 zones
agro écologiques comparativement à OYCrV qui a été
identifié que dans deux zones agro écologiques (Littoral et
Savane sèche). Ces résultats montrent que cette maladie est
méconnue au Togo. L'impact du Cotton leaf curl Gezira virus
(CLCuGeV) sur le rendement (nombre de fruits par plante, longueur,
diamètre et poids du fruit) a été évalué au
Burkina Faso sur quatre accessions de gombo (cultivar local,`Man Yanga') et
quatre cultivars commerciaux (Clemson spineless, Indiana, Lima et
Volta) auchamp en 2007 et 2008 (Tiendrébéogo et al.,
2010). Des pertes de rendement importantes ont été
observées pour le cultivar local (26-55%) comparativement aux cultivars
commerciaux (4,4-9,6%). Ces pertes ont été estimées
économiquement à 11100 et 1950 dollars à l'hectare,
respectivement chez le cultivar local et les cultivars commerciaux. Ainsi, la
présence de ces deux espèces virales (CLCuGeV et OYCrV)
constituerait un véritable problème non seulement pour la
production du gombo, mais aussi pour celle du coton au Togo comme les autres
pays producteurs du coton en Afrique de l'Ouest notamment : le Burkina Faso, le
Bénin, la Côte d'Ivoire, le Mali et le Sénégal. Pour
cela, des mesures rigoureuses doivent être prises en vue d'un
contrôle efficace de la propagation de ces virus afin de sécuriser
durablement la culture du gombo et celle du coton dont l'exportation contribue
considérablement au PIB des différents pays producteurs.
CONCLUSION, RECOMMANDATIONS
ET PERSPECTIVES
46
Conclusion, recommandations et perspectives
Conclusion
Première du genre au Togo, cette étude a
été conduite au Burkina Faso au Centre de Recherches
Environnementales, Agricoles et de Formation de Kamboinsé (CREAF/K) de
l'Institut de l'Environnement et de Recherches Agricoles (INERA). L'objectif
était d'évaluer la diversité des Begomovirus
infectant le gombo au Togo. La prospection et la collecte des
échantillons ont été effectuées dans les 4 zones
agroécologiques. Cependant, divers symptômes ont été
observés : déformation, enroulement et jaunissement des feuilles.
L'analyse moléculaire effectuée sur la base du gène codant
pour la protéine de capside (CP) a permis d'associer 2 espèces de
Begomovirus à la maladie de l'enroulement foliaire du gombo au
Togo. Il s'agit du Cotton leaf curl Gezira virus (CLCuGeV) et du
Okra yellow crinkle virus (OYCrV). Le CLCuGeV a été
retrouvé dans toutes les zones prospectées tandis que OYCrV quant
à lui, a été identifié que dans le littoral et la
savane sèche. Cette caractérisation a permis non seulement de
connaitre la diversité génétique de ces 2 espèces
virales, mais également de déterminer leur répartition
géographique. Toutefois ces deux espèces virales constituent une
menace pour la culture du gombo et du coton au Togo tout comme dans les autres
pays de l'Afrique de l'Ouest. Ainsi, des mesures de contrôle
s'avèrent indispensables pour contrer la propagation de ces virus afin
d'accroitre les rendements de la production du gombo et celle du coton.
Recommandations
Au vu des résultats obtenus dans cette étude,
les recommandations suivantes peuvent être formulées à
l'endroit du Ministère de l'Agriculture, de la Production Animale et
Halieutique (MAPAH):
? D'accorder une importance particulière aux deux
espèces virales identifiées afin de sécuriser durablement
la culture du gombo et du coton au Togo.
? De renforcer la capacité des agents techniques
d'agriculture sur la reconnaissance des symptômes et les
stratégies de lutte contre ces virus à travers des ateliers de
formation. Ces derniers pourraient donc accompagner les producteurs dans la
mise en oeuvre de ces stratégies.
Tout cela, afin d'intensifier la production du gombo et celle
du coton au Togo et partant de là à lutter contre
l'insécurité alimentaire tout en améliorant les conditions
de vie des producteurs.
47
Conclusion, recommandations et perspectives
Perspectives
Le travail réalisé dans le cadre de ce
mémoire est loin d'être exhaustif. Afin de prendre en compte de
façon exhaustive toute la diversité de ces espèces virales
au Togo, il serait judicieux de :(i) poursuivre les travaux de
caractérisation moléculaire sur un grand nombre
d'échantillons collectés aussi bien sur le gombo que sur le
cotonnier. Cela permettra de mieux appréhender la diversité des
deux espèces virales dans les différentes régions du pays.
(ii) Faire une RCA (Rolling Circle Amplification) et un clonage des
deux virus identifiés afin d'obtenir leur génome complet. Cela
nous renseignera davantage sur l'identité réelle de ces virus et
nous permettra d'avoir une taxonomie définitive. Aussi, il serait
intéressant d'étendre l'étude sur d'autres cultures comme
les cultures maraichères (Tomate, poivron, piment, etc.), les fruitiers
et les mauvaises herbes. Tous ces travaux permettront d'obtenir des
informations scientifiques à l'endroit des producteurs et des
décideurs politiques pour une agriculture durable et plus rentable.
RÉFÉRENCES BIBLIOGRAPHIQUES
48
Références
bibliographiques
Références bibliographiques
Abouzid A.M., Freitas-Astua J., Purcifull D.E.,
Polston J.E., Beckham K.A., Crowford W.E., Petersen M.A., Peyser B., Patte B.,
Hiebert E., 2002. Serological studies using polyclonal antisera
prepared against the viral coatprotein of four Begomoviruses expressed in
Escherichia coli. Plant Diseases 86:
1109-1114.
Al Bitar L. et Luisoni, E., 1995. Tomato
yellow leaf curl geminivirus: Serological evaluation of an improved
purification method. Bulletin OEPP, 25, 269-276.
Al Rwahnih M., Alabi O.J., Westrick N. M., Golino D.
et Rowhani A., 2017. Description of a novel monopartite geminivirus
and its defective subviral genome in grapevine. Phytopathology,
107, 240-251.
Anderson P.K., Cunningham A.A., Patel N.G., Morales
F.J., Epstein P.R. et Daszak P., 2004. Emerging infectious diseases of
plants: pathogen pollution, climate change and agrotechnology drivers.
Trends in Ecology and Evolution, 19, 535-544.
Antignus Y., 2007. The management of Tomato
yellow Leaf Curl Virus in Greenhouses and the Open Field, a Strategy of
Manipulation BT - Tomato Yellow Leaf Curl Virus Disease: Management,
Molecular Biology, Breeding for Resistance. Tomato Yellow Leaf Curl
Virus Disease (ed. by H. Czosnek), pp. 263-278.
Antignus Y., Nestel D., Cohen S. et Lapidot M., 2001.
Ultraviolet-deficient greenhouse environment affects whitefly
attraction and flight-behavior. Environmental Entomology,
30, 394-399.
Anwar R., Rashid U., Ashraf M., Nadeem M.,
2010. Okra (Hibiscus esculentus) seed oil for biodiesel
production. Applied Energy, 87(3), 779-785.
Arguello-Astorga G., Ascencio-Ibáñez
J.T., Dallas M. B., Orozco B. M. et Hanley-Bowdoin L.,
2007.High-frequency reversion of geminivirus replication protein
mutants during infection. Journal of Virology, 81,
11005-15.
Atzmon G., Van Oss H., Czosnek H., 1998. PCR
amplification of tomato yellow leaf curl virus (TYLCV) DNA from squashes of
plants and whitefly vectors: Application to the study of TYLCV acquisition and
transmission. European Journal of Plant Pathology
104: 189-194.
49
Références
bibliographiques
Barro N., Tiendrébéogo F.,
Traoré A.S., Konaté G., 2007. Principales viroses des
plantes maraîchères au Burkina Faso : symptômes et quelques
aspects écologiques. Annal de Botanique d'Afrique de l'Ouest
4:1-12.
Barro N., 1994. Caractérisation
sérologique, biologique et aspects écologiques de quelques virus
infectant les plantes maraîchères au Burkina Faso. Thèse de
doctorat. Université de Ouagadougou, 171p.
Barro N., Konaté G., 1994.
Caractérisation biologique d'un isolat du virus de la
mosaïque du gombo et aspects écologiques de la maladie au Burkina
Faso. Sciences et Techniques, série sciences naturelles
21 (1): 6-18.
Bigarré L., Chazly M., Salah M., Ibrahim M.,
Padidam M., Nicole M., Peterschmitt M., Fauquet C. et Thouvenel J. C.,
2001. Characterization of a new Begomovirus from Egypt
infecting hollyhock (Althea rosea). European Journal of Plant
Pathology, 107, 701-711.
Borssum Waalkes J. Van , 1966. Malesian
Malvaceae revised. Blumea 14 (1) 1-151
Briddon R. et Stanley J., 2006. Subviral
agents associated with plant single-stranded DNA viruses. Virology,
344, 198-210.
Brown J. K., 1994. The status of Bemisia
tabaci Genn. as a pest and vector in world agroecosystems. FAO Plant
Protection Bulletin 42: 3-32.
Brown J. K., 1997. The Biology and Molecular
Epidemiology of the Geminiviridae Subgroup III.pp 125 - 133, in
Stacey, G. and Keen, N. T. 1997.Plant- Microbe Interactions.2:
220 pp.
Brown J. K., Bird J., 1992.
Whitefly-transmitted geminiviruses and associated disorders in the Americas and
Caribbean Basin. Plant Disease 76: 220-225.
Brown J. K., Idris A. M., Torres-Jerez I., Banks G.
K., Wyatt S. D., 2001. The core region of the coat protein gene is
highly useful for establishing the provisional identification and
classification of Begomoviruses. Archives of virology
146: 1581-1598.
Brown J. K., Fauquet C. M., Briddon R. W., Zerbini M.,
Moriones E. et Navas- Castillo J., 2011. Family
Geminiviridae.Virus Taxonomy: Ninth Report of theInternational
Committee on Taxonomy of Viruses. pp. 351-373.
Brown J. K., Zerbini F. M., Navas-Castillo, J.,
Moriones E., Ramos-Sobrinho R., Silva J. C. F., Fiallo-Olivé E., Briddon
R. W., Hernández-Zepeda C., Idris A., Malathi V. G., Martin D. P.,
Rivera-Bustamante R., Ueda S. et Varsani A., 2015. Revision of
50
Références
bibliographiques
Begomovirus taxonomy based on pairwise sequence
comparisons. Archives of Virology, 160, 1593-1619.
Bull S. E., Briddon, R. W., Sserubombwe W. S., Ngugi
K., Markham P. G. et Stanley J., 2007. Infectivity, pseudo
recombination and mutagenesis of Kenyan cassava mosaic Begomoviruses.
Journal of General Virology, 88, 1624-1633.
Charrier A., 1983. Étude des ressources
génétiques du genre Abelmoschus Med. (Gombo). Rome. 61
p.
Clifford L. D., Anderson D. J., 2004. Food
limitation explains most clutch size variation in the Nazca booby. Journal
of Animal Ecology, 70, 539-545
Cohen S. et Antignus Y., 1994. Tomato yellow
leaf curl virus, a whitefly-borne geminivirus of tomatoes. Advances in
Disease Vector Research, 10, 259-288.
Czosnek, H., 2007. Interactions of Tomato Yellow
Leaf Curl Virus with its Whitefly Vector, Tomato Yellow Leaf Curl Virus
Disease, 157-170.
De Barro P. J., Liu S. S., Boykin L. M., Dinsdale A. B.,
2011. Bemisia tabaci: a statement of species status.
Annual Review of Entomology 56, 1-19
De Castro A. P., 2005. Evaluation of breeding
tomato lines partially resistant to Tomato yellow leaf curl virus -
Sardinia and Tomato yellow leaf curl virus derived from
Lycopersicon chilense. Canadian Journal of Plant Pathology
27, 268-275.
De Lannoy G., 2001.Abelmoschus esculentus
(L.) Moench. In : Agriculture en Afrique Tropicale. Direction
Générale de la coopération Internationale (DGCI). pp
478-484.
Delatte H., Martin D. P., Naze F., Golbach R. W.,
Reynaud B., Peterschmitt M. et Lett J. M. 2005b. South West Indian
Ocean islands tomato begomovirus populations represent a new major monopartite
begomovirus group. J Gen Virol 86, 1533-1542.
Delatte H., Duyck P. F., Triboire A.,
David P., Becker N., Bonato O., Reynaud B., 2007. Differential
invasion success among biotypes: case of Bemisia tabaci.
Biological Invasions, 11, 1059-1070.
Dkhil B., Denden M., 2010. Effects of heat
stress on the germination, protein degradation and mineral content in seeds of
okra (Abelmoschus esculentus L.). Continental Journal of
Biological Sciences, 3, 51-62.
51
Références
bibliographiques
Doyle J.J., Doyle J.L. (1987). A rapid DNA
isolation procedure for small quantities of fresh leaf tissue.
Phytochemistry Bulletin 19: 11-15.
Duffy S. et Holmes E. C., 2009. Validation of
high rates of nucleotide substitution in geminiviruses: Phylogenetic evidence
from East African cassava mosaic viruses. Journal of General Virology,
90, 1539-1547.
Duncan J. M., Torrrance L., 1992. Techniques for
the Rapid Detection of Plant Pathogens. Blackwell Scientific
Publications. FAO, 1989, Production Year Book, 43.
Ekra, 2010. Étude comparée de
l'efficacité des extraits aqueux de neem (Azadirachta indica
J.) et de la feuille d'eucalyptus (Eucalyptus camaldulensis) dans
la lutte contre les insectes du gombo (Abelmoschus esculentus L.),
Mémoire d'Ingénieur d'agriculture. Institut National
Polytechnique Felix Houphouet-Boigny, pp 136-142
FAOSTAT, 2019.
http://faostat.fao.org/site/339/default.aspx,
consulté le 18/10/2019 à 14h
FAOSTAT,2014.
http://faostat.fao.org/site/339/default.asp,
consulté le 11/12/2018 à 11h FAOSTAT, 2013.
http://faostat.fao.org/site/339/default.asp,
consulté le 14/011/2019 à 21h
Fauquet C. M., Bisaro D. M., Briddon R. W., Brown J.
K., Harrison B. D., Ribicki E. P., Stenger D. C., Stanley J., 2003.
Revision of taxonomic criteria for species demarcation in the family
Geminiviridae, and an updated list of Begomovirus species. Archives of
Virology 148: 405-421.
Fauquet C. M., Briddon R. W., Brown J. K., Moriones
E., Stanley J., Zerbini M., Zhou X., 2008. Geminivirus strain
demarcation and nomenclature. Archives of
Virology153: 783-821.
Fiallo-Olivé E., Martínez-Zubiaur Y.,
Moriones E. et Navas-Castillo J., 2012. A novel class of DNA
satellites associated with New World Begomoviruses. Virology,
426, 1-6.
Fondong V. N., 2013. Geminivirus protein
structure and function.Molecular Plant Pathology, 14,
635-649.
Fontenele R. S., Lamas N. S., Lacorte C., Lacerda A.
L. M., Varsani A. et Ribeiro S. G. , 2017. A novel geminivirus
identified in tomato and cleome plants sampled in Brazil. Virus
Research.
Gafni Y. et Epel B. L., 2002.The role of host
and viral proteins in intra- and intercellular tracking of geminiviruses.
Physiological and Molecular Plant Pathology, 60,
231-241.
52
Références
bibliographiques
Garcia-Diaz M. et Bebenek K., 2007. Multiple
functions of DNA polymerases. Critical reviews in plant sciences,
26, 105-122.
Ge L., Zhang J., Zhou X. et Li H., 2007.
Genetic structure and population variability of tomato yellow leaf
curl China virus. Journal of Virology, 81,
5902-5907.
Grubben G. J. H., Denton O.A., 2004. Ressources
végétales de l'Afrique tropicales 2 Légumes. In : PROTA,
p.737.
Guignard J. L., 1993. Botanique. Ed. Masson.
Paris, France. 276p.
Guo J., Lai X. P., Li J. X., Yue J. Q., Zhang S. Y., Li
Y. Y., Gao J. Y., Wang Z. R., Duan H. F. et Yang J. D., 2015. First
Report on Citrus Chlorotic Dwarf Associated Virus on Lemon in Dehong
Prefecture, Yunnan, China. Plant Disease, 99,
1287.
Gutierrez C., 1999. Geminivirus DNA replication.
Cellular and Molecular Life Sciences, 56, 313-
329.
Hall J. J., 1980. Resistance at the TM - 2 loci
in the tomato, to Tomato mosaic virus. Euphytica
(Netherlands) 29: 189 - 197.
Hamon S. et Charrier A., 1997. Les gombos.
In: L'amélioration des plantes tropicales. Centre de coopération
internationale en recherche agronomique pour le développement (CIRAD) et
Institut français de recherche scientifique pour le développement
en coopération (ORSTOM), Montpellier, France. pp. 313-333.
Hamon S., 1987. Organisation évolutive
du genre Abelmoschus (gombo) : co-adaptation et évolution de
deux espèces de gombo cultivées en Afrique de l'ouest (A.
esculentus et A. caillei).In: Orstom (Eds.), paris,
france,p.191.
Hamon S., 1988.Organisation évolutive
du genre Abelmoschus (gombo). Coadaptation et évolution de deux
espèces de gombo cultivées en Afrique de l'Ouest, A.
esculentus et A. cailli. Paris, France : ORSTOM, Travaux et
documents microédités n° 46. 191pp.
Hamon S., Sloten. D. H.
1995. Okra, Abelmoschus esculentus, A. Callei,
A.manihot.,A. moschatus (Malvaceae). In: Evolution of Crop
Plants, Edited by Smart 1. Simmonds N. W. London, England, 350-355.
Hanley-Bowdoin L., Bejarano E.R., Robertson D. et
Mansoor S., 2013.Geminiviruses: masters at redirecting and
reprogramming plant processes. Nature Reviews Microbiology,
11, 777- 788.
53
Références
bibliographiques
Hanley-Bowdoin L., Elmer J.S. et Rogers S.G., 1989.
Functional Expression of the Leftward Open Reading Frames of the A
Component of Tomato Golden Mosaic Virus in Transgenic Tobacco Plants. The
Plant Cell, 1, 1057-1067.
Hanley-Bowdoin L., Settlage S.B., Orozco B.M.,
Robertson D., Settlage S.B., Orozco B.M., Hanley-bowdoin L., Settlage S.B.,
Orozco B.M., Nagar S. et Robertson D. , 1999. Geminiviruses: models
for plant DNA replication, transcription, and cell cycle regulation.
Critical Reviews in Plant Sciences, 18, 71-106.
Harrison B.D. et Robinson D.J., 1999. Natural
genomic and antigenic variation in whitefly-transmitted geminiviruses
(Begomoviruses).Annual Review of Phytopathology, 37,
369-398.
Hirose K., Endo K., Hasegawa K., 2004. A
convenient synthesis of lepidimoide from okra mucilage and its growth-promoting
activity in hypocotyls. Carbohydrate polymers, 339,
919p.
https://www.tela-botanica.org/2009/11/article3354/,
consulté le 17/10/2019 à 13h
ICVT, 2012. Virus Taxonomy: Ninth Report of
the International Committee on Taxonomy of Virus. Elsevier Inc.
2001:471-477
Idris A. L. I. M. et Brown J. K., 2002.
Molecular Analysis of Cotton Leaf Curl Virus- Sudan Reveals an
Evolutionary History of Recombination. Virus Genes,
24, 249-256.
Idris, A. M. et Brown J. K., 2005. Evidence
for interspecific-recombination for three monopartite begomoviral genomes
associated with the tomato leaf curl disease from central Sudan Brief Report.
Archives of Virology, 150, 1003-1012.
Idris, A. M., Shahid M. S., Briddon R. W., Khan a J.,
Zhu J. K. et Brown J. K., 2011. An unusual alphasatellite associated
with monopartite Begomoviruses attenuates symptoms and reduces betasatellite
accumulation. Journal of General Virology, 92,
706-17.
Igor M. De Rosa, Jose M. K., Debora P., Carlo S. et
Fabrizio S., 2009.Morphological thermal and mechanical
characterization of okra (A. esculentus) fibres as potential
reinforcement in polymer composite. Polym Degrad Stabil,
2, 179-190.
Inoue-Nagata, A. K., Albuquerque L. C., Rocha W. B. et
Nagata T., 2004. A simple method for cloning the complete Begomovirus
genome using the bacteriophage phi29 DNA polymerase. Journal of Virological
Methods, 116, 209-211.
54
Références
bibliographiques
Isnard M., Granier M., Frutos R., Reynaud B. et
Peterschmitt M., 1998. Quasispecies nature of three maize streak virus
isolates obtained through different modes of selection from a population used
to assess response to infection of maize cultivars. Journal of General
Virology, 79, 3091-3099.
Iqbal M. M. Naeem U., Azi J. Afzal et M. Alam Khan,
2014. An overview of Cotton Leaf Curl Virus Disease, persistant
challeng for Cotton production, Bulgarian Journal of Agricultural
Science, 20 (2) 2014, 405-415
Jayaseelan C., Ramkumar R., Rahuman A. A., Perumal P.,
2013.Green synthesis of gold nanoparticles using seed aqueous extract
of Abelmoschus esculentus and its antifungal activity. Industrial
Crops and Products, 45, 423-429.
Jenkins G.M., Rambaut A., Pybus O.G. et Holmes E.C.,
2002. Rates of molecular evolution in RNA viruses: A quantitative
phylogenetic analysis. Journal of Molecular Evolution,
54, 156- 165.
Jeske H., 2009.Geminivirus.TT
Viruses - The Still Elusive Human Pathogens (ed. By E.-M. De Villiers),
185-226.
Jeske H., Lu, M. et Preiss W., 2001. DNA
forms indicate rolling circle and recombination-dependent replication of
Abutilon mosaic virus. The EMBO Journal, 20,
6158-6167.
Jiro H., Sawadogo M., Millogo J.,
2011.Caractérisations agro-morphologique et anatomique du gombo
du Yatenga et leur lien avec la nomenclature locale des variétés.
Sciences etNature 8, (1), 23 - 36.
Jones D.R., 2003. Plant viruses transmitted
by white flies. European Journal of Plant Pathology,
109, 195-219.
Jones R., 2009. Plant virus emergence and
evolution: origins, new encounter scenarios, factors driving emergence, effects
of changing world conditions, and prospects for control. Virus
Research, 141, 113-130.
Kennedy C., Hoornweg D., 2012. Mainstreaming
Urban Metabolism.Wiley online Library, 16,780-782.
https://doi.org/10.1111/j.1530-9290.2012.00548.x
Koechlin J., 1989.Les gombos
africains (Abelmoschusssp) : Étude de la diversité en
vue de l'amélioration. Thèse Doctorat, Institut National
Agronomique. Paris-Grignon, France, 180pp.
55
Références
bibliographiques
Kon T., Rojas M.R., Abdourhamane I.K. et Gilbertson
R.L., 2009. Roles and interactions of Begomoviruses and satellite DNAs
associated with okra leaf curl disease in Mali, West Africa. Journal of
General Virology, 90, 1001-1013.
Konaté G., Barro N., Fargette D. et Swanson
M.M., 1995. Occurrence of whitefly transmitted geminiviruses in crops
in Burkina Faso, and their serological detection and differentiation.Annals
of Applied Biology, 126, 121-130.
Koonin E. V. et Ilyina T. V., 1992.
Geminivirus replication proteins are related to prokaryotic plasmid
rolling circle DNA replication initiator proteins. Journal of General
Virology, 73, 2763-2766.
Krake L.R., Rezaian M.A. et Dry I.B., 1998.
Expression of the Tomato Leaf Curl Geminivirus C4 Gene Produces
Viruslike Symptoms in Transgenic Plants. Molecular Plant- Microbe
Interactions, 11, 413-417.
Krupovic M., Ravantti J.J. et Bamford D.H., 2009.
Geminiviruses: a tale of a plasmid becoming a virus. BMC
Evolutionary Biology, 9, 112.
Kumar S., Dagnoko S., Haougui A., Ratnadass A.,
2010. Okra (Abelmoschus spp.) in West and Central Africa:
Potential and progress on its improvement. African Journal of Agricultural
Research, 25, 3590-3598.
Lapointe A., 1987. Plantes
médicinales. Fiche Technique.161pp.
Legg J., Kumar P.L., Makeshkumar T., Tripathi L.,
Ferguson M., Kanju E., Ntawuruhunga P. et Cuellar W., 2014a. Cassava
Virus Diseases: Biology, Epidemiology, and Management. Advances in Virus
Research, 85-142.
Legg J., Somado E.A., Barker I., Beach L., Ceballos
H., Cuellar W., Elkhoury W., Gerling D., Helsen J., Hershey C., Jarvis A.,
Kulakow P., Kumar L., Lorenzen J., Lynam J., McMahon M., Maruthi G., Miano D.,
Mtunda K., Natwuruhunga P., Okogbenin E., Pezo P., Terry E., Thiele G., Thresh
M., Wadsworth J., Walsh S., Winter S., Tohme J. etFauquet C., 2014b.A
global alliance declaring war on cassava viruses in Africa.Food
Security, 6, 231-248.
Leke W.N., Sattar M.N., Ngane E.B., Ngeve J.M.,
Kvarnheden A. et Brown J.K., 2013. Molecular characterization of
Begomoviruses and DNA satellites associated with okra leaf curl disease in
Cameroon. Virus Research, 174, 116-125.
56
Références
bibliographiques
Leung W.-T.W., Busson F., Jardin C., 1968.
Food composition table for use in Africa.FAO, Rome, Italy. 306 pp
Liang P., Navarro B., Zhang Z., Wang H., Lu, M., Xiao
H., Wu Q., Zhou X., Di Serio F. et Li S., 2015.Identification and
characterization of a novel geminivirus with a monopartite genome infecting
apple trees. Journal of General Virology, 96,
2411-2420.
Lim L., Chai C., 2007. Performance of seven
okra accessions.Senior Officers Conference. Departement of Agriculture,
Sarawak.p.12.
Loconsole G., Saldarelli P., Doddapaneni H., Savino
V., Martelli G.P. et Saponari M., 2012. Identification of a
single-stranded DNA virus associated with citrus chlorotic dwarf disease, a new
member in the family Geminiviridae. Virology, 432,
162-172.
Lozano G., Trenado H.P., Fiallo-Olivé E.,
Chirinos D., Geraud-Pouey, Francis Briddon R.W. et Navas-castillo J., 2016.
Characterization of Non-coding DNA Satellites Associated with
Sweepoviruses (Genus Begomovirus, Geminiviridae) Definition of a Distinct Class
of Begomovirus-Associated Satellites. Frontiersin Microbiology,
7, 1-13.
Lu Q.-Y., Wu Z.-J., Xia Z.-S. et Xie L. H., 2015.
Complete genome sequence of a novel monopartite geminivirus identified
in mulberry. Archives of Virology, 160, 1-4.
Ma Y., Navarro B., Zhang Z., Lu M., Zhou X., Chi S.,
Serio F. Di et Li S., 2015. Identification and molecular
characterization of a novel monopartite geminivirus associated with mulberry
mosaic dwarf disease. Journal of General Virology,
96, 2421-2434.
Macquet A., 2007. Caractérisation de
mutants d'Arabidopsis thaliana affectés dans la
production de mucilage par les téguments de la graine. Thèse,
Agroparistech, pp 1-154
Mansoor S., Khan S.H., Bashir A., Saeed M., Zafar Y.,
Malik K., Briddon, R., Stanley, J. et Markham, P.G.,
1999.Identification of a Novel Circular Single- Stranded DNA
Associated with Cotton Leaf Curl Disease in Pakistan. Virology,
259, 190-199.
Marius C., Gerard V. et Antoine G., 1997. Le
gombo, Abelmosehus eseulentus (L.) Moench, une source possible de
phospholipides. Agronomie et Biotechnologies, Oléagineux, corps gras,
lipides. 4 (5), 389-392.
Martin F. W., Rhodes A. M., Manuel O., Diaz F.,
1981. Variation in Okra. Euphytica, 7, 30, 697705.
57
Références
bibliographiques
Martin F., 1982. Absorption, assimilation et
transport de l'azote inorganique chez le Pin noir d'Autriche et l'Aulne
glutineux. Influence des mycorhizes et des actinorhizes. Thèse de
doctorat Université de Nancy, 241p.
Matthews R. E. F., 1991. Transmission,
movement and host range. In: Plant Virology. Third Edition, Academic
Press, Inc. Harcourt Brace Jovanovich, Publishers, pp. 339-343.
Mivedor A. S., Pita, J. S., Adjata K. D., Duclercq, J.
et Gumedzoe, Y.M.D., 2017. Genetic Diversity of Tomato (Solanum
lycopersicum L.) Begomovirus in Togo. Agricultural Sciences,
8, 1402-1414.
https://doi.org/10.4236/as.2017.812100
Monci F., Sanchez-Campos S., Navas-Castillo J. et
Moriones E., 2002. A natural recombinant between the geminiviruses
Tomato yellow leaf curl Sardinia virus and Tomato yellow leaf curl virus
exhibits a novel pathogenic phenotype and is becoming prevalent in Spanish
populations. Virology, 303, 317-326.
Morilla G., Janssen D., García-Andrés
S., Moriones E., Cuadrado I.M. et Bejarano E. R., 2005. Pepper
(Capsicum annuum) is a dead-end host for Tomato yellow leaf curl
virus. Phytopathology, 95, 1089-1097.
Mugnier, J., 2008. Nouvelle flore
illustrée du Sénégal et des régions voisines.
N'Guessan K. P., Fargette D., Fauquet, C. et Thouvenel
J., 1992. Aspects off the epidemiology of okra leaf
curl virus in Côte d'lvoire. Tropical Pest Management,
38, 122-126.
Nacoulma O., 1996. Plantes médicinales
et pratiques médicales traditionnelles au Burkina Faso. Cas du plateau
central. Thèse d'état, Tome I. Université de
Ouagadougou. 261pp.
Nana R., 2005. Influence de la
fréquence et de la période d'arrosage sur l'économie
d'eau: adaptation physiologique du gombo (Abelmosehus eseulentus (L.)
Moench) en culture de contre saison. Mémoire de DEA,Université de
Ouagadougou, Burkina Faso, p.67.
Navas-Castillo J., Fiallo-Olivé E. et
Sanchez-Campos S., 2011. Emerging Virus Diseases Transmitted by
Whiteflies. Annual Review of Phytopathology, 49,
219-248.
Nzikou J. M., Mvoula T., Matouba E., Ouamba J. M.,
Kapseu C., Parmentier M., Desobry S., 2006. A study
on gumbo seed grown in Congo Brazzaville for its food and industrial
applications African, Journal of Biotechnology, 5
(24), 2469-2475.
Osborne L. S., 1988. The not so sweet
sweetpotato whitefly. Florida Foliage. 8-15.
Ouedraogo Z. A., 2009. Caractérisation
agromorphologique comparée de cinq variétés de Gombo
58
Références
bibliographiques
(Abelmoschus esculentus (L.) Moencb). Mémoire
d'Ingéniorat, Université Polytechnique de Bobo-Dioulasso, Burkina
Faso, p.61
Oyen L. P. N. et Lemmens R. H. M. J., 2002.
Ressources végétales de l'Afrique tropicale. PROTA
précurseur, ISBN 90-77114-03-3.
Padidam M., Beachy R.N., Fauquet C.M. 1995.
Tomato leaf curl geminivirus from India has a bipartite genome and coat protein
is not essential for infectivity. Journal of general virology
76: 25-35.
Papayiannis L.C., Katis N.I., Idris, A.M. et Brown
J.K., 2011. Identification of Weed Hosts of Tomato yellow leaf
curl virus in Cyprus.Plant Disease, 95,
120-125.
Patel V.P., Rojas M.R., Paplomatas, E.J. et Gilbertson
R.L., 1993. Cloning biologically active geminivirus DNA using PCR and
overlapping Primers. Nucleic Acids Research, 21,
1325- 1326.
Péréfarres F., Thébaud G.,
Lefeuvre P., Chiroleu F., Rimbaud L., Hoareau M., Reynaud B. et Lett J. M.,
2014. Frequency-dependent assistance as a way out of competitive
exclusion between two strains of an emerging virus. Proceedings of the
Royal Society B: Biological Sciences, 281, 1-9.
Perring T.M., Cooper A.D., Rodriguez R.J., Farrar
C.A., Billows T.S., 1991. Identification of a whitefly species by
genomic and behavioural studies. Science 259:
74-77.
Pico B., Diez M.J., Nuez F., 1996. Viral
disease causing the greatest economic losses to the tomato crop. II. The Tomato
yellow leaf curl virus a review. Scientia Horticulturae
67: 151 196.
Picó B., Díez M.J. et Nuez, F.,
1996.Viral diseases causing the greatest economic losses to the tomato
crop. II. The Tomato yellow leaf curl virus. A review. Scientia
Horticulturae, 67, 151-196.
Polston J.E., Anderson P.L., 1997.The
emergence of whitefly transmitted geminiviruses in tomato in the Western
Hemisphere. Plant Disease 81: 1358-1369.
Polston, J.E. et Lapidot, M., 2007.Management
of Tomato yellow leaf curl virus: US and Israel Perspectives BT -
Tomato Yellow Leaf Curl Virus Disease: Management, Molecular Biology,
Breeding for Resistance. Tomato Yellow Leaf Curl Virus Disease,
pp. 251-262.
Preiss W. et Jeske H., 2003. Multitasking in
replication is common among geminiviruses. Journal of Virology,
77, 2972-2980.
59
Références
bibliographiques
Programme PIP, 2008. Itinéraire
technique, gombo (Abelmoschus esculentus).
www.coleacp.org.
Rohwer J. G., 2012. Guide des plantes tropicales. In :
Delachaux et Niestlé (Eds.), p.266.
Rojas M. R., Hagen C., Lucas W.J. et Gilbertson R. L.,
2005. Exploiting chinks in the plant's armor: evolution and emergence
of geminiviruses. Annual Review of Phytopathology,
43, 361-394.
Rojas M. R., Jiang H., Salati R., Xoconostle-ca B.,
Lucas W. J. et Gilbertson, R. L., 2001. Functional Analysis of
Proteins Involved in Movement of the Monopartite Begomovirus , Tomato Yellow
Leaf Curl Virus. Virology, 291, 110-125.
Sánchez-Campos S., Navas-Castillo J., Camero
R., Soria C., Díaz J. A. et Moriones E., 1999. Displacement of
Tomato Yellow Leaf Curl Virus (TYLCV) -Sr by TYLCV-Is in Tomato Epidemics in
Spain. Virology, 89, 1039-1043.
Sanderfoot A. A., Ingham D .J. et Lazarowitz S. C.,
1996. Vira1 Movement Protein as a Nuclear Shuttle: The Geminivirus BR1
Movement Protein Contains Domains Essential for lnteraction with BL1 and
Nuclear Localization. Plant Physiology, 110,
23-33.
Saunders K. et Stanley J., 1999. A
nanovirus-like DNA component associated with yellow vein disease of Ageratum
conyzoides: evidence for interfamilial recombination between plant DNA viruses.
Virology, 264.
Sawadogo M., Balma D., Nana R., Sumda M. K. T. L. R.,
2009. Diversité agro-morphologique et commercial du gombo
(Abelmoschus esculentus L.) à Ouagadougou et ses environs.
Int. J. Biol. Chem. Sci. 3 (2): 326-336.
Sawadogo M., Balma D., Zombre G., 2006.
Expression de différents écotypes de gombo (A.
esculentus (L.)) au déficit hydrique intervenant pendant la
boutonnisation et la floraison. BASE, Biotechnologie, Agronomie,
Société, Environnement.10 (1): 43-54.
Sayed Sartaj Sohrab, Esam I., Azhar, Mohammad A. Kamal,
P.S. Bhattacharya , D. Rana, 2014. Genetic variability of Cotton leaf
curl betasatellite in Northern India, Saudi Journal of Biological Sciences,
21, 626-631.
http://dx.doi.org/10.1016/j.sjbs.2014.11.006
Séka K, Ouattara A, Assiri K. P., Kra KD,
Hoareau M., Lefeuvre P., Atta Diallo H., Lett J. M., 2016. First
reports of Cotton leaf curl Gezira virus and Okra yellow crinkle virus
associated with okra leaf curl disease in Côte d'Ivoire. New Disease
Reports 34, 8.
http://dx.doi.org/10.5197/j.2044-0588.2016.034.008
60
Références
bibliographiques
Shamsul A., Arifuzzaman K., 2007. Chemical
Analysis of Okra Bast Fiber (Abelmoschus esculentus) and Its
Physico-chemical Properties. Journal of textile and Apparel, Technology and
management.,5 (4).
Sheu S. C., Lai M. H., 2012. Composition
analysis and immuno-modulatory effect of okra (Abelmoschus
esculentusL.) extract. Food chemistry, 134 (4),
1906-1911.
Shih S. L., Green S. K., Tsai W. S., Lee, L. M. et
Levasseur V., 2007. First report of a distinct Begomovirus associated
with okra yellow crinkle disease in Mali. Plant Pathology,
56, 718.
Shih, S. L., Kumar, S., Tsai, W. S., Lee, L. M. et Green,
S. K., 2009. Complete nucleotide sequences of okra isolates of Cotton
leaf curl Gezira virus and their associated DNA- b from Niger. Archives of
Virology, 154, 369-372.
Siemonsma J. S., Hamon S., 2004- Abelmoschus
esculentus (L.) Moench. In: Ressources végétales de
l'Afrique Tropicale 2. Fondation PROTA.Wageningen. Pays-Bas, p.25-30.
Siemonsma J. S., 1982. West African okra:
morphological and cytological indications for theexistence of a natural
amphiploid of Abelmoschus esculentus (L.) Moench and
A.manihot (L.) Medikus.Euphytica,
31, 241-252.
Silva J. C. F., Carvalho T. F. M., Basso M. F.,
Deguchi M., Pereira W. A., Sobrinho R.R., Vidigal P. M. P., Brustolini O.J.B.,
Silva F .F., Dal-Bianco M., Fontes R. L. F., Santos A. A., Zerbini, F. M.,
Cerqueira F.R. et Fontes E. P. B., 2017.Geminivirus data warehouse: a
database enriched with machine learning approaches. BMC
Bioinformatics, 18, 240.
Spichiger R. E., Savolainen V. V., Figeat M.,
2000. Botanique systématique des plantes : Une approche
phylogénétique nouvelle des Angiospermes des régions
tempérées et tropicales. Première édition. 373 p
Swanson, M. M. et Harrison, B. D., 1993.
Serological relationships and epitope profile of an isolate of okra
leaves curl geminivirus from Africa and Middle East.
Biochimie, 75, 707-711.
Terrell E. E., Winters H. F., 1974. Changes
in scientific names for certain crop plants. Hortscience, 9
(4): 324, 325.
Thresh J. M., Otim-Nape G. W., 1994.
Strategies for controlling African cassava mosaic geminivirus.
Adv. Dis. VectorRes. 10: 215-236.
Tiendrébéogo F., 2006.
Identification sérologique et moléculaire de Begomovirus
au Burkina Faso. Mémoire de DEA, Université de Ouagadougou,
Burkina Faso, 54 p.
61
Références
bibliographiques
Tiendrébéogo F., 2010.
Caractérisation et aspects épidémiologiques des
Begomovirus infectant les plantes maraîchères et le manioc au
Burkina Faso. Thèse de doctorat. Université de Ouagadougou,
191p.
Tiendrébéogo F., Lefeuvre P., Hoareau
M., Villemot J., Konaté G., Traoré A. S., Barro N.,
Traoré, V.S., Reynaud, B., Traoré, O. et Lett, J.-M.,
2010. Molecular diversity of cotton leaf curl Gezira virus isolates
and their satellite associated with okra leaf curl disease in Burkina Faso.
Virology Journal, 7, 48.
Tiendrébéogo F.., Traoré E. V.
S., Barro N., Konaté G., Traoré A. et Traoré O., 2008.
Characterization of Pepper yellow vein Mali virus in Capsicum sp. In
Burkina Faso. Plant Pathology Journal, 7, 155-161.
Tshomba K. J., Esoma O. B., Muyambo Mu. E., Useni Si.
Y., Nyembo K. L., 2015. Facteurs influençant le profit de la
culture de gombo dans les conditions pédoclimatiques et
socio-économiques de Lubumbashi en RDC, International Journal of
Innovation and Applied Studies, 12, 820-830.
van der Walt E., Martin D. P., Varsani, A., Polston
J.E. et Rybicki E. P., 2008. Experimental observations of rapid Maize
streak virus evolution reveal a strand specific nucleotide substitution bias.
Virology Journal, 5, 104.
Vanitharani R., Chellappan P. et Fauquet C. M.,
2005. Geminiviruses and RNA silencing. Trends in Plant
Science, 10, 144-151.
Varma A., Malathi V. G., 2003. Emerging
geminivirus problems. A serious threat to crop production. Annals of
Applied Biology 142: 145-164.
Varsani A., Navas-Castillo J., Moriones E.,
Hernández-Zepeda C., Idris A., Brown J. K., Murilo Zerbini, F. et
Martin, D. P., 2014. Establishment of three new genera in the family
Geminiviridae: Becurtovirus, Eragrovirus and Turncurtovirus. Archives of
Virology, 159, 2193-2203.
Varsani A., Roumagnac P., Fuchs M., Moriones E., Idris
A., Briddon R.W., Zerbini F. M. et Martin, D. P., 2017.Capulavirus and
Grablovirus: two new genera in the family Geminiviridae. Archives of
Virology, 162, 1819-1831.
Wezel R., Van Liu H., Tien P., Stanley J. et Hong Y.,
2001. Gene of the Monopartite Geminivirus Tomato yellow leaf curl
virus -China Encodes a Pathogenicity Determinant That Is Localized in the
Nucleus. Molecular Plant-Microbe Interactions, 14,
1125-1128.
62
Références
bibliographiques
Wu J. X., Shang, H. L., Xie, Y., Shen, Q. T.et Zhou,
X. P., 2012. Monoclonal Antibodies Against the Whitefly-Transmitted
Tomato Yellow Leaf Curl Virus and Their Application in Virus Detection.
Journal of Integrative Agriculture, 11, 263-268.
Wyatt S. D., Brown J. K., 1996. Detection of
subgroup III geminivirus isolates in leaf extracts by degenerate primers and
polymerase chain reaction. Phytopathology 86:
1288-1293.
Yang Y., Sherwood T. A., Patte C. P., Hiebert E., Polston
J E., 2004. Use of Tomato yellow leaf curl virus (TYLCV) rep
gene sequences to engineer TYLCV resistance in tomato.Phytopathology
94: 490-496.
Zerbini F. M., Briddon R. W., Idris A., Martin D. P.,
Moriones E., Navas-Castillo J., Rivera-Bustamante R., Roumagnac P. et Varsani
A., 2017.ICTV Virus Taxonomy Profile: Geminiviridae. Journal of
General Virology, 98, 131-133.
Zhang W., Olson N. H., Baker T. S., Faulkner L.,
Agbandje-McKenna M., Boulton M. I., Davies J. W. et McKenna, R., 2001.
Structure of the Maize streak virus geminate particle.
Virology, 279, 471-7.
Zhou X., 2013.Advances in understanding
Begomovirus satellites.Annual review of phytopathology,
51, 357-81.
Zhou X., Liu Y., Calvert L., Munoz C., Otim-Nape G.
W., Robinson D. J. et Harrison B. D., 1997. Evidence that DNA-A of a
geminivirus associated with severe cassava mosaic disease in Uganda has arisen
by interspecific recombination. Journal of General Virology,
78, 2101- 2111.
ANNEXES
63
Annexes
Annexes
Annexe 1:Procédure d'extraction des ADNs
1. Préchauffer de l'eau au bain marie à
60°C
2. Peser 20 à 30 mg de feuilles séchées,
mettre dans les tubes eppendorf de 2ml, ajouter 2 à 3 billes
stériles, puis broyer àl'aide d'un broyeur bille.
3. Ajouter le tampon d'extraction au CTAB (2%
cétyltrimethyl ammonium bromide, 1,4 M NaCl, 0,2%, 20mM EDTA, 100Mm
Tris-HCl, PH=8) préchauffé à 60°C au bain-marie et
vortexer.
4. Incuber le mélange à 60°C au bain-marie
sous agitation douce (à chaque 10 minutes) pendant 60 min.
5. Ajouter un égal volume de CIA (Chloroform Isoamylic
Alcool) dans les proportions (24 :1) en agitant doucement pendant 5 min.
6. Centrifuger à 12000 rpm pendant 15 min à
température ambiante pour concentrer les phases. On obtient trois phases
: une phase aqueuse (en surface) contenant les acides nucléiques, une
phase organique (au fond du tube) contenant les protéines et lipides
dégradés ainsi que d'autres composés secondaires, et une
phase intermédiaire entre les deux contenant les déchets et
débris cellulaires ainsi que beaucoup de protéines
dégradées.
7. Transférer 800ul le surnageant dans un tube
eppendorf de 1,5 ml, ajouter 0,8 de volume d'isopropanol froid (-20°C) et
mélanger doucement pour précipiter les acides nucléiques.
Incuber à -20°c pendant 30 min si nécessaire et centrifuger
à 12000 rpm pendant 15 min,
8. jeter le surnageant et ajouter 500 ul d'éthanol 70%
pour le lavage, vortexer et laisser à température ambiante
pendant au moins 20 min.
9. Centrifuger à 12000 rpm pendant 15 min, vider
soigneusement le surnageant et faire sécher brièvement à
température ambiante pour éliminer l'éthanol.
10. Ré suspendre les acides nucléiques dans
100ul de H2O et conserver à 4°C.
64
Annexes
Annexe 2:Procédure de préparation du gel et
de la migration électrophorétique des ADN amplifiés par
PCR
1. Peser 1g d'agarose et mélanger avec 100 ml de
tampon TAE 0,5% (Tris base/Acétate acétique /EDTA 0.5M pH8) dans
une bouteille en verre avec couvercle et chauffer dans une microonde pendant 5
min pour fondre l'agarose et homogénéiser la solution.
2. Ajouter 4jil de bromure d'éthidium (BET) à
0,5 jig/ml à 60°C de la solution et homogénéiser
lentement. Le BET, agent intercalant de l'ADN, permet la visualisation des
bandes sous illumination UV. Il est hautement mutagène et doit
être manipulé avec beaucoup de précautions.
3. Couler le mélange sur une plaque
d'électrophorèse sur laquelle il faut placer des peignes avec une
épaisseur du gel de 3 à 5 mm.
4. Laisser polymériser le gel et retirer les
peignes.
5. Placer la plaque de gel dans la cuve
d'électrophorèse contenant du tampon TAE (0,5%) pouvant immerger
totalement le gel.
6. Ajouter 10% de tampon de charge (0,040%
bromophénol, 7% glycérol, 6 mM EDTA) aux produits PCR,
homogénéiser et déposer 12,5jil de ce mélange dans
les puits selon l'ordre d'étiquetage des échantillons. Le
marqueur de poids moléculaires (4 jil) est déposé dans le
premier puits. Les dépôts se font du côté de la
cathode qui est chargée négativement. L'ADN migrera en fonction
de la taille vers l'anode qui est chargée positivement.
7. Couvrir la cuve et démarrer la migration
électrophorétique à 100 volts pendant 35 à 40
min.
8. À la fin de la migration, retirer la plaque de gel
et déposer le gel sur un trans illuminateur pour la visualisation des
bandes sous illumination UV.
Annexe 3:Préparation des Mix et programmes PCR
? Composition du Mix de détection des virus
65
|
|
|
Annexes
|
|
|
|
|
|
Réactifs
|
Volume pour une réaction (ìl)
|
Conc. initiale
|
Conc. Finale
|
|
ADN
|
2
|
|
|
|
VD360
|
1
|
10 uM
|
0,5 uM
|
|
CD1266
|
1
|
10 uM
|
0,5 uM
|
|
H2O qsp20
|
13
|
|
|
|
Taq Polymerase (Hot firepol)
|
3
|
5U/ul
|
1U
|
|
Vol total mix
|
20
|
|
|
|
V' Composition du Mix de détection des
Betasatellites
|
|
|
Réactifs
|
Volume pour une réaction (ìl)
|
Conc. initiale
|
Conc.
finale
|
|
ADN
|
2
|
|
|
|
Beta 01
|
1
|
10 uM
|
0,5 uM
|
|
Beta 02
|
1
|
10 uM
|
0,5 uM
|
|
H2O qsp20
|
13
|
|
|
|
Taq Polymerase (Hot firepol)
|
3
|
5U/ul
|
1U
|
|
Vol total mix
|
20
|
|
|
|
V' Composition du Mix pour l'amplification des
isolats à séquencer
|
|
Réactifs
|
Volume pour une réaction (ìl)
|
Conc. initiale
|
Conc.
finale
|
|
ADN
|
2
|
|
|
|
Beta 01
|
1
|
10 uM
|
0,5 uM
|
|
Beta 02
|
1
|
10 uM
|
0,5 uM
|
|
H2O qsp20
|
13
|
|
|
|
Taq Polymerase (Hot firepol)
|
3
|
5U/ul
|
1U
|
|
Vol total mix
|
20
|
|
|
Annexes
Annexe 4: Composition de quelques tampons
utilisés
V' Composition du tampon d'extraction CTAB pour
100 ml
Éléments Composition
Volume/Masse
CétylTrimétyl Ammonium Bromide 2 % 2 g
(CTAB)
NaCl 1,4 M 8,18 g
â-mercapto-éthanol 0,2 % 200 ?l
Etylène diamine tétra acétate (EDTA) 20
mM 744,68 mg
Tris-HCl ou Trizma base pH 8 100 mM 10 ml
- 100
H2O
66
V' Composition du Chloroforme-alcool
isoamylique (CIA) pour 500 ul
Éléments Volume Rapport
Chloroforme 480 ul 24/1
Alcool isoamylique 20 ul
V' Composition du Tampon TAE 50X
utilisé pour les électrophorèses
Tris Base 242 g
Acide acétique .57,1 ml
EDTA solution mère 0,5 M pH 8 100 ml
Ajuster à pH 8,5 avant de mélanger 10 ml de TAE 50X
avec 990 ml d'eau distillée pour les électrophorèses.
| 


