I.2 Le processus de l'ECD biologiques
Avec le récent développement des
études à l'échelle génomique et protéomique,
les données biologiques se sont considérablement
multipliées et diversifiées. Ces données se
présentent alors sous la forme de séquences ou d'informations qui
proviennent de soumissions directes effectuées par les auteurs, par
l'intermédiaire d'Internet ou d'autres moyens électroniques
appropriés.
Nous trouvons alors des :
· des séquences et des données
d'expression de gènes (ADN, ARN, Protéines) ;
· des informations d'annotations (fonctions, ...)
de gènes et de protéines, etc.
Ces données biologiques sont stockées
dans des banques de données généralistes ou
spécialisées. On trouve alors des banques de données
:
· d'ADN : GenBank, DDBJ, EMBL, ...;
· d'ARN : RNAdatabases, QTL, ... ;
· de protéines : PIR ,Swiss-Prot, TrEMBL,
PDB, SCOP, ... ;
· de gènes : NCBI, dbEST, UniGene, Gis, ...
;
· ..etc.
L'ECD biologiques est un peu particulière
parce qu'en fait les données biologiques sont souvent dans un format
textuel (voir Figure 0.2) et ne se prêtent pas directement
à une exploitation par des systèmes classiques. Pour cela nous
présenterons ce processus dans son contexte biologique. Bien que le
processus général de l'ECD est particulièrement standard,
il présente néanmoins des traitements spécifiques d'une
étape à une autre et ce par rapport à la nature des
données traitées. Nous allons présenter une
démarche qui comprend les cinq étapes suivantes : la
sélection des données, le prétraitement, la
transformation, la fouille de données, l'évaluation et
l'interprétation des connaissances, en montrant d'une étape
à une autre, les particularités du processus d'ECD.
Chapitre I : L'extraction de connaissances à
partir de données biologiques - 8 -
(1) La sélection des
données
L'accès aux données se fait, dans notre
cas, à travers Internet via des interfaces spécialisées
pour le téléchargement d'échantillons expérimentaux
sélectionnés selon des critères fixés par
l'utilisateur. On utilise alors le système d'accès et de
récupération de données, ENTREZ de NCBI1.
Celui-ci permet d'interroger une collection de séquences disponibles
sous le format texte brut. Il permet aussi la recherche et l'extraction de
données relatives aux séquences nucléotidiques ou
protéiques, aux références bibliographiques
associées, et aux collections de séquences génomiques et
structurales, à l'aide d'une simple interrogation du serveur de NCBI
(National Center for Biotechnology Information).
Ensuite, ces données sont
récupérées sous la forme d'un ensemble de fichiers textes
bruts. À l'intérieur de ces fichiers, chaque séquence est
contenue dans une structure appelée « entrée »,
celle-ci comprend des informations liées à la séquence
considérée : structure, rôle biologique, organisme
d'origine...etc. Les données intéressantes sont stockées
au niveau de « champs » bien définis.
A l'intérieur de ces fichiers, la
donnée biologique peut être représentée sous
différents formats. Nous présentons les formats les plus
utilisés :
· FASTA (le format le plus simple)
· PIR (spécifique à la Bdd
PIR)
· STADEN
· Texte Brut.
Format FASTA
FASTA est sans doute le plus répandu et l'un
des plus pratiques. La séquence est décrite sous forme de lignes
de 80 caractères maximum, et précédée d'une ligne
de titre (nom, définition, ...) qui doit commencer par le
caractère ">". Plusieurs séquences peuvent être mises
dans un même fichier (voir Figure 1.1).
>entête de la séquence 1 Séquence
1
>entête de la séquence 2 Séquence
2
1
http://www.ncbi.nlm.nih.gov

Chapitre I : L'extraction de connaissances à partir de
données biologiques - 9 -
|
>gi|22777494|dbj|BAC13766.1| glutamate dehydrogenase
[Oceanobacillus iheyensis]
MVADKAADSSNVNQENMDVLNTTQTIIKSALDKLGYPEEVFELLKEPMRILTVRIPVRMDDGNV
LGGSHGRESATAKGVTIVLNEAAKKKGIDIKGARVVIQGFGNAGSFLAKFLHDAGAKVVAISDA
YGALYDPEGLDIDYLLDRRDSFGTVTKLFNNTISNDALFELDCDII
>EM|U03177|FL03177 FELINE LEUKEMIA VIRUS CLONE
FELV-69TTU3-16. AGATACAAGGAAGTTAGAGGCTAAAACAGGATATCTGTGGTTAAGCACCTG
GCCAGCAGTCTCCAGGCTCCCCA
|
Figure 1.1 : Exemple du format FASTA d'une
séquence protéique.
|
CODE
|
SIGNIFICATION
|
|
">"
|
Début de séquence.
|
|
gi|22777494
|
GenInfo Identifier
|
|
dbj|BAC13766.1|
|
Un enregistrement de séquence peut être
enregistré dans
plusieurs banques de données donc il y aura
un
identifiant dans la banque de données dans cet exemple c'est DNA
Database of Japan sous le n° dbj|BAC13766.1
|
|
BAC13766.1|
|
". 1" la séquence a été
révisée une fois
|
|
"glutamate dehydrogenase"
|
nom de la protéine
|
|
[Oceanobacillus iheyensis]
|
nom de l'organisme à partir duquel elle a
été déterminée.
|
Tableau 1.1 : Description du fichier FASTA de
l'exemple de la Figure 1.1.
Format STADEN

STADEN est le plus ancien et le plus simple. C'est une
suite de lettres par ligne terminée par un retour à la ligne (80
caractères maximum par ligne). Ce format n'autorise qu'une
séquence par fichier (voir Figure 1.2).
|
lovelace$ more zfmtsec
SESLRIIFAGTPDFAARHLDALLSSGHNVVGVFTQPDRPAGRGKKLMPSPVKVLAEEKGL
PVFQPVSLRPQENQQLVAELQADVMVVVAYGLILPKAVLEMPRLGCINVHGSLLPRWRGA
APIQRSLWAGDAETGVTIMQMDVGLDTGDMLYKLSCPITAEDTSGTLYDKLAELGPQGLI
TTLKQLADGTAKPEVQDETLVTYAEKLSKEEARIDWSLSAAQLERCIRAFNPWPMSWLEI
EGQPVKVWKASVIDTATNAAPGTILEANKQGIQVATGDGILNLLSLQPAGKKAMSAQDLL
NSRREWFVPGNRLV
|
Figure 1.2 : Exemple du format STADEN d'une
séquence protéique.
Format PIR
La première ligne commence par ">" suivi du
code de la séquence et du nom de la protéine. La deuxième
ligne contient une description textuelle de la séquence suivent
plusieurs lignes descriptives de la séquence elle-mêm,e et se
termine par une marque de fin de séquence "*" (voir Figure
1.3).

Chapitre I : L'extraction de connaissances
à partir de données biologiques -
10 -
|
>P1;1h7wa1
structureX:1h7wa1: 2 :A: 183 :A:undefined:undefined:
1.90:99.90
APVLSKDVADIESILALNPRTQSHAALHSTLAKKLDKKHWKRNPDKNCFHCEKLENNFD
DIKHTTLGERGALREACLKCADAPCQKSCPTHLDIKSFITSISNKNYYGAAKMIFSDNPLG
LTCGMVCPTSDLCVGGCNLYATEEGSINIGGLQQFASEVFKAMNIPQIRNPCLPSQEKMP*
|
Figure 1.3 : Exemple du format PIR d'une
séquence protéique.
|
CODE
|
SIGNIFICATION
|
|
|
|
">P1"
|
Début de la ligne
|
|
|
|
1h7wa1
|
Code de la protéine
|
|
|
|
structureX:1h7wa1: 2 :A: 183
:A:undefined:undefined: 1.90:99.90
|
description textuelle
séquence
|
de
|
la
|
|
"*".
|
Fin de la séquence
|
|
|
Tableau 1.2 : Description du fichier PIR de
l'exemple de la Figure 1.3.
Format Texte Brut
L'information biologique est décrite dans un
fichier au format texte brut ou chaque ligne a un sens bien précis,
comme par exemple, un code, un nom, etc. (voir Figure 1.4)

1: aac
aminoglycoside 2-N-acetyltransferase [Mycobacterium
tuberculosis CDC1551]
Other Aliases: MT0275
Annotation: NC_002755.2 (314424..314969,
complement)
GeneID: 923198
4270: tRNA-Pro-3
tRNA [Mycobacterium tuberculosis CDC1551] Annotation:
NC_002755.1 (4118796..4118872) GeneID: 922697
This record was discontinued.
Figure 1.4 : Exemple de fichier à
l'état brut de de la séquence génomique de
la souche MT
CDC1551 au format texte brut.
(2) Le prétraitement des
données
Le prétraitement consiste à nettoyer et
mettre en forme les données dans un formalisme approprié pour une
exploitation efficiente, i.e. l'élimination des données sans
importances particulières dans le processus d'ECD, et qui sont
susceptibles de réduire l'exactitude des modèles à
extraire. Ceci commence par un nettoyage des fichiers
Chapitre I : L'extraction de connaissances
à partir de données biologiques -
11 -
par enlèvement des lignes inutiles, des termes
ou morceaux de texte, tels que n° ligne, caractères spéciaux
inutiles. La Figure 1.5 montre un morceau de séquence de
gène nettoyé, et la Figure 1.6, montre le
résultat final de cette étape.

1: aac
aminoglycoside 2-N-acetyltransferase [Mycobaterium
Tuberculosis CDC1551] GeneID: 923198
2: accD
acetyl-CoA carboxylase, carboxyl transferase, beta
subunit [Mycobaterium Tuberculosis CDC1551]
GeneID: 926242
Figure 1.5 : Morceau de la séquence
génomique nettoyée, de la souche Mt CDC1551.

aac |aminoglycoside 2-N-acetyltransferase | Mycobaterium
Tuberculosis CDC1551 | 923198
accD | acetyl-CoA carboxylase, carboxyl transferase,
beta subunit | Mycobaterium Tuberculosis CDC1551 | 926242
Figure 1.6 : Morceau de la séquence
génomique mise en forme, de la souche Mt CDC1551.
(3) La transformation des
données
Cette étape consiste à transformer les
données et les convertir en données appropriées (voir
Figure 1.6), pour exploitation. Ce sera une transformation vers un
formalisme base de données (attribut, valeur), à partir des
descripteurs possibles qui peuvent être dégagées à
cette étape. Ces descripteurs ou attributs vont aider à «
binariser » les entités dégagées et serviront ainsi
à alimenter une base de données.

aac |aminoglycoside 2-N-acetyltransferase | Mycobaterium
Tuberculosis CDC1551
| 923198
accD | acetyl-CoA carboxylase, carboxyl transferase, beta
subunit | Mycobaterium Tuberculosis CDC1551 | 926242
aceA-1 | isocitrate lyase |
Mycobaterium Tuberculosis CDC1551 | 923830
Figure 1.7 : Morceau de la séquence
génomique structurée, de la souche Mt CDC1551.
|
Séquence génomique
structurée
|
|
code_gene
|
nom_gene
|
id_gene
|
|
aac
|
aminoglycoside 2-N-
acetyltransferase
|
923198
|
|
accD
|
acetyl-CoA carboxylase, carboxyl transferase, beta
subunit
|
926242
|
|
aceA-1
|
isocitrate lyase
|
923830
|
Chapitre I : L'extraction de connaissances à
partir de données biologiques - 12 -
(4) La fouille de données
C'est le coeur du processus d'ECD biologiques.
L'extraction de connaissances est faite à partir de cette étape.
Elle consiste à dégager un ensemble de connaissances brutes
à partir des données prétraitées. Un exemple est la
recherche d'associations.
(5) Evaluation et interprétation des
connaissances
Dans la plupart du temps, les connaissances extraites
au terme de la précédente étape ne sont pas toutes
profitables. En effet, il est difficile d'avoir directement des connaissances
valides et utilisables par l'utilisateur humain, le Data Miner. Il existe, pour
la plupart des techniques de fouille de données, des méthodes
d'évaluation des modèles ou motifs extraits. Ces méthodes
peuvent aussi aider à corriger les modèles, et à les
ajuster aux données.
Selon le degré d'exactitude des connaissances
retournées par ces méthodes, l'expert du domaine décide
d'arrêter le processus d'ECD ou au contraire le reprendre à partir
d'une étape antérieure (le processus est itératif)
jusqu'à ce que les connaissances obtenues soient nouvelles,
interprétables, valides et utiles au Data Miner. Ce dernier peut les
utiliser directement ou les incorporer dans un système de gestion de
connaissances.
La Figure 1.8, ci-dessous montre l'aspect
itératif du processus, i.e. la possibilité de retourner à
n'importe quelle étape afin d'obtenir des connaissances de
qualité.
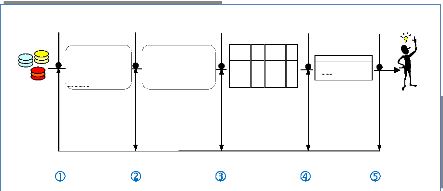
Banques de Données Biologiques (NCBI,
...)
Selection Prétraitement Transformation
Données biologiques cibles (séquence
génomique, séquence protéique,..)
1: aac
aminoglycoside 2-N-acetyltransferase [Mycobacterium tuberculosis
CDC1551]
Other Aliases: MT0275
Annotation: NC_002755.2 (314424..314969, complement) GeneID:
923198
Données Nettoyées et mises en
forme
accD | acetyl-CoA carboxylase, carboxyl transferase, beta
subunit | Mycobaterium Tuberculosis CDC1551 | 926242
aac |aminoglycoside 2-N-acetyltransferase | Mycobaterium
Tuberculosis CDC1551 | 923198
CDC1551 aac aminoglyco side 2-N-acetyltransf erase
Données Structurées
souche
Code gene
Nom gene
9231
98
Id gene
Fouille de données
aac -> ackA (75.0, 100.0) ackA -> aac (75.0,
100.0)
Motifs
Evaluation, interpretation
Connaissances
Figure 1.8 : Processus d'ECD
Biologiques.
Chapitre I : L'extraction de connaissances
à partir de données biologiques -
13 -
| 


