|
UNIVERSITE MONTPELLIER II
SCIENCES ET TECHNIQUES DU
LANGUEDOC
THESE
pour obtenir le grade
de
DOCTEUR DE L'UNIVERSITE MONTPELLIER II
Discipline : Biostatistique
Formation Doctorale :
Biostatistique
Ecole Doctorale : Information Structures Systèmes
présentée et soutenue
publiquement
par
Ibnou DIENG
le 24 janvier 2007
Titre :
Prediction de l'interaction genotype ×
environnement par
linearisation et regression PLS-mixte
JURY
M. Robert SABATIER Directeur de Thèse
M. Eric GOZ'E Codirecteur de Thèse
M. Alain CHARCOSSET Rapporteur
M. Jean-Jacques DAUDIN Rapporteur
M. Gilles DUCHARME Examinateur
Mme Christèle ROBERT-GRANIER Examinateur
Résumé: Ce travail porte sur la
pr'ediction de l'interaction g'enotype × environnement et est appliqu'e au
contexte sah'elien. Après un tour d'horizon des principales m'ethodes
d'analyse de la litt'erature, nous proposons la m'ethode APLAT. Le rendement de
g'enotypes pr'edit a` l'aide de covariables d'environnement par un
modèle de simulation de cultures est d'evelopp'e en s'erie de Taylor a`
l'ordre 1 au voisinage du vecteur de paramètres d'un g'enotype de
r'ef'erence. Nous nous ramenons alors approximativement a` un modèle
lin'eaire o`u la matrice des r'egresseurs est remplac'ee par la matrice des
d'eriv'ees partielles par rapport aux paramètres. Le nombre important de
paramètres vari'etaux g'en'eralement constat'e chez les modèles
de simulation de cultures conduit a` un nombre important de r'egresseurs; d'o`u
une estimation par r'egression Partial Least Squares (PLS).
Par la suite, nous proposons APLAT-mixte, une extension de APLAT. Pour ce
modèle mixte, nous maintenons le rendement des g'enotypes lin'earis'e
dans la partie fixe auquel s'ajoute un effet al'eatoire de l'environnement,
responsable d'interactions G×E dont il faut estimer la variance. Nous
introduisons a` cet effet la technique PLS-Mixte pour estimer les composantes
de variance dans un modèle o`u il y a plus de r'egresseurs que
d'observations. L'algorithme it'eratif propos'e, qui consiste a` imbriquer la
r'egression PLS dans l'algorithme Expectation Maximization
(EM), est fond'e sur les m'ethodes de vraisemblance Maximum
Likelihood (ML) et Restricted Maximum Likelihood
(REML).
Table des matières
|
1
2
|
Introduction générale
1.1 Problématique
1.2 Présentation des données de l'étude
1.2.1 L'essai pluriannuel
1.2.2 L'essai multilocal
Les méthodes classiques d'analyse des interactions
G×E
2.1 Le modèle d'analyse de variance a` deux
facteurs
2.1.1 Le modèle
2.1.2 Illustration avec les données de l'essai multilocal
. . .
2.2 La méthode de régression conjointe
2.2.1 Le modèle
2.2.2 Illustration avec les données de l'essai multilocal
. . .
2.3 La méthode AMMI
2.3.1 Le modèle
|
4
4
11
12
14
18
22
22
23
26
26
27
28
28
|
2.3.2 Illustration avec les données de l'essai
multilocal . . . . 31
2.4 La régression factorielle 33
2.4.1 Le modèle 33
2.4.2 Illustration avec les données de l'essai multilocal
. . . 40
2.5 Un modèle de simulation de cultures : SarraH 42
2.6 Limites des méthodes classiques d'étude des
interactions G×E 46
3 La méthode APLAT 48
3.1 La régression Partial least squares 48
3.2 La méthode APLAT : linéarisation autour d'un
témoin . . . 52
3.2.1 Le modèle proposé 52
3.2.2 Illustration avec les données de l'essai pluriannuel
. . 58
3.2.3 Illustration avec les données de l'essai multilocal
. . . 64
3.2.4 Conclusion 64
4 La méthode APLAT-mixte 67
4.1 Le modèle mixte 69
4.2 La régression PLS sur un modèle de variance
connue 73
4.3 La méthode PLS-Mixte 74
4.3.1 La méthode PLS-Mixte sur un modèle a`
effets aléatoires
indépendants de variances homogènes 75
4.3.2 La méthode PLS-Mixte sur un modèle a`
effets aléatoires
corrélés de variances
hétérogènes 86
5 Conclusion g'en'erale 104
R'ef'erences cit'ees 110
Annexes 120
A. Modèle de R'egression factorielle 120
B. Article au C.R. Biologie 124
Chapitre 1
Introduction générale
1.1 Problématique
Au Sahel, les pr'ecipitations annuelles ont diminu'e de
l'ordre de 20 a` 30 % dans la dernière moiti'e du 20e
siècle (Baterburry et Warren, 2001) et leur d'eficit y demeure la plus
forte contrainte a` l'agriculture (Tucker, 1991).
Dans de telles conditions, les paysans, qui constituent la
majorit'e de la population, parviennent difficilement a` g'en'erer des revenus
r'eguliers tout en g'erant durablement les ressources naturelles.
L'agriculture, qui est pourtant le principal moteur du d'eveloppement
'economique et social du Sahel, ne peut ainsi jouer pleinement son
ràole.
Dans ce cadre difficile, le ràole des d'ecideurs
('Etats, ONG, Organismes de coop'eration, Recherche) est de r'epondre a` la
demande sociale, principalement »la promotion d'une agriculture
productive, diversifi'ee, durable [...]». Ce sont les termes employ'es
dans le Cadre strat'egique de s'ecurit'e alimentaire (CSSA), le document de
r'ef'erence en matière de s'ecurit'e alimentaire du Comit'e permanent
inter-'etats de lutte contre la s'echeresse au Sahel (CILSS).
Ce cadre stratégique qui vise aussi
»l'amélioration durable des conditions d'accès des groupes
et zones vulnérables a` l'alimentation», a ététraduit
en programmes de sécuritéalimentaire pour la plupart des pays
sahéliens (CILSS, 2000). Ce document est venu confirmer le mandat
déjàattribuéau Centre d'étude régional pour
l'amélioration de l'adaptation a` la sécheresse (CERAAS) par le
Réseau de recherche sur la résistance a` la sécheresse
(R3S) du CILSS.
Le CERAAS est un laboratoire national de recherche a` vocation
régionale sous double tutelle. C'est un laboratoire de l'Institut
sénégalais de recherches agricoles (ISRA), qui a la mission
d'exécuter le programme national de recherche sur l'adaptation des
plantes a` la sécheresse. Il est aussi une Base Centre du Conseil ouest
et centre africain pour la recherche et le développement agricole
(CORAF), chargéde conduire les recherches sur la thématique de
l'adaptation des plantes a` la sécheresse et celle de la création
variétale.
Le CERAAS conduit des recherches suivant quatre axes principaux
:
1. la compréhension de la réponse des plantes;
2. la modélisation du fonctionnement des plantes;
3. l'amélioration de la méthodologie de la
sélection
4. l'amélioration des systèmes de culture pour une
meilleure adaptation a` la sécheresse.
Au troisième axe, l'objectif est d'identifier et de
sélectionner du matériel végétal mieux
adaptéa` la sécheresse, stabilisant ainsi le déficit
alimentaire dans les pays des régions sèches. Pour atteindre cet
objectif, les activités de recherche visent a` fournir des solutions
techniques pour réduire l'effet dépressif de la sécheresse
sur les productions agricoles. Ces solutions consistent a` proposer des
méthodes de sélection, de suivi des cultures et des
itinéraires techniques tenant compte du milieu ciblé, qui est
soumis a` la contrainte hydrique.
La s'election porte sur des caractères ph'enologiques,
physiologiques et mol'eculaires permettant une production am'elior'ee en
conditions de d'eficit hydrique par rapport aux cultivars g'en'eralement
vulgaris'es. Elle est valid'ee par des essais en plein champ, r'ealis'es dans
des conditions qui reflètent la variabilit'e du milieu auquel les
vari'et'es sont destin'ees. Ces essais peuvent avoir lieu la même ann'ee,
sur plusieurs endroits (essai multilocal) ou sur plusieurs ann'ees, au
même endroit (essai pluriannuel) ou sur plusieurs ann'ees, sur plusieurs
endroits. Par la suite, nous d'esignerons tous ces types d'essais sous le nom
g'en'erique d'essai multienvironnement et emploierons ce terme chaque fois
qu'il n'y aura pas d'ambigu·ýt'es et que nous voulons nommer un
ensemble d'essais.
Dans cette zone du Sahel a` fort risque climatique, il est
souvent constat'e une variabilit'e de l''ecart entre le rendement des
g'enotypes lors de ces essais multienvironnements de s'election vari'etale.
Cette variabilit'e est connue des s'electionneurs sous le nom d'interaction
g'enotype × environnement (G×E) a`
laquelle nous nous int'eressons dans ce travail.
Un cas particulier d'interactions 'etant un changement de
classement direct des g'enotypes. Si trois g'enotypes A, B et
C sont test'es sur plusieurs environnements, nous sommes en
pr'esence d'interactions G×E si pour le premier
environnement, les g'enotypes se classent A-B-C selon leurs
rendements et pour le deuxième environnement, ils se classent par
exemple B-A-C. Dans ce cas et même dans le cas plus
g'en'eral o`u les diff'erences entre g'enotypes dependent de l'environnement,
il sera difficile pour un environnement cible o`u les g'enotypes n'ont pas
encore 'et'e test'es, de pr'edire le meilleur g'enotype.
Dune manière g'en'erale, les essais multienvironnements
ne peuvent être assez pr'ecis. En effet, en dehors d'une ou de deux
vari'et'es t'emoin g'en'eralement reconduites dune ann'ee a` lautre, chaque
vari'et'e n'est vue que deux a` cinq ans. Au regard de la forte interaction
g'enotype × ann'ee (G×A), ces deux
a` cinq ans sont un 'echantillon de taille trop faible. Le s'electionneur
doit
souvent extrapoler a` partir de ce nombre d'années
faible ou d'un essai multi-local o`u l'interaction génotype x
lieu (GxL) ne reproduit qu'imparfaitement
l'interaction GxA.
Les interactions GxE gênent donc la
sélection variétale et constituent un obstacle aux
recommandations éventuellement formulées aux paysans pour
l'adoption de cultivars adaptés a` leurs milieux. Une solution est de
modéliser ces interactions GxE dans le but de les
prédire pour une situation nouvelle en fonction de variables
environnementales dont on connaàýt la valeur (ex : nature et
profondeur du sol) ou la loi de probabilité(ex : précipitations)
(Piepho, Denis et van Eeuwijk, 1998).
Nous proposons alors dans ce travail, une méthode de
modélisation des interactions GxE, qui permette de
tenir compte de l'impact aléatoire de l'environnement induit
principalement au Sahel par une variabilitéclimatique, pour une
meilleure prédiction de la réponse des variétés.
Plusieurs méthodes d'analyse des interactions ont
étéproposées dans la littérature et sont
exposées au chapitre 2. Ces méthodes peuvent être
rangées, a` notre sens, en deux catégories : celles qui utilisent
les caractéristiques des environnements et celles qui ne les utilisent
pas. Pour ces dernières, dont nous pouvons citer la méthode
Additive Main effects and Multiplicative Interactions (AMMI)
ainsi que la régression conjointe, la critique majeure est qu'elles ne
tiennent pas compte justement des environnements cibles pour y prédire
le rendement des génotypes. Ce n'est pas adaptédans cette zone du
Sahel, car comme nous avions annoncé, les interactions
GxE y sont la conséquence principalement de la grande
variabilitéclimatique des environnements. Mais ce qui gêne en
réalité, c'est la présence de l'interaction
GxA qui est imprévisible, au contraire de l'interaction
GxL. Talbot (1997) a établi que l'interaction
GxAxL est plus importante que l'interaction
GxA, ellemême plus importante que l'interaction
GxL. C'est certainement le cas au Sahel, o`u la
variabilitéclimatique interannuelle est forte. En effet, s'il pouvait
y être possible, pour chaque lieu, de pr'evoir les
conditions climatiques d'une ann'ee sur l'autre, il suffirait de mener un essai
multilocal sur un ensemble de lieux repr'esentatifs et avec la m'ethode AMMI
par exemple, pouvoir pr'edire avec assez de pr'ecision le rendement des
cultures. Mais les importantes variations climatiques d'une ann'ee sur l'autre
empêchent cette pr'ediction sans passer par la prise en compte des
conditions du milieu.
Parmi les m'ethodes qui utilisent les caract'eristiques
environnementales, figure la r'egression factorielle, dont une limite est
qu'elle suppose une action lin'eaire des environnements sur le rendement. Dans
le contexte sah'elien, cette m'ethode utilise le modèle d'analyse de
variance a` deux facteurs, g'enotype et environnement, o`u les interactions
G×E sont expliqu'ees par des covariables climatiques
mesur'ees souvent au pas de temps d'ecadaire voire journalier sur chacun des
environnements et des covariables mesur'ees sur chacun des g'enotypes. En
g'en'eral, les variables climatiques mesur'ees sur les environnements sont
très nombreuses (s'eries temporelles) et la prise en compte de
l'ensemble d'entre elles par cette m'ethode est impossible.
Les modèles de simulation de cultures sont aussi
utilis'es comme m'ethode de pr'ediction de rendement des cultures tenant compte
de l'environnement. Ces modèles ont certes l'avantage d'être plus
r'ealistes et considèrent le rendement d'un g'enotype dans un
environnement particulier comme une fonction non lin'eaire des
paramètres du g'enotype et des caract'eristiques de l'environnement. Ils
pr'esentent cependant l'inconv'enient de ne pas être applicables a` tout
g'enotype. En effet, les paramètres de tels modèles de simulation
de cultures ne sont pour la plupart connus que pour un petit nombre de
g'enotypes, car leur 'evaluation demande une exp'erimentation sp'ecifique et
des mesures coàuteuses.
La premi`ere méthode proposée : APLAT.
Lors d'essais multienviron-
nements, figure g'en'eralement un
g'enotype de r'ef'erence dont les paramètres
sont bien connus, dans le but de comparer sa performance aux
autres g'enotypes. Tenant compte de l'information souvent disponible pour ce
g'enotype de r'ef'erence, nous proposons notre première m'ethode
d'estimation qui consiste a` lin'eariser le rendement des g'enotypes pr'edit
par un modèle de simulation de cultures autour du vecteur de
paramètres de ce g'enotype de r'ef'erence. Le but 'etant d'estimer les
paramètres de ces g'enotypes a` l'aide des r'esultats d'essais
multienvironnements, sans refaire le travail de »param'etrisation» en
station exp'erimentale n'ecessaire a` l'estimation de ceux du t'emoin. Cela
per-met de se ramener approximativement a` un modèle lin'eaire o`u la
matrice des variables explicatives est remplac'ee par la matrice des d'eriv'ees
partielles (sensibilit'es) par rapport aux paramètres.
Cette m'ethode appel'ee Approximation par lin'earisation
autour d'un t'emoin (APLAT) est d'ecrite au chapitre 3. Pour estimer ainsi la
performance de tout g'enotype i dans un environnement
j, nous adjoignons a` la performance de ce g'enotype i
pr'edite par un modèle de simulation de cultures pour
l'environnement j lin'earis'ee autour du vecteur de
paramètres du t'emoin, un biais de ce modèle qui ne d'epend que
de l'environnement et une erreur al'eatoire r'esiduelle. Pour que cette
m'ethode ait un int'erêt, il faut que les interactions
G×E soient bien reproduites par le modèle de
simulation de culture, et que la m'ethode d'estimation supporte les abandons de
g'enotypes au cours du temps comme cela se pratique habituellement. C'est
pourquoi, elle a 'et'e test'ee sur les donn'ees d'un essai pluriannuel men'e
sur la station exp'erimentale du CERAAS au S'en'egal o`u tous les g'enotypes
n''etaient pas observ'es tous les ans.
Pour la plupart des modèles de simulations de cultures,
il existe un nombre important de paramètres pour les g'enotypes. Ces
paramètres, g'en'eralement connus pour le g'enotype de r'ef'erence, et
que nous cherchons a` r'eestimer pour tout nouveau g'enotype, conduisent a` un
nombre important de r'egresseurs
pour notre méthode proposée. Ils ont
étéestimés a` l'aide la régression Partial
least square (PLS).
La deuxième méthode proposée :
APLAT-Mixte. Au chapitre 4, nous étendons la méthode
APLAT au cas d'essais a` plusieurs composantes de variance. Pour cette
méthode, nous estimons qu'un modèle de simulation de cultures ne
permet pas de prendre en compte totalement l'effet aléatoire des
interactions G×E, même si nous pouvons concevoir
qu'il le permette pour une grande part. Nous rajoutons alors au modèle
APLAT un effet résiduel de l'environnement et un effet des interactions
G×E aléatoires, dont il faudra estimer les
composantes de variance.
Le recours a` un modèle de simulation de culture
induit, nous l'avons vu, un nombre important de régresseurs. Comme il
s'agit également d'estimer la variance de l'effet de l'environnement et
de l'effet des interactions G×E supposés
aléatoires, nous nous retrouvons avec un modèle avec un nombre
important de régresseurs et des composantes de variance. Nous proposons
donc dans ce chapitre, une méthode originale d'estimation des
paramètres fixes et des composantes de variance dans un modèle
o`u le nombre de régresseurs est important par rapport aux observations.
Cette méthode d'estimation, dénommée APLAT-Mixte, se fait
par le principe d'une méthode combinée de réduction de
dimension et de modèle mixte, que nous avons appeléPLSMixte.
Considérant d'une part les algorithmes itératifs
d'estimation des paramètres inconnus dans le cadre du modèle
mixte, et d'autre part les techniques de réduction de dimension, nous
proposons d'imbriquer la régression PLS dans l'algorithme EM. Puisque
nos données d'interaction G×E
s'appréhendent a` l'aide d'un modèle o`u les erreurs
aléatoires sont corrélées, nous appliquons dans un premier
temps cette technique a` des données de NIRS (Near infrared
spectroscopy) avec des erreurs indépendantes, o`u nous ne nous occupons
que
de la double contrainte de la dimension du modèle et de
la pr'esence des composantes de variance. Par la suite, nous nous int'eressons
a` r'esoudre le problème suppl'ementaire des erreurs corr'el'ees qui
r'esultent des donn'ees de notre probl'ematique d'interaction
G×E.
1.2 Présentation des données de
l'étude
Les m'ethodes qui ont 'et'e propos'ees ici se sont appuy'ees
sur des r'esultats d'essais multienvironnements. Ces exp'erimentations
vari'etales, sur quoi finalement repose toute cette 'evaluation du comportement
des g'enotypes en rapport avec les caract'eristiques du milieu, sont la base de
la s'election vari'etale. Les objectifs de tels essais peuvent être
divers et d'ependent des questions auxquelles l'exp'erimentateur veut apporter
des r'eponses. Mais l'objectif principal est la comparaison des performances,
souvent le rendement, de diff'erents g'enotypes au regard de diff'erentes
conditions environnementales.
Plusieurs g'enotypes sont en g'en'eral test'es dans plusieurs
environnements et ces derniers sont choisis avec un souci qu'ils soient aussi
divers que possible. Le but recherch'e est de couvrir la quasi totalit'e des
types d'environnements susceptibles de recevoir les g'enotypes. Ce but est
difficile a` atteindre au Sahel tant sont variables, nous l'avons dit, les
conditions environnementales d'une ann'ee a` l'autre.
Les donn'ees utilis'ees dans le cadre de cette 'etude
proviennent de deux types d'exp'erimentations. D'une part, nous nous sommes
servi des r'esultats d'un essai pluriannuel, men'e a` la station exp'erimentale
du CERAAS, de 1994 a` 1998. D'autre part, un essai multilocal a 'et'e mis en
place durant l'hivernage 2005, sur 11 localit'es. L'hivernage d'esigne la
saison des pluies dans les r'egions tropicales. Au S'en'egal, il a lieu chaque
ann'ee entre mai et octobre et sa dur'ee varie de trois a` six mois selon un
gradient Nord-Sud.
FIG. 1.1 - Localisation du CERAAS et de la station
exp'erimentale de Bambey au S'en'egal.
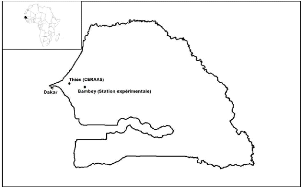
Toutes ces exp'erimentations qui ont donc 'et'e men'ees au
S'en'egal, ont concern'e
l'arachide (Arachis hypogaea L.),
qui y demeure la principale culture de rente.
1.2.1 L'essai pluriannuel
Cet essai vari'etal n'a concern'e qu'un seul site, la station
exp'erimentale du CERAAS situ'ee a` Bambey (14°42 N et 16°28 O). Il
est dit pluriannuel et a 'et'e men'e de 1994 a` 1998 (figure 1.1).
Cet essai a 'et'e conduit avec 26 g'enotypes a` cycle de
d'eveloppement de 90 jours. Les rendements moyens par g'enotype et par ann'ee
sont pr'esent'es dans le tableau 1.1. Tous les g'enotypes ne sont pas observ'es
toutes les ann'ees, ce qui est habituel pour les programmes de s'election
vari'etale. En effet, le s'electionneur a le loisir au cours de tels
programmes, d'enlever certains g'enotypes s'il est av'er'e qu'ils donnent de
faibles productions après une ou deux ann'ees d'exp'erimentation. De
même, au cours d'un même programme,
le s'electionneur peut tout aussi bien faire entrer de nouveaux
g'enotypes, toujours dans le but de les tester.
|
G'enotype
|
|
1994
|
|
1995
|
|
1996
|
1997
|
1998
|
Moyenne
|
|
55-113
|
|
|
1
|
337,4
|
1
|
114,9
|
351,1
|
937,9
|
935,3
|
|
55-140
|
|
|
1
|
236,5
|
|
908,2
|
|
931,8
|
1 025,5
|
|
55-15
|
|
|
1
|
234,4
|
1
|
052,6
|
|
|
1 143,5
|
|
55-16
|
|
|
1
|
268,4
|
|
937,7
|
|
|
1 103,1
|
|
55-17
|
|
|
1
|
246,8
|
|
991,6
|
|
|
1 119,2
|
|
55-437†
|
|
571,3
|
1
|
081,0
|
|
707,1
|
421,2
|
1 074,6
|
771,1
|
|
57-111
|
|
|
|
|
|
|
401,7
|
964,7
|
683,2
|
|
57-115
|
|
|
|
|
|
|
556,5
|
977,9
|
767,2
|
|
57-120
|
|
|
|
|
|
|
452,4
|
1064,0
|
758,2
|
|
57-123
|
|
|
|
|
|
|
596,7
|
985,7
|
791,2
|
|
57-125
|
|
|
|
|
|
|
651,6
|
1 110,8
|
881,2
|
|
57-126
|
|
|
|
|
|
|
665,2
|
1 232,3
|
948,8
|
|
57-14
|
|
|
|
|
|
649,4
|
244,1
|
|
446,8
|
|
FLEUR11
|
1
|
420,1
|
1
|
624,9
|
1
|
065,3
|
941,6
|
1 098,0
|
1 230,0
|
|
S-45
|
|
|
1
|
194,3
|
|
936,6
|
|
|
1 065,5
|
|
S-46
|
|
|
|
|
1
|
001,5
|
641,4
|
1 081,8
|
908,2
|
|
SR-1-1
|
|
657,8
|
|
|
|
|
|
|
657,8
|
|
SR-1-11
|
|
492,0
|
|
|
|
|
|
|
492,0
|
|
SR-1-12
|
|
437,7
|
|
|
|
|
|
|
437,7
|
|
SR-1-2
|
|
772,7
|
1
|
170,1
|
|
|
|
|
971,4
|
|
SR-1-22
|
|
692,2
|
1
|
113,3
|
|
934,7
|
474,4
|
|
803,7
|
|
SR-1-23
|
|
582,5
|
|
|
|
|
|
|
582,5
|
|
SR-1-4
|
|
563,0
|
1
|
205,6
|
|
870,9
|
|
|
879,8
|
|
SR-1-6
|
|
672,8
|
|
|
|
|
|
|
672,8
|
|
SR-1-9
|
|
572,7
|
|
|
|
|
|
|
572,7
|
|
US-83
|
|
957,7
|
1
|
307,0
|
|
|
|
|
1 132,4
|
|
Moyenne
|
|
699,4
|
1
|
251,7
|
|
930,9
|
533,2
|
1 041,8
|
888,8
|
† g'enotype de r'ef'erence
Donn'ees de l'essai pluriannuel de 26 g'enotypes
d'arachide a` la
TAB. 1.1 - station exp'erimentale de Bambey au S'en'egal
de 1994 a` 1998 (rendement en kg ha-').
Le g'enotype de r'ef'erence choisi est le
55-437, une vari'et'e de 90 jours; sa longueur de cycle est la
même que celles des autres g'enotypes. Les donn'ees de ce g'enotype sont
disponibles pour la dur'ee totale des exp'erimentations, ce qui autorise sa
comparaison aux autres vari'et'es utilis'ees dans cet essai.
Le tableau 1.1 r'evèle que les g'enotypes ont des
productions moyennes très vari'ees et certains font même plus que
doubler leur production d'une ann'ee sur l'autre. Par exemple, le rendement de
la vari'et'e 55-437 est de 1 081 kg ha-' en 1995 alors qu'il
n'est que de 571,3 kg ha-'en 1994, soit une augmentation d'un peu
moins de 90% entre deux ann'ees cons'ecutives.
Les productions moyennes par ann'ee sont aussi très
contrast'ees même si d'une ann'ee sur l'autre, nous n'avons pas les
mêmes g'enotypes. L'ann'ee 1995 (1251,7 kg ha-') est l'ann'ee
la plus productive et les g'enotypes qui ont les rendements les plus 'elev'es
sur l'ensemble des cinq ann'ees ont tous 'et'e observ'es cette ann'ee. Il
s'agit des g'enotypes FLEUR11 avec 1 230 kg ha-',
55-15 avec 1 143,5 kg ha-', US-83avec 1 132,4
kg ha-', etc. L'ann'ee 1997 (533,2 kg ha-') est l'ann'ee la moins
productive.
1.2.2 L'essai multilocal
Ces essais ont 'et'e men'es durant l'hivernage 2005 et visent
a` mesurer la production et la qualit'e des graines dans des conditions
environnementales diff'erenci'ees et connues afin de mod'eliser les
interactions G×E.
Pour ces essais, 6 g'enotypes (55-437,
FLEUR11, GC 8-35, JL24,
55-128, 55-33) ont 'et'e test'es sur 11
localit'es. Ces localit'es ont 'et'e choisies dans la principale zone de
culture de l'arachide au S'en'egal, appel'ee bassin arachidier. Ce bassin a
'et'e divis'e en trois zones a` l'int'erieur desquelles les essais ont 'et'e
implant'es : 3 essais dans la zone Nord, 4 dans la zone Centre dont un a` la
station exp'erimentale de Bambey et 4 dans la zone Sud dont un a` la station
exp'erimentale de Nioro (figure 1.2). Cet 'echantillonnage par stratification a
'et'e mis en place pour tenir compte de la variabilit'e climatique constat'ee
dans ce pays selon le gradient Nord-Sud.
Dans chaque localit'e, un dispositif en blocs complets
randomis'es avec quatre r'ep'etitions, soit 24 parcelles, a 'et'e adopt'e. La
parcelle 'el'ementaire est constitu'ee
FIG. 1.2 - Localisation des sites des essais multilocaux
dans le bassin arachidier au S'en'egal durant l'hivernage 2005.
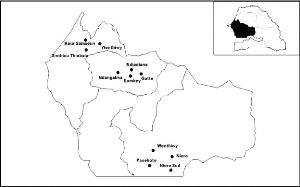
par 5 lignes de 6 m avec un 'ecartement de 50 cm entre les
lignes et de 15 cm entre les poquets. Les trois lignes centrales constituent la
parcelle utile a` l'int'erieur de laquelle les pr'elèvements de plantes
ont 'et'e r'ealis'es.
Le semis a 'et'e effectu'e dès l'installation effective
de l'hivernage sur des parcelles avec un pr'ec'edent cultural arachide ou mil.
Pour les conditions exp'erimentales, un labour superficiel et un piquetage ont
'et'e effectu'es et le semis fait a` la main quand le sol 'etait humide; deux
graines trait'ees au granox ont 'et'e sem'ees par poquet. De l'engrais NPK
6-20-10 a` raison de 150 kg ha-' a 'et'e appliqu'e a` la lev'ee. Un
d'emariage a` un pied par poquet a 'et'e fait vers 10 jours après le
semis et enfin, des sarclo-binages et des traitements phytosanitaires ont 'et'e
r'ealis'es a` la demande.
Les rendements en gousses et en fanes ont 'et'e mesur'es et un
suivi journalier des paramètres climatiques (temp'erature minimale et
maximale, humidit'e relative minimale et maximale, vitesse du vent, dur'ee
d'insolation) effectu'e. Les rendements moyens par g'enotype et par localit'e
sont pr'esent'es dans le tableau 1.2.
Les mesures des données climatiques pour les essais de
Nioro et de Bambey
sont celles des postes météorologiques de
ces stations. Ces mesures ont servi
aussi pour les sites autour de ces deux
localités a` l'exception des mesures
de pluviométrie, réputées plus variables
dans l'espace. La localitéde Meckhéétait
équipée d'une station météorologique portable.
Chacun des 11 sites a disposéd'un pluviomètre.
|
Localit'es
|
|
55-128
|
|
55-33
|
55-437
|
|
F11
|
GC-8-35
|
|
JL24
|
Moyenne
|
|
Keur Fary
|
|
263,0
|
|
209,3
|
189,0
|
|
392,5
|
|
281,7
|
|
239,3
|
|
262,5
|
|
Keur Samseun
|
|
103,7
|
|
91,5
|
119,2
|
|
260,9
|
|
141,8
|
|
151,7
|
|
144,8
|
|
Sinthiou Thiabala
|
|
317,9
|
|
198,1
|
244,0
|
|
459,9
|
|
275,8
|
|
554,2
|
|
341,7
|
|
Moyenne Nord
|
|
228,2
|
|
166,3
|
184,1
|
|
371,1
|
|
233,1
|
|
315,1
|
|
249,6
|
|
Bambey
|
|
248,1
|
|
196,6
|
298,7
|
|
472,3
|
|
134,0
|
|
350,8
|
|
283,4
|
|
Gatte
|
|
121,8
|
|
116,0
|
209,3
|
|
261,9
|
|
86,3
|
|
283,4
|
|
179,8
|
|
Ndangalma
|
|
162,3
|
|
202,1
|
223,9
|
|
237,5
|
|
183,6
|
|
313,8
|
|
220,5
|
|
Ndiadiane
|
|
72,6
|
|
107,4
|
234,9
|
|
236,9
|
|
159,9
|
|
368,7
|
|
196,7
|
|
Moyenne Centre
|
|
151,2
|
|
155,5
|
241,7
|
|
302,2
|
|
140,9
|
|
329,2
|
|
220,1
|
|
Nioro
|
|
928,0
|
|
866,3
|
807,0
|
|
856,5
|
|
614,1
|
|
780,7
|
|
808,8
|
|
Nioro Sud
|
1
|
593,6
|
1
|
013,0
|
670,7
|
1
|
115,8
|
1
|
115,8
|
1
|
255,4
|
1
|
127,4
|
|
Paoskoto
|
1
|
164,0
|
1
|
254,2
|
908,3
|
1
|
231,6
|
1
|
157,8
|
|
712,7
|
1
|
071,4
|
|
Winthewy
|
|
189,2
|
|
97,4
|
369,8
|
|
447,3
|
|
244,9
|
|
528,1
|
|
312,8
|
|
Moyenne Sud
|
|
968,7
|
|
807,7
|
688,9
|
|
912,8
|
|
783,1
|
|
819,2
|
|
830,1
|
|
Moyenne
|
|
469,5
|
|
395,6
|
388,6
|
|
543,0
|
|
399,6
|
|
503,5
|
|
450,0
|
Donn'ees de l'essai multilocal de 6 g'enotypes d'arachide
sur TAB. 1.2 - 11 localit'es au S'en'egal durant l'hivernage
2005 (rendement en
kg ha-').
L'examen des données du tableau 1.2
révèle une variabilitéde rendement des génotypes
selon les trois principales zones de l'étude : le Nord avec une
production moyenne de 249,6 kg ha-1 et le Centre avec une production
moyenne de 220,1 kg ha-1 s'opposent au Sud qui affiche la meilleure
production moyenne avec 830,1 kg ha-1. A l'intérieur des
zones, la variabilitérelative est plus importante au Nord o`u le
rendement du génotype le plus productif représente 123,2% de
celui du génotype le moins productif et au Centre o`u ce pourcentage est
de 133,6%, qu'au Sud o`u il passe a` 40,6%.
Par ailleurs, il est a` noter que le g'enotype FLEUR11
qui a la meilleure production moyenne pour les trois zones (543,0 kg
ha-1), n'est pas le meilleur partout. Il ne donne le meilleur
rendement qu'au Nord alors que le g'enotype GC-8-35 domine au
Centre et le g'enotype 55-128 au Sud. Ce constat renforce
l'id'ee que dans cette zone sah'elienne marqu'ee par une variabilit'e
environnementale importante, une recommandation pour le choix de g'enotypes
sp'ecifiques aux lieux est pr'ef'erable et plus pertinente qu'une
recommandation unique du g'enotype le meilleur en moyenne.
Chapitre 2
Les méthodes classiques
d'analyse des interactions G×E
Dans ce chapitre, nous parlerons des outils classiques
d'analyse des interactions G×E et présenterons les
modèles de simulation de cultures comme méthode alternative pour
prédire le rendement des cultures. Nous allons également
soumettre nos données a` ces différentes méthodes
présentées et évaluer comment elles prennent en compte les
éventuelles interactions décelées. Nous allons toutefois
appliquer ces différents modèles uniquement sur les
données de l'essai multilocal. En effet, la plupart des méthodes
classiques nécessitent des données complètes et seules le
sont celles de l'essai multilocal.
Les raisons de la présence des interactions
G×E peuvent être de deux ordres. De telles
interactions sont, d'une part, attendues en présence d'une large
variation des caractéristiques de résistance aux stress des
génotypes, le stress hydrique par exemple. D'autre part, en
présence, d'une large variation des environnements au niveau de ce
même stress. Mais généralement, c'est l'une et l'autre de
ces conditions qui les favorisent, même si au Sahel, la grande
variabilit'e climatique y s'evissant dont nous consid'erons
qu'elle caract'erise essentiellement les environnements, contribue pour une
large part a` la pr'esence de ces interactions.
Le terme g'enotype fait r'ef'erence a` un cultivar,
c'est-à-dire un mat'eriel g'en'etique qui peut être
homogène ou h'et'erogène et l'environnement a` un ensemble de
conditions climatiques, de types de sol et de pratiques culturales d'un essai
conduit dans un lieu donn'e, une ann'ee donn'ee (Annicchiarico, 2002).
Deux types d'interactions G×E sont a`
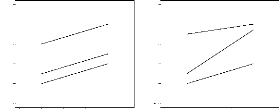
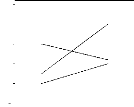
distinguer (figure 2.1). Les interactions sont dites quantitatives ou
noncrossovers, si les classements des g'enotypes entre les
diff'erents environnements sont conserv'es mais que l''ecart entre les
g'enotypes est modifi'e. Par contre, elles sont dites qualitatives ou
crossovers lorsque les classements sont invers'es (Baker,
1988; Baril, 1992).
Dans les essais multienvironnements, il peut être
envisag'e de s'electionner les g'enotypes de plus grande production moyenne sur
l'ensemble des environnements test'es ou de les choisir en fonction de leurs
performances selon les environnements. Pour cela, les informations issues de
ces exp'erimentations, sont 'etudi'ees afin d'être synth'etis'ees en
dissociant les effets du g'enotype, de l'environnement et des interactions
G×E au travers des modèles statistiques
(Brancourt-Hulmel, Biarnès-Dumoulin et Denis, 1997).
Plusieurs modèles des interactions
G×E ont donc 'et'e propos'es. Dans ce qui suit, nous en
ferons un tour d'horizon et en pr'esenterons les principaux : le modèle
d'analyse de variance a` deux facteurs, la r'egression conjointe, la m'ethode
AMMI, la r'egression factorielle et les modèles de simulation de
cultures.
Mais avant de pr'esenter ces diff'erentes m'ethodes fond'ees
principalement sur
le modèle d'analyse de variance, il est a`
remarquer qu'il est aussi possible
de concevoir, a` travers deux
statistiques descriptives, l''etude des interactions
FIG. 2.1 - Types d'interactions G×E pour trois
g'enotypes A, B et C. (1) : sans interactions; (2) : interactions
quantitatives; (3) : interactions qualitatives.
(1) (2)
1 2 3 4 5 6
environnement
1 2 3 4 5 6
environnement
(3)
1 2 3 4 5 6
renaement
renaement
2 9 4 5 0
A
B
C
A
B
C
A
B
C
C
A
B
renaement
2 9 4 5 0
A
B
C
B
C
A
environnement
G×E pour décrire le comportement des
génotypes sur un échantillon d'environnements.
Pour cela, la variabilitéintrinsèque du
génotype sur un ensemble d'environnements est étudiée a`
l'aide de la variance environnementale S2 i
(Becker, 1981; Lin, Binns et Lefkovitch, 1986; Piepho, 1998).
L'écart a` la valeur moyenne des performances du génotype, compte
tenu du nombre de milieux sur lequel il est testé, représente une
mesure de son instabilité. Cette variance environnementale est
estimée par
S2 i = XJ (Yij -
Yi.)2/(J - 1) j=1
o`u Yij est la réponse du
génotype i de l'environnement j,
Yi. la moyenne des réponses du
génotype i des différents environnements et
J le nombre d'environnements. Par la suite, l'opérateur
(.) désigne la moyenne sur l'indice qu'il remplace.
Quant a` l'écovalence variétale W i
2 (Becker, 1981; Becker et Léon, 1988), elle est mesurée
par la stabilitérelative du génotype et est estimée par
W i 2 = XJ (Yij - Yi. - Y.j +
Y..)2 j=1
C'est la somme des carrés des termes d'interaction
propres au génotype i. A la différence de
S2 i, la somme de
carrés W i 2 n'est pas divisée par les
degrés de liberté(ddl) correspondants.
Cependant, la liste des méthodes d'étude des
interactions G×E présentée dans ce chapitre
n'est pas exhaustive. D'autres méthodes, qui ne sont pas décrites
ici, existent par ailleurs :
- structuration de l'interaction (Denis et Vincourt, 1982)
- modèles multiplicatifs (Cornelius, Seyedsadr et Crossa,
1992; Crossa, Cornelius, Seyedsadr et Byrne, 1993; Crossa, Cornelius, Sayre et
Ortiz-Monasterio, 1995);
- application de l'analyse canonique (Seif, Evans et Balaam,
1979; Calinski, Czajkaet Kaczmarek, 1987);
- variantes des modèles de regression factorielle (Denis,
1988; van Eeuwijk 1992, 1995; van Eeuwijk, Denis et Kang, 1996);
- régression Partial Least Squares
(Aastveit et Martens 1986; Talbot et Wheelwright 1989; Vargas, Crossa,
Sayre, Reynolds, Ram`ýrez et Talbot, 1998);
- une méthode récente fondée sur l'approche
bayésienne (Theobald, Talbot et Nabugoomu, 2002).
2.1 Le mod`ele d'analyse de variance a` deux facteurs
2.1.1 Le mod`ele
Le mod`ele linéaire mixte généralement
considérésur les moyennes par génotype et par
environnement est le suivant
Yij = m + gi + Ej + (gE)ij + eij (2.1)
o`u Yijest la réponse du
génotype i de l'environnement
j, m la moyenne générale et
gi l'effet fixe du génotype i. L'effet
Ej de l'environnement j,
l'interaction (gE)ij et le terme d'erreur
eij sont supposés aléatoires, iid et indépendants
les uns des autres avec
E(Ej) =E[(gE)ij]
=E(eij) = 0 et Var(Ej) =
ó2E, Var[(gE)ij]
= ó2gE et
Var(eij) =
ó2e
o`u la fonctionE(
·) désigne
l'espérance et Var(
·) la variance.
Dans l'optique de prédire la performance des
génotypes dans les différents environnements
considérés, l'option qui consiste a` prendre les génotypes
comme fixes et les environnements comme aléatoires est argumentée
par Denis, Piepho et van Euwijk (1997). En effet, ces auteurs justifient ce
choix par le fait qu'il s'agit d'étudier un nombre fini de
génotypes, d'o`u l'effet génotype fixe. Au contraire, les
environnements ne sont pas considérés pour eux-mêmes, mais
en tant qu'échantillons dans une population plus vaste d'environnements
possibles auxquels les variétés sont destinées. Pour nous,
cela s'appliquera aux années plutôt qu'aux lieux.
Les effets principaux du génotype et de l'environnement
sont considérés par rapport a` la moyenne générale,
alors que le terme d'interaction du modèle représente la
variabilitédes performances du génotype avec l'environnement qui
n'est pas prise en compte dans les effets additifs du génotype et de
l'environnement.
D'après le modèle 2.1, les estimations des effets
sont, pour un dispositif équilibré:
bgi = Yi. - Y..
bEj = Y.j - Y..
[(gE)ij = Yij -
Yi. - Y.j + Y..
Dans l'estimation des termes du modèle qui portent
l'indice j, nous retrouvons Y.j.
Cette moyenne traduit le potentiel de l'environnement. Or l'environnement
étant fortement variable au Sahel, les termes en j ne
sont pas bien prévisibles a` moins de disposer d'un échantillon
de nombreux environnements qui fait généralement défaut.
Cependant si nous considérons la différence entre deux
variétés i et i',
l'imprévisibilitéde l'effet environnement Ej
disparaàýt lors de l'estimation de cette
différence, si le dispositif est complet. En effet, il viendra :
Yij - Yi'j = gi - gi' + (gE)ij -
(gE)i'j
Par contre, le problème demeure pour les interactions
qu'il faudra modéliser afin de prédire plus finement la
différence des performances des génotypes.
2.1.2 Illustration avec les données de l'essai
multilocal
Avec le modèle d'analyse de variance a` deux facteurs,
génotype et environ-
nement, appliquéaux données de
l'essai multilocal, nous sommes intéressés
tout premièrement a` tester la significativit'e des
interactions G×E. Dans ce cas, les deux effets principaux
sont consid'er'es comme 'etant fixes.
Nous rappelons, que les donn'ees proviennent d'un r'eseau
d'essais vari'etaux effectu'es au S'en'egal durant l'hivernage 2005 (tableau
1.2, page 16). Six g'enotypes d'arachide ont 'et'e test'es sur 11 sites dans le
bassin arachidier s'en'egalais qui est la r'egion principale de production de
cette l'egumineuse.
Le tableau 2.1 fournit les r'esultats de l'analyse de variance a`
deux facteurs appliqu'ee a` ces donn'ees.
Effet d.l Somme Carr'e Statistique F Niveau
de
de carr'es moyen signification
G'enotype 5 23 2765,0 46 553,0 2,42 0,0485
Environnement 10 8 100 921,7 3 810 092,2 42,1
0,0000
R'esidus 50 962 311,6 19 246,2
Tableau d'analyse de variance des donn'ees des essais
multilocaux TAB. 2.1 - de 6 g'enotypes d'arachide sur 11
localit'es au S'en'egal durant l'hivernage 2005.
En n'egligeant dans un premier temps l'interaction, nous
concluons qu'au seuil de 5%, les effets g'enotype et environnement sont
significatifs. A l'instar de Denis et Vincourt (1982), nous allons 'evaluer et
comparer l'ordre de grandeur des r'esidus et l'ordre de grandeur de l'effet
g'enotype. Si CMr est le carr'e moyen des r'esidus,
l'ordre de grandeur de ces r'esidus peut être estim'e par
vCMr ; et si CMg est le
carr'e moyen du facteur g'enotype, l'ordre de grandeur de l'effet g'enotype
peut être estim'e par ,/CMg -
CMr/J. Nous notons alors que l'ordre de grandeur des
r'esidus (138,7) est grand par rapport a` celui de l'effet g'enotype (15). Il
s'agira alors d'essayer de r'eduire ces r'esidus en ajoutant une interaction au
modèle additif. L'interaction peut être mise en 'evidence avec la
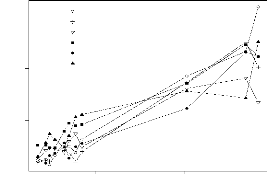
figure 2.2 o`u sont repr'esent'es les rendements des g'enotypes de l'essai
multilocal. Les rendements moyens par lieu sont rang'es par ordre croissant et
mis en abscisse. Nous constatons sur ce graphique un changement
de classement des variétés d'un site a` un autre.
Nous remarquons également que l'écart entre les génotypes
augmente avec la moyenne du lieu.
Variation des rendements des six g'enotypes de l'essai
multilo-
FIG. 2.2 - cal. En abscisse sont mis, par ordre
croissant, les performances moyennes des lieux.
200 400 600 800 1000
Rendement
500 1000 1500
55-128 55-33 55-437 F11 GC-8-35 JL24
Pour espérer formuler tout de même des
recommandations dans ce milieu très contrastépour l'adoption de
cultivars les mieux adaptés a` chaque environnement, la solution
consiste a` tenter de réduire la part imprévisible de ces
interactions en les modélisant; ce qui peut se faire a` travers
différentes méthodes que nous allons présenter
ci-dessous.
2.2 La methode de regression conjointe
2.2.1 Le modèle
Ce modèle a étéproposépour la
première fois par Yates et Cochran (1938). Il
a ensuite
étérepris par plusieurs autres auteurs dont les principaux sont
Fin-
lay et Wilkinson (1963), Eberhart et Russell (1966) et Perkins et Jinks
(1968).
Plutôt proposépour les essais multilocaux que
pour les essais pluriannuels, ce modèle pose l'effet des interactions
G×E comme une fonction linéaire de l'effet
Ej qui représente le potentiel du milieu j
pour les génotypes; une valeur positive de Ej
traduisant un potentiel élevétandis qu'une valeur
négative, un faible potentiel.
L'interaction est de la forme :
(gE)ij = ciEj + dij
o`u bci est le coefficient de la
régression pour le génotype i; cette pente
caractérise la sensibilitédifférentielle du
génotype i au milieu (Denis et Vin-court 1982). Le
terme dij désigne la déviation du
modèle, c'est-à-dire les interactions G×E
résiduelles.
Ces coefficients bci et la performance
moyenne des génotypes sont les paramètres d'intérêt
pour une analyse de stabilité. En effet, la performance d'un
génotype dans un lieu donnédépend de la performance
moyenne des génotypes du lieu et des interactions
G×E qui y sont espérées. Une valeur
largement positive de bci associée a` une performance
moyenne d'un site relativement importante, concourent a` caractériser ce
site comme étant favorable. De l'autre côté, un site est
considérécomme non favorable si la valeur de bci
associée a` la performance moyenne des génotypes de ce
site est négative.
Les coefficients bci sont obtenus facilement
:
|
bci =
|
X bEj
[(gE)ij
j
|
|
XbE2 j
j
|
Un cas particulier de ce modèle a 'et'e d'evelopp'e par
Tukey (1949) : les coefficients c s''ecrivent Kg et l'interaction se r'eduit a`
Kg E3.
2.2.2 Illustration avec les données de l'essai
multilocal
Les donn'ees sont celles du tableau 1.2 de la page 16. Le tableau
2.2 pr'esente les paramètres mb + bgi et 1 +
bci des diff'erents g'enotypes des essais.
|
G'enotype
|
mb + bgi
|
1 + bci
|
|
55-128
|
488,9
|
1,381
|
|
55-33
|
341,3
|
1,138
|
|
55-437
|
327,2
|
0,698
|
|
FLEUR11
|
636,0
|
0,963
|
|
GC-8-35
|
349,2
|
1,043
|
|
JL24
|
557,1
|
0,777
|
Paramètres estim'es par r'egression conjointe de 6
g'enotypes d'ara-
TAB. 2.2 - chide lors d'essais multilocaux au S'en'egal
durant l'hivernage 2005.
Le g'enotype 55-128 qui a la pente (1
+ bci) la plus 'elev'ee, est plus sensible aux variations du milieu
que la moyenne des g'enotypes des essais, suivi du g'enotype
55-33. En revanche, le g'enotype de r'ef'erence 55-437
et le g'enotype JL24 semblent moins affect'es que la
moyenne aux variations du milieu. Quant aux g'enotypes GC-8-35
et FLEUR11 avec des pentes proches de l'unit'e, ils
repr'esentent par leurs valeurs, de bonnes indications des variations du
milieu.
2.3 La méthode AMMI
2.3.1 Le modèle
La méthode AMMI, Additive main effect and
multiplicative interaction, a étéintroduite par Williams
(1952) et reprise par Gollob (1968), puis par Mandel (1961, 1971) et par Bradu
et Gabriel (1978). Elle fut développée a` l'origine pour les
domaines du social et de la physique et son application a` la recherche
agricole a étéproposée par Kempton (1984) et Zobel, Wright
et Gauch (1988). Mais il faut attendre Gauch (1992) pour qu'elle devienne
répandue. C'est une méthode assez générale et Gauch
et Zobel (1996) ont soulignéque son champ d'application potentiel va au
delàde l'étude
des méthodes d'interactions GxE.
Beaucoup d'autres auteurs ont étudiéles interactions
GxE a` l'aide de cette méthode :Vargas, Crossa, van
Eeu-
wijk, Ram`ýrez et Sayre (1999), Yan et Hunt (2001), Ebdon
et Gauch (2002a, 2002b), Gonz'alez, Crossa et Cornelius (2003a, 2003b), Gauch
(2006).
La méthode AMMI associe l'analyse de variance et
l'analyse en composantes principales (ACP). Sont d'abord estimés les
effets principaux des variétés et des environnements par une
analyse de variance du modèle additif, c'est-àdire du
modèle sans les interactions GxE. Ensuite, la partie
non additive du modèle est étudiée par une analyse en
composantes principales (Crossa, 1990).
L'interaction est décrite de cette façon
(gE)ij =
è1á1iâ1j
+
è2á2iâ2j
+ + èháhiâhj
c'est a` dire
Yij = m + gi + Ej + ( Xh
èkákiâkj) + eij
k=1
o`u èk est la valeur singulière
du ke axe (è2
k 'etant la valeur propre),
áki est le vecteur propre du
ie g'enotype pour le ke
axe, âkj est le vecteur propre du
je environnement pour le ke axe et
avec les contraintes
X Xá2
ki = Xâ2 kj = 1 et
Xákiák'i = âkjâk'j =
0
i j i j
Les paramètres du terme d'interaction sont donc
estim'es par d'ecomposition en valeur singulière (DVS) de la matrice des
r'esidus obtenus après ajustement des deux effets principaux.
Le nombre h de paramètres pour chaque
terme multiplicatif de l'interaction qui constitue le nombre d'axes principaux
peut être d'etermin'e soit par validation crois'ee (Gauch et Zobel, 1988;
Crossa, 1990, Piepho, 1994) o`u les r'ep'etitions sont tour a` tour retir'ees
et non les lieux, soit par des tests statistiques (Gollob, 1968; Cornelius,
1993; Piepho, 1995).
Le nombre d'axes principaux retenu est g'en'eralement compris
entre z'ero, on parle dans ce cas de AMMI-0 c'est-à-dire du
modèle additif et le minimum entre (I - 1) et
(J - 1), o`u I constitue le nombre de
g'enotypes et J le nombre d'environnements. Le modèle
complet (AMMI-F, F faisant r'ef'erence a` full pour
full model), avec tous les axes principaux, fournit une
estimation parfaite. Mais g'en'eralement, lorsque les interactions
G×E sont significatives, les modèles avec un
(AMMI-1) ou deux (AMMI-2) axes principaux sont les plus utilis'es a` cause de
leur simplicit'e.
Les tests statistiques propos'es pour d'eterminer le nombre
optimal d'axes sont tous fond'es sur la statistique
t2
k/s2 o`u t2
k est l'estimation de la valeur singulière
èk obtenue par DVS et s2,
avec f degr'es de libert'e, est le carr'e moyen des r'esidus
du modèle additif divis'e par le nombre de r'ep'etitions par
environnement (Piepho, 1995).
Sous l'hypothèse nulle (H0 :
èk = 0), les statistiques de tests sont les suivantes :
1.
t2k/s2
suit une loi de Fisher a` (J +I-1-2k) et f
degrés de liberté(Gollob, 1968)
2. FGH1 =
gt2k/h1fs2
suit une loi de Fisher avec h1 et g
degrés de liberté, o`u h1 =
2v1u1/v2, g = 2 + 2(f -
2)v1/v2, v1 =
u22 + u21 + (f -
4)u1 et
v2 = (f -
2)u22 +
2u21 (Cornelius, 1993) ; u1
et u2 sont des approximations fournies par Cornelius
(1980).
3. FGH2 =
t2k/u1s2
est distribuée selon la loi de Fisher avec h2
et f degrés de liberté, o`u
h2 = 2u21/u22
(Cornelius, 1993).
En outre, une statistique de test FR, plus
simple a` calculer, a étéproposée par Cornelius et al.
(1992). Ce test utilise la somme des carrés résiduels
après ajustement du modèle AMMI avec q axes
principaux. Sachant que sous l'hypothèse nulle, la somme de
carrés résiduels est approximativement une variable de chi-deux,
la statistique suivante FR
|
[
FR = E E(Yij - Yi. - Y.j +
Y..)2 - i j
|
X q
k=1
|
?t2 ?
/f2s2 k
|
suit une distribution de Fisher avec
f2 = (J - 1 - q)(I - 1 - q) et f
degrés de liberté. La statistique FR
significative révèle qu'il y a au moins un axe principal
supplémentaire a` prendre en compte en plus des q
déjàutilisés.
Dans le cas des essais multienvironnements, il est
supposéque les erreurs du modèle sont indépendantes et
normalement distribuées avec des variances entre les environnements
homogènes. Même si l'indépendance des erreurs peut
être assurée par une randomisation des niveaux des facteurs et que
les erreurs peuvent être considérées comme gaussiennes, les
variances résiduelles sont généralement
hétérogènes d'un environnement a` l'autre au Sahel. Or, en
présence d'erreurs expérimentales
hétérogènes, Piepho(1995) a montréque la
statistique de test FR est plus robuste que
FGH1, FGH2 et celle de Gollob.
2.3.2 Illustration avec les données de l'essai
multilocal
Les données de l'essai multilocal (6 génotypes
et 11 sites) ont étésoumises a` la méthode AMMI dans le
but d'obtenir l'estimation des performances des génotypes dans les
différents environnements. Il est espéréque l'estimation
d'un génotype dans un environnement particulier soit plus précise
que la simple moyenne des performances de ce même génotype dans
les autres environnements o`u il a étéobservé.
Pour ces données, le nombre maximum d'axes principaux
a` retenir est égal a` cinq. Pour cela, cinq modèles (AMMI-0 a`
AMMI-4) vont être testés étape par étape a` l'aide
de la statistique de test FR. Si le modèle AMMI-1 est
significatif, c'est-à-dire la statistique associée au premier axe
principal est significative, le modèle AMMI-2 est alors testéa`
son tour et ainsi de suite jusqu'au modèle AMMI-q non
significatif o`u l'on devra s'arrêter. Le meilleur modèle est
alors le modèle AMMI-q.
Les valeurs singulières calculées sur les
données de l'essai multilocal sont égales a` 723,8 pour le
premier axe, 577,6 pour le second, 259,2 pour le troisième, 182,1 pour
le quatrième et 66,5 pour le cinquième tandis que le tableau 2.3
montre les vecteurs propres des facteurs pour les quatre premiers axes
principaux
Les résultats de la méthode AMMI sont
présentés au tableau 2.4. Le test pour le deuxième axe
principal est significatif, ce qui signifie qu'il y a au moins un axe
supplémentaire intéressant dont il faut tenir compte. Et comme le
test pour le troisième axe n'est pas significatif, nous adoptons, pour
ces données, le modèle AMMI-3. A la figure 2.3 sont
représentés les scores des génotypes et des environnements
du deuxième axe principal en fonction de ceux du premier. Ce graphique
double, est plus connu sous le nom de biplot (Kempton, 1984).
Sur ce graphique, un génotype proche de l'origine présente
Axe 1 Axe 2 Axe 3 Axe 4
Vecteurs propres des g'enotypes pour les 4
axes
|
55-128
|
18.0
|
-7.7
|
-5.5
|
1.8
|
|
55-33
|
6.0
|
11.1
|
-5.0
|
-5.0
|
|
55-437
|
-15.6
|
7.3
|
-5.6
|
-0.8
|
|
F11
|
-2.0
|
4.2
|
3.7
|
11.2
|
|
GC-8-35
|
3.8
|
2.8
|
12.6
|
-4.9
|
|
JL24
|
-10.2
|
-17.8
|
-0.2
|
-2.3
|
Vecteurs propres des environnements pour les 4
axes
|
Wenthiwy
|
-9.9
|
-4.5
|
3.2
|
3.3
|
|
Niorosud
|
18.5
|
-14.9
|
0.5
|
-0.4
|
|
Paoskoto
|
12.3
|
16.2
|
3.9
|
0.5
|
|
Nioro
|
2.6
|
3.1
|
-13.2
|
-0.2
|
|
Keur Samseun
|
-1.4
|
3.1
|
3.2
|
-0.1
|
|
Keur Fary
|
1.1
|
3.1
|
4.6
|
1.2
|
|
Sinthiou Thiabala
|
-3.4
|
-6.4
|
2.9
|
2.1
|
|
Ndiadiane
|
-7.5
|
-1.5
|
1.4
|
-6.1
|
|
Gatte
|
-4.9
|
0.2
|
-2.4
|
-1.1
|
|
Ndangalma
|
-3.3
|
0.8
|
-0.9
|
-7.6
|
TAB. 2.3 - Vecteurs propres des facteurs g'enotype et
environnement pour l'essai multilocal.
une faible interaction tandis qu'un genotype qui s'en eloigne est
au contraire interactif.
Comme avec la regression conjointe, les genotypes
55-33 et 55-128 presentent de fortes
interactions et les genotypes GC-8-35 et F11
semblent les moins interactifs c'est-à-dire les plus conformes
au comportement au comportement moyen de l'ensemble des genotypes. Mais au
contraire, cette analyse classe les genotypes JL24 et 55-437 parmi les
genotypes les plus interactifs alors qu'ils etaient classes parmi ceux qui
affichent les plus faibles interactions par la regression conjointe.
FIG. 2.3 - Scores des g'enotypes et des environnements
pour le deuxième axe en fonction de ceux du premier.

Score de l'axe 2
-15 -10 -5 0 5 10 15
Ndiadiane Gatte
Wenthiwy
55-128

Sinthiou Thiabala
Nioro Sud
JL24

55-437

Keur Samseun
Bambey
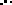
F11
Ndangalma
Keur Fary
Nioro
GC-8-35
55-33

Paoskoto
-15 -10 -5 0 5 10 15 20
Score de l'axe 1
2.4 La r'egression factorielle 2.4.1 Le modèle
C'est le modèle additif auquel trois séries de
termes de régression sont ajoutés (Denis, 1980). Ces
régresseurs sont des covariables décrivant les génotypes
(ex : poids des graines) et les environnements (ex : pluviométrie en
aoàut) et aussi l'interaction par les produits deux a` deux de ces
régresseurs.
Un modèle similaire de celui de régression
factorielle a étéutilisépar Freeman et Perkins (1971) qui
n'avaient considéréqu'une covariable associée au facteur
milieu non calculée sur les données. Wood (1976), lui, a
utiliséune combinaison linéaire de covariables
élémentaires liées au milieu.
|
Effet
|
d.l
|
|
Somme de carrés
|
|
Carrémoyen
|
Statistique F
|
Niveau de signification
|
|
Génotype
|
5
|
|
23 2765,0
|
|
46 553,0
|
2,42
|
0,0485
|
|
Environnement
|
10
|
8
|
100 921,7
|
3
|
810 092,2
|
42,1
|
0,0000
|
|
G×E
|
50
|
|
962 311,6
|
|
19 246,2
|
|
|
|
AMMI axe 1
|
14
|
|
523 953,0
|
|
37 425,2
|
3,1
|
0,003
|
|
AMMI axe 2
|
12
|
|
333 578,0
|
|
27 798,1
|
6,4
|
0,000
|
|
AMMI axe 3
|
10
|
|
67 186,2
|
|
6 718,62
|
2,5
|
0,057
|
|
AMMI axe 4
|
8
|
|
33 172,6
|
|
4 146,6
|
5,6
|
0,026
|
|
Résidus G×E
|
6
|
|
4 422,5
|
|
|
|
|
|
Total
|
65
|
|
929 600
|
|
|
|
|
TAB. 2.4 - Tableau d'analyse des données de
l'essai multilocal avec la méthode AMMI.
Nous allons pr'esenter le modèle de r'egression
factorielle de deux manières diff'erentes. Dans la pr'esentation
matricielle, la plus simple, le but est d'expliquer la matrice des observations
Y de dimension I ×J a` partir de deux
matrices de covariables associ'ees aux deux facteurs 'etudi'es. Dans la
pr'esentation indicielle, la plus g'en'erale, les observations Y
sont dans un vecteur de longueur IJ.
Nous pouvons disposer en g'en'eral d'un tableau X
de p covariables associ'ees
aux g'enotypes et d'un
tableau Z de q covariables associ'ees aux
environne-
|
ments. Les matrices X et Z
s''ecrivent dans ce cas :
x1 1 x2 1
· · · xp 1 z1 1
? x1 2 x2 2
· · · xp 2 ? ? z1
2
X= Z=
? ? ? ? ?
? ? ? ? ?
... ... ... ????? ...
x1I x2 I
··· xp I z1 J
|
z21
z2 2
...
z2 J
|
···
· · ·
· · ·
|
zq 1
zq 2 ?
... ?????
zq J
|
|
La matrice X est de dimension I
× p et Z de dimension J ×
q. Par souci de simplicit'e dans la pr'esentation, nous ne
consid'erons alors que le cas d'une unique observation par g'enotype pour un
environnement donn'e, qui peut être une moyenne ou une moyenne ajust'ee
sur le dispositif de cet environnement. Les matrices X et
Z sont consid'er'ees par la suite centr'ees.
Alors le modèle de r'egression factorielle peut se
pr'esenter sous forme matricielle, plus commode a` manipuler
2Z') +
XZ'
+g3Z' + X['
Y = 1Im1' J
+ (g1 + Xg2)1'
J + 1I(I' 1
+uEi' 3 + e (2.2)
- Y de dimension I x J est la
variable r'eponse
- m est la moyenne g'en'erale
- (g1 +
Xg2)1'
efficients des covariables 1 a` p dans la
r'egression de l'effet g'enotype et (g1 1
+uEI' J est l'effet principal du g'enotype
o`u (g2 est le vecteur des coles I r'esidus
de cette r'egression.
- 1I(i'
2Z') est l'effet principal de
l'environnement d'ecompos'e de la même
facon
- XZ' est la partie de l'interaction
expliqu'ee par le produit des deux covariables X et
Z
-g3Z' est la partie de l'interaction
expliqu'ee par la covariable Z, une fois tenu compte de
l'explication fournie par la covariable X
- Xi'3 est la partie de
l'interaction expliqu'ee par la covariable X, une fois tenu
compte de l'explication fournie par la covariable Z
Ce modèle 'etant lin'eaire, les proc'edures
d'estimation usuelles sont employ'ees, ce qui donne les r'esultats suivants
pour l'estimation des paramètres inconnus (voir Annexe A) :
mb =
1'IY1J/IJ
bg2 =
(X'X)-1X'Y1J/J
bE' 2 =
1'IYZ(Z'Z)-1/I
I/I - X(X'X)-1X')Y1J/J
bg1 = (II - 1I1'
bE' 1 =
1'IY(IJ -
1J1' J/J -
Z(Z'Z)-1Z')I
b =
(X'X)-1X'YZ(Z'Z)-1
I/I - X(X'X)-1X')YZ(Z'Z)-1
bg3 = (II - 1I1'
bE' 3=
(X'X)-1X'Y(IJ
-
Z(Z'Z)-1Z')
Cependant, cette facon d'ecrire le mod`ele et les
calculs qui en decoulent sont lies a` la structure des donnees. En effet, dans
ce que nous avons presente, les observations sont supposees àetre dans
une matrice a` I lignes et J colonnes, ce qui
prevoit cette matrice de donnees compl`ete. Cela s'interpr`ete dans le cas d'un
essai multienvironnement par exemple, par le fait que chaque genotype doit
àetre present dans chaque environnement. Or la realiteest souvent
autre.
Pour les essais o`u toutes les varietes ne sont pas dans tous
les environnements, cette methode, qui est ici generalisee avec
p covariables associees au facteur genotype et q
covariables liees au facteur environnement, peut se presenter sous forme
indicielle.
Yij = m (E
p
|
xiMp + gi) + (E
q
|
zj áq 3
E ) + E
p,q
|
xp. zqjã+
e
·
· (2.3)
z pq z3
|
|
o`u gi represente la part des effets moyens
non expliquee par les covariables X et Ej
celle non expliquee par les covariables Z ;
xpi etant la valeur de la
pe covariable associee au genotype i
et zqj la valeur de la
qe covariable associee a` l'environnement
j.
Les vecteurs ã =
|
r11
lfp 713.
|
, á =
|
(
|
á1
...
áq
|
?
? ? ?
|
et â =
|
(
â1
)
.
âp
|
|
sont estimes par regression du vecteur Y des
observations, de longueur IJ,
respectivement sur
le produit semi-tensoriel ligne (Dieng, 2003) des deux
matrices des covariables, sur la matrice de covariable
attach'ee au facteur environnement et sur la matrice de covariable attach'ee au
facteur g'enotype. Nous reprenons ci-après la d'efinition du produit
semi-tensoriel ligne.
D'efinition 1 Produit semi-tensoriel ligne Si A est une
matrice rectangulaire (m,r),
Si B est une matrice rectangulaire (m,s),
A ? B est dit produit semi-tensoriel ligne de A par B.
C'est une matrice rectangulaire (m,rs) qui se présente ainsi
a11 · B[1,]
?
a21 · B[2,]
? ? ? ? ? ...
am1 · B[m,]
|
a12 · B[1,]
a22
· B[2,]
...
am2 · B[m,]
|
· ·
· ·
· ·
|
·
·
·
|
a1r · B[1,]
?
a2r · B[2,]
... ?????
amr · B[m,]
|
o`u B[m,] est la me ligne
de B. Autrement dit, A ? B est la juxtaposition de tous les produits termes a`
termes possibles entre une colonne de A et une colonne de B.
Nous nous servons du produit semi-tensoriel ligne pour obtenir
la matrice d'incidence pour l'interaction a` partir des matrices d'incidence
des effets simples.
Avec cette pr'esentation indicielle, la r'egression
factorielle est effectu'ee en deux 'etapes : d'abord la r'egression des
observations sur le produit semitensoriel ligne des deux matrices de
covariables et celle sur les deux matrices des covariables auxquelles est
ajout'ee par la suite l'estimation des r'esidus de ces r'egressions.
Nous pr'esentons ci-dessous un modèle, plus g'en'eral, o`u
tous les paramètres sont estimables en une seule 'etape.
Y = m1IJ + X ·a1
+X·n2 + Z ·b2
+L·Ib2 + X ? Z ·
(ab)11
- Xn1 est la régression sur les
covariables génotypes
+x? Z ·
(ab)21 + X ?7L·
(ab)12
+X?7L·
(ab)22 + e
- Y est le vecteur des observations, de longueur
IJ
- m1IJ est la moyenne générale des
observations
->Xn2 est l'écart des effets génotype a` la
régression sur les covariables génotypes,
-7Lb2 est l'écart des effets environnements a` la
régression sur les covariables - Zb2 est la
régression sur les covariables environnements
Xétant la matrice d'incidence des génotypes
-x?Z(ab)21 est l'effet des
covariables environnements modulépar les génotypes - X ?
Z(ab)11 est l'effet des covariables environnements
modulépar les cova-
environnements,7Létant la matrice d'incidence des
environnements
riables génotypes
non expliquépar les covariables génotypes
- X ?L(nb)12
est l'effet des covariables génotypes modulépar les
environnements non expliquépar les covariables environnements
-x?7L(ab)22
est l'interaction G×E expliquée ni par les
covariables génotypes, ni par les covariables environnements.
Ainsi, les paramètresa1,a2,b2,b2,
(ab)11,
(ab)21,
(ab)12,
(rb)22, sont estimés par simple
régression linéaire sur les matrices et vecteurs
adéquats.
Exemple d'écriture pour un essai multilocal :
Pour un essai multilocal de 3 génotypes effectuéen 2
lieux différents o`u nous avons 2 covariables associées au
facteur lieu et 1 covariable associée au facteur variété,
les vecteurs et matrices se présentent comme suit :

39
?
? ? ? ? ? ? ? ? ? ?
?
? ? ? ? ? ? ? ? ? ?
Y11
Y21
Y31
Y12
Y22
Y32
Y=
?
? ? ? ? ? ? ? ? ? ?
X=
?
? ? ? ? ? ? ? ? ? ?
?
? ? ? ? ? ? ? ? ? ?
?
? ? ? ? ? ? ? ? ? ?
Z=
X=
X111 X211
X311 X121 X221
X321
?
? ? ? ? ? ? ? ? ? ?
?
? ? ? ? ? ? ? ? ? ?
XeZ=
X111Z111
X111Z112
X211Z121
X211Z122
X311Z211
X311Z212
X121Z221
X121Z222
X221Z311
X221Z312
X321Z321
X321Z322
?
? ? ? ? ? ? ? ? ? ?
I
?
? ? ? ? ? ? ? ? ? ?
Z=
1 0 1 0 1 0
0 1 0 1 0 1
Z111 Z112
Z121 Z122 Z211
Z212 Z221 Z222
Z311 Z312 Z321
Z322
1 0 0 0 1 0 0 0
1 1 0 0 0 1 0 0 0
1
?
? ? ? ? ? ? ? ? ? ?
|
?
XeZ=
? ? ? ? ? ? ? ? ? ?
|
Z111
0
0
Z221
0
0
|
Z112
0
0
Z222
0
0
|
0
Z121
0
0
Z311
0
|
|
0
0
Z212
X e
0
0
Z322 1
|
?
? ? ? ? ? ? ? ? ? ?
|
X111
X211
X311
0
0
0
|
0
?
0
0
X121
X221 ? ? ? ? ? ? ? ? ? ?
X321
|
0 0
Z122 0
0 Z211
0 0
Z312 0
0 Z321
|
0
|
0
|
0
|
0
|
0
|
|
0
|
1
|
0
|
0
|
0
|
|
0
|
0
|
0
|
1
|
0
I
|
|
0
|
0
|
0
|
0
|
0
|
|
0
|
1
|
0
|
0
|
0
|
|
0
|
0
|
0
|
1
|
0
|
1 0 0
1 0 0
x? =
?
? ? ? ? ? ? ? ? ? ?
Jusqu'àmaintenant les covariables sont supposées
continues. Toutefois, cette méthode est encore valable dans le cas o`u
elles sont de type qualitatif. Il suffit de la même manière que
l'on passe de la régression a` l'analyse de variance, de remplacer
chaque colonne de la matrice de covariables par des colonnes indicatrices des
niveaux de la colonne de covariable qualitative considérée
(Denis, 1980).
Dans le cas o`u les covariables sont trop nombreuses,
différentes méthodes de sélection sont
présentées dans (Denis, 1980).
2.4.2 Illustration avec les données de l'essai
multilocal
Les données sont celles du tableau 1.2 de la page 16.
Des variables climatiques ont étémesurées
quotidiennement sur les sites des essais. Ces variables (pluie, rayonnement
solaire, vitesse du vent, etc.) se trouvent en fait plus nombreuses que les
essais, ce qui rend impossible leur complète utilisation dans un
modèle de régression factorielle. Pour décrire les
différents lieux, nous avons uniquement retenu la pluviométrie
totale sur le cycle de culture qui, a` notre sens, permet de bien les
caractériser.
Pour la covariable génotype, nous pouvons utiliser des
indices de tolérance ou de sensibilitéqui font intervenir le
rendement en conditions de sécheresse et le rendement en conditions
optimales. Mais le mode de calcul de ces indices pénalise très
fortement les génotypes a` rendement potentiel élevé; des
valeurs
très élevées caractérisent
plutôt des génotypes rustiques a` faible productivitéque
des génotypes a` rendement stable et élevé(Belhassen, This
et Monne-
veux, 1995). Nous privilégions le taux de croissance de
culture (C) décrit par Turner, Wright et Siddique
(2002) comme étant :
C = (PF + (1,65 PG))/T
o`u PF est le poids des fanes, PG
le poids des gousses et T la durée
floraisonrécolte. Ce taux permet effectivement de caractériser
les génotypes selon leur sensibilitéa` la sécheresse car
plus une variétéproduit de biomasse totale et de gousses en un
temps relativement court, mieux elle est adaptée a` la
sécheresse. Le calcul de C s'est fait sur des
données qui n'ont pas servi a` la modélisation de l'interaction
G×E.
Les résultats de la régression factorielle
tenant compte de la covariable C attachée aux
génotypes et de la covariable Pluie liée aux
environnements, sont présentés dans le tableau 2.5. La somme des
carrés résiduels (751 771,62) est ce qui reste des interactions
G×E une fois que nous avons utiliséles covariables
pour les interpréter. Sans ces covariables, cette somme des
carrés était égale a` 962 311,6 (modèle additif,
tableau 2.1 de la page 24).
|
Effet
|
d.l
|
|
Somme de carrés
|
|
Carrémoyen
|
Statistique F
|
Niveau de signification
|
|
Génotype
|
5
|
|
232
|
764,98
|
46
|
552,99
|
2,23
|
0,0725
|
|
Environnement
|
10
|
8
|
100
|
921,68
|
810
|
092,17
|
38,79
|
0,0000
|
|
C×Pluie
|
1
|
|
8
|
896,50
|
8
|
896,50
|
0,43
|
0,5181
|
|
C×Env.
|
9
|
|
162
|
944,57
|
18
|
104,95
|
0,87
|
0,5624
|
|
Génotype×Pluie
|
4
|
|
38
|
698,94
|
9
|
674,74
|
0,46
|
0,7622
|
|
Résidus
|
36
|
|
751
|
771,62
|
|
|
|
|
TAB. 2.5 - Régression factorielle des
données de l'essai multilocal.
Nous notons tout de même que les covariables
associées aux effets principaux ne sont pas significatives (0,5181 pour
le produit des deux covariables, 0,5624 pour la covariable liée a`
l'environnement et 0,7622 pour celle liée au facteur génotype).
Cela indique que la décomposition de l'interaction n'est pas pertinente
avec de telles covariables et qu'il faudrait en sélectionner d'autres.
Ce qui pose le problème du choix de covariables caractérisant le
mieux le climat des environnements. En effet, face au nombre important de
variables climatiques quotidiennement mesurées sur les lieux, se pose la
difficultéde
les r'esumer efficacement. Dans ce cas, l'utilisation de
modèles de simulation de culture pour les synth'etiser peut être
une alternative.
2.5 Un modèle de simulation de cultures : SarraH
Les modèles de simulation de cultures permettent de
pr'edire la production de l'ensemble du peuplement v'eg'etal cultiv'e d'une
parcelle. Pour cela, ils reproduisent le fonctionnement du système
sol-plante-atmosphère et permettent la comparaison de divers
itin'eraires techniques en fonction de l'espèce utilis'ee, du type de
sol, des donn'ees climatiques, etc. Moyennant un changement d'echelle
convenable, ces modèles peuvent alors être utilis'es a` diverses
fins : pr'evisions des r'ecoltes et planification de la politique agricole et
alimentaire d'un pays, analyse prospective des changements climatiques,
protection de l'environnement, etc. (Affholder, 1997).
Au Sahel, les modèles pr'ecurseurs fond'es sur une
estimation du bilan hydrique des cultures en cours de cycle, ont 'et'e 'ecrits
avant le d'ebut des ann'ees 1980 (Franquin et Forest, 1977). Le bilan hydrique
se situe a` l''echelle de la plante ou du champ et permet de comparer les
quantit'es d'eau fournies et les quantit'es d'eau utilis'ees par la plante. Il
tient aussi compte de la constitution de r'eserves d'eau et des
pr'elèvements ult'erieurs sur ces r'eserves. Les apports d'eau fournis
par les pr'ecipitations sont mesur'es. Les pertes se composent de la
combinaison de l''evaporation et de la transpiration des plantes, plus connue
sous le nom d''evapotranspiration.
L''equation du bilan hydrique, sous sa forme la plus g'en'erale,
se fonde sur l''equation de conservation de la masse
P = ÄS + D + ETR + R
o`u P d'esigne la pluviom'etrie, ÄS
la variation de stock d'eau dans le sol, D le
drainage, ETR l''evapotranspiration et R les
ruissellements.
L'estimation de l''evapotranspiration peut se faire en deux
'etapes. Pour une culture, l''evapotranspiration en conditions non limitantes
d'alimentation hydrique ou 'evapotranspiration maximale (ETM)
est d'abord calcul'ee a` l'aide de l''evaporation Bac Classe A (EV
A) et du coefficient cultural K'c
caract'eristique de la culture et de son stade de d'eveloppement.
ETM = K'c × EV
A
Ensuite, Eagleman (1971) a propos'e une m'ethode d'estimation
de l''evapotranspiration r'eelle qui est fonction de l'evapotranspiration
maximale et de l'humidit'e relative du sol.
Doorenbos et Jassam (1979) ont d'evelopp'e une approche de
mod'elisation des rendements r'eels fond'ee sur les consommations r'eelles et
maximales d'eau et la productivit'e potentielle des cultures. Dans les zones
o`u les flux hydriques sont les plus d'eterminants, la mod'elisation du bilan
hydrique et la mise en relation de ses paramètres avec la production
aboutit a` une estimation bien adapt'ee des rendements des cultures.
Le modèle Arachide bilan hydrique (Arabhy) constitue la
première g'en'eration de modèle semi d'eterministe d'evelopp'e
par le CERAAS en collaboration avec le CIRAD, pour l'estimation de la
production de l'arachide au S'en'egal tenant compte du bilan hydrique (Annerose
et Diagne, 1990, 1994). Il ajoute a` la simulation du bilan hydrique des
cultures, celle des r'eactions de la plante aux stress hydriques. Ce
modèle estime en fin de cycle la production potentielle.
Le modèle de Système d'analyse r'egional des
risques agroclimatiques (Sarra) simule des indicateurs hydriques de production
(Forest et Clopes, 1991). Ces indicateurs sont fond'es sur la consommation en
eau r'eelle et maximale de la plante en relation avec les techniques
culturales. Pour ce modèle qui est
utilis'e pour mesurer l'impact du climat sur une culture
annuelle, il est suppos'e que la performance d'une culture est une fonction
simple d'une combinaison d'indicateurs hydriques cumul'es au cours d'un cycle
v'eg'etatif.
Cette première g'en'eration de modèles
param'etr'es pour les zones sah'eliennes, utilise en entr'ee une base de
donn'ees a` l'echelle locale de la parcelle sur le climat, le sol et les
cultures. Ces modèles sont assez simples et sont 'ecrits sans module de
bilan de carbone ni prise en compte de la notion de densit'e du peuplement.
Le bilan de carbone est fond'e sur l'assimilation du carbone
selon l'interception de l''energie lumineuse en fonction du taux de couverture
foliaire et la conversion de la fraction de rayonnement intercept'ee en
matière sèche. Associ'e au bilan hydrique, le bilan de carbone
constitue un 'el'ement essentiel dans le processus de croissance et de
productivit'e des cultures.
Le modèle de simulation de cultures SarraH (Baron,
2002) est quant a` lui, une version plus d'etaill'ee de Sarra et fait
intervenir, en plus de la simulation du bilan hydrique, plusieurs autres effets
tels que celui de la temp'erature et celui de la radiation solaire sur la
production. Il est fond'e sur la notion de l'effets multiplicatif de
l'efficience de l'eau (WUE, Water use efficiency) et de
l'efficience de l'energie radiative (RUE,Radiation use
efficiency) pour simuler l''elaboration des biomasses.
Le modèle SarraH fonctionne au pas de temps journalier
et la production de trois types de plantes peuvent y être simul'ee : mil,
arachide et palmier. Nous avons pris l'arachide comme plante test pour cette
'etude.
De par la complexit'e inh'erente au système
sol-plante-atmosphère, les processus simul'es sont formul'es par des
ensembles d''equations que l'on peut regrouper par grand ensembles de (bilan
hydrique, ph'enologie, etc.) et optimis'e par sous ensembles de modules
('equations d'ecrivant une 'etape d'un
processus). Le lien entre les modules et les processus
s''etablissant au travers
de variables d''etats d'ecrivant la plante, le sol
et l'environnement climatique.
D'evelopp'e sous cette approche modulaire, SarraH permet de
simuler
- la biomasse initiale qui est fonction de la densit'e de
semis et du poids sec moyen d'un grain; ce qui permet de d'eduire la densit'e
de peuplement disposant du taux de lev'ee;
- la ph'enologie de la plante qui se d'efinit en plusieurs
phases en relation avec la somme de temp'erature : phase v'eg'etative, phase
reproductive, phase de maturation des graines. Chaque phase, d'une dur'ee
constante, est exprim'ee en temps thermique qui est fonction des temp'eratures
maximale, minimale et de base. La temp'erature de base est celle au-dessous de
laquelle la plante ne se d'eveloppe pas. Les dur'ees des phases ainsi que la
temp'erature de base sont des caract'eristiques vari'etale des plantes
simul'ees (pour la vari'et'e d'arachide qui est simul'ee dans SarraH, elle est
'egale a` 13C;
- le bilan hydrique qui d'etermine la dynamique de l'eau dans
le sol en fonction des contraintes climatiques ('evaporation du sol) et de la
consommation en eau de la plante (transpiration). Cette consommation en eau de
la plante permet de d'efinir un frein hydrique qui est le rapport entre la
consommation r'eelle et la demande en eau de la plante;
- le bilan de carbone qui d'etermine la fabrication
journalière d'assimilats en fonction du coefficient de conversion de
l'espèce, du rayonnement intercept'ee et donc de taux de couverture
foliaire. A cette production potentielle d'assimilat est appliqu'e le frein
hydrique;
- la r'epartition des assimilats, racine, feuille, tige et
organes reproducteurs qui 'evolue en fonction des phases ph'enologiques de la
plante, ces lois de r'epartition sont fond'ees sur des relations
allom'etriques.
2.6 Limites des méthodes classiques d'étude
des interactions G×E
Le modèle 2.1, qui correspond a` un modèle
d'analyse de variance a` deux facteurs, permet uniquement de tester la
significativitédes interactions, mais il ne permet aucunement de les
explorer. Dans ce cas, l'information contenue dans ces interactions serait
inexploitée si aucune analyse supplémentaire n'était faite
(Crossa, 1990). Cette logique sous-tend la modélisation des interactions
G×E qui permet alors d'améliorer la
prédiction des performances des génotypes dans de nouveaux
environnements en contrôlant la variation importante de ces interactions
et en l'enlevant de la partie significative du modèle (Gauch, 1990,
1992).
La régression conjointe parait peu adaptée a`
cette forte variation d'une année sur l'autre ou d'un site a` l'autre.
Cette méthode permet d'interpréter l'interaction par le potentiel
du milieu, estimépar l'effet moyen du milieu. Pour un nouveau milieu non
encore couvert par une expérimentation, nous n'avons pas d'estimation du
potentiel, donc pas de prédiction de l'interaction. De ce fait, elle
permet de décrire uniquement les résidus du modèle additif
et n'utilise aucune information supplémentaire du milieu pour
modéliser l'interaction.
La méthode AMMI est un outil permettant de comprendre
des données complexes, notamment celles obtenues dans le cadre des
interactions G×E. Cependant, elle n'a qu'un grand
intérêt descriptif et constitue une technique appropriée
uniquement dans une perspective d'analyse préliminaire. 'Etant
donnéqu'une ACP est effectuée sur les résidus du
modèle additif, la méthode AMMI permet tout juste
d'étudier les corrélations entre composantes principales et
covariables de l'environnement et du génotype, alors que le but est de
prédire les unes a` partir des autres (Yan et al., 2001). Elle
péche ainsi par le fait que cette modélisation de l'interaction
se fait sans l'utilisation
des données climatiques des environnements, car, de
même que l'analyse de variance, les effets estimés pour les
environnements sont imprévisibles.
La régression factorielle, elle, tient compte des
conditions climatiques des environnements pour prédire la réponse
des génotypes. Mais elle suppose que l'action de l'environnement sur la
production est linéaire, ce qui n'est pas certain. De plus, le nombre
important de variables climatiques généralement mesurées
sur les lieux d'essais fait qu'elles ne peuvent pas être totalement
prises en compte par cette méthode.
Il s'agira alors, d'améliorer la prédiction de
la performance des génotypes en réduisant l'impact de la
variabilitéclimatique sur la précision de cette estimation. Une
solution serait de modéliser les variations de la réponse des
génotypes en fonction de l'environnement par l'utilisation de
modèles de simulation de cultures tels que Diagnostic hydrique des
cultures (DHC, Forest et Cortier, 1990), Irrigation scheduling information
system (IRSIS, Fao, 1987), SarraH. Seulement, les paramètres de tels
modèles ne sont connus que pour un petit nombre de génotypes.
Chapitre 3
La methode APLAT
Ce chapitre traite de notre première m'ethode propos'ee
qui consiste a` lin'eariser la performance des g'enotypes pr'edite par le
modèle SarraH au voisinage d'un g'enotype de r'ef'erence. Une fois la
lin'earisation effectu'ee, l'estimation des paramètres, du fait du
nombre important de r'egresseurs dont nous disposions, s'est faite par
r'egression Partial least squares. Nous commencerons alors par
pr'esenter a` la section 1.1 cette technique de r'egression et terminerons ce
chapitre en pr'esentant a` la section 1.2, la m'ethode d'estimation APLAT.
3.1 La regression Partial least squares
La r'egression PLS, Partial least squares est
devenue aujourd'hui, une m'ethode très utilis'ee dans le cas des
r'egressions sur donn'ees corr'el'ees. Aussi, est-elle une bonne alternative
s'il y a plus de r'egresseurs que d'observations (Wold, Albano, Dunn, Esbensen,
Hellberg, Johansson, Sjöström 1983; Tenenhaus, 2001).
Un petit nombre de variables appel'ees »facteurs» ou
»variables latentes» sont construites l'une après l'autre de
façon it'erative et permettent de remplacer l'espace initial des
nombreux r'egresseurs par un espace de plus faible dimension. Ces facteurs
deviennent les nouvelles variables explicatives dans un modèle de
r'egression lin'eaire classique.
Les facteurs sont orthogonaux, et sont des combinaisons
lin'eaires des variables explicatives initiales. A ce titre, ils renvoient aux
composantes principales de la RCP, R'egression sur composantes principales.
Mais alors que ces dernières ne sont calcul'ees qu'àpartir des
variables explicatives (et donc sans r'ef'erence a` la variable a` expliquer),
les facteurs de la r'egression PLS maximisent les corr'elations successives
entre les r'egresseurs et la variable a`
expliquer, tout en maintenant la contrainte d'orthogonalit'e
avec ceux d'ejàconstruits.
La r'egression PLS s'effectue selon le principe de
l'algorithme NIPALS, Nonlinear estimation by iterative partial least
squares d'evelopp'e par Herman Swold (1966) pour l'analyse en
composantes principales. Cette r'egression s'inspire de l'approche PLS (Wold,
1975) pour l'estimation des modèles d''equation structurelles reliant
plusieurs blocs de variables entre eux.
A pr'esent, pour d'ecrire cette m'ethode, nous nous
plaçons dans le cadre du modèle lin'eaire classique. Le vecteur
des observations Y de dimension n × 1
est suppos'e suivre le modèle suivant
Y = Xâ + e (3.1)
o`u le vecteur â d'ordre p est le
paramètre inconnu a` estimer, X la matrice de dimension
n × p des variables explicatives, et le vecteur e
un terme d'erreur al'eatoire.
Nous supposerons qu'il n'y a pas de données manquantes
et qu'il n'y a qu'une seule variable a` expliquer pour une explication plus
claire de la méthode. L'algorithme PLS calcule les variables latentes
t1,··· ,th étape par
étape. Ces variables latentes th = Xwh sont des
combinaisons linéaires des X qui sont orthogonales
entre elles et qui maximisent Cov(th, Y) sous la contrainte
II wh 11= 1.
A l'étape 1, w1 = (w1 1
· · ·
wp1)' est solution
du problème d'optimisation
? ?
?
max Cov(Xw1, Y) k w1
11= 1
Pour déterminer w1, il suffit
d'écrire l'expression du Lagrangien.
L(w1,ë) =
Cov(Xw1,Y) - ë(w'
1w1 - 1)
= w1
1Cov(X1,Y) + ··· +
wp 1Cov(Xp,Y) -
ë[(w1 1)2 + ·
· · + (wp
1)2 - 1]
o`u ë est le multiplicateur de Lagrange
associéa` la contrainte.
Les solutions a` ce problème d'optimisation sont
obtenues en dérivant L(w1, ë) par
rapport a` w11,··· ,
wp 1, ë. Les p + 1
équations aux dérivées partielles ou
équations normales s'écrivent
|
? ???????
???????
|
Cov(X1,Y) -
2ëw1 1 = 0
...
Cov(Xp,Y) - 2ëw1 p =
0
(w11)2
+ ···(wp
1)2 = 1
|
o`u Xp est la
pecolonne de X
En remplacant dans la dernière équation
de ce système les composantes de w1 tirées dans
les p premières équations, nous obtenons
[Cov(X1,Y)/2ë]2
+ · · · + [Cov(Xp,Y)/2ë]2=
1
D'o`u
X p
j=1
[Cov(Xj,Y)]2 =
4ë2
Et
ë =
/P[Cov(Xj,Y)]2/2
En reportant cette valeur de ë dans chacune
des p premières équations nor-males, nous
avons
wj 1 =
Cov(Xj,Y)/V/P[Cov(Xj,Y)]2
Ainsi, la première composante t1
= w1 1X1 +
· · · + wp 1Xp
est construite. Puis, il est effectuéune régression
simple de Y sur t1
Y = c1t1 +
Y1
o`u c1 est le coefficient de régression
et Y1 le vecteur des résidus.
S'il reste encore de l'information, il est construit une
deuxième variable latente t2?t1. Cette
deuxième variable latente est combinaison linéaire des colonnes
de X1, résidu de la régression linéaire
de X sur t1.
A l'étape 2, w2 = (w1 2
· · · wp
2)' est solution du problème
d'optimisation
|
? ?
?
|
max Cov(X1w2,
Y1) II w2 11= 1
|
La deuxième variable latente t2
construite, il est effectuéune régression linéaire
multiple de Y sur t1 et
t2
Y = c1t1 +
c2t2 + Y2
Cette proc'edure it'erative peut ainsi continuer en utilisant les
r'esidus Y2, X2 des r'egressions de
Y, X sur t1 et
t2.
Le nombre de composantes
t1,
·
·
· ,tH a`
retenir avec H rang(X), peut être d'etermin'e a` l'aide
de trois critères : l'ajustement de l''echantillon d'apprentissage
(X, Y) par
(bX,
bY), la pr'ediction sur un
'echantillon externe et la pr'ediction interne aux donn'ees d'apprentissage
appel'ee validation crois'ee.
3.2 La méthode APLAT : linéarisation
au-tour d'un témoin
Cette m'ethode a fait l'objet d'un article publi'e aux Comptes
rendus de l'acad'emie des sciences dont l'original se trouve en Annexe B.
I. Dieng, E. Goz'e, R. Sabatier
Lin'earisation autour d'un t'emoin pour pr'edire la r'eponse de
cultures C. R. Biologies 329 (2006) 148-155
3.2.1 Le modèle proposé
Si nous partons du modèle de simulation de cultures,
chacune des sorties de ce modèle, le rendement d'un g'enotype i
dans un environnement j est la somme :
1. d'un rendement potentiel pr'edit avec le modèle de
simulation;
2. d'un biais, esp'erance de l''ecart du potentiel au
r'ealis'e;
3. d'une erreur al'eatoire.
Yij = f(Zj, èi) + îj + uij
(3.2)
o`u Zj est le vecteur des variables telles
que la pluie, la temperature, etc. mesurees sur l'environnement
j et èi le vecteur de longueur
P des paramètres du genotype i. Nous supposons
que le biais îj ne depend que de l'environnement
j : il est donc le màeme pour tous les genotypes d'un
màeme environnement. L'erreur uij est supposee
aleatoire avecE(uij) = 0 et Var(uij) =
ó2u.
Comme dit precedemment, les paramètres des
modèles de simulation de cultures ne sont generalement connus que pour
un petit nombre de genotypes. Considerons un modèle de simulation de
cultures et un genotype de reference dont les paramètres sont connus et
appelons è0 le vecteur de ses paramètres. Alors,
supposons f de classe C1 dans un
voisinage de è0 et f' derivable sur ce
voisinage. De plus supposons èi au voisinage de
è0. En pratique, les genotypes dont nous chercherons a` estimer les
paramètres seront choisis de telle sorte qu'ils ne soient pas trop
eloignes du genotype de reference. Alors, un developpement en serie de Taylor
a` l'ordre 1 nous donne :
|
f(Zj, èi) = f(Zj,
è0) +
|
XP ?f
[?è(p)
(e) - è()) + ?
[(èi -
è0)'
(èi - è0)] (3.3)
p=1 0=00
,Z=Zj
|
avec
è(p)
iet
è0p) la
pe composante du vecteur de paramètres
respectivement du genotype i et du genotype de reference.
Posons
X4p)
= [?Zd0=00,Z=Zj
c'est une fonction de l'environnement j, et
â(p)
i=
è(p) i--
è(p)
0
une fonction du g'enotype i.
La fonction X(p)
j est la d'eriv'ee partielle de la sortie du
modele de simulation de cultures pour l'environnement j par
rapport a` la pe composante du vecteur de
parametres de la vari'et'e de r'ef'erence. Comme la fonction f
n'est pas g'en'eralement connue analytiquement, ces sensibilit'es peuvent
àetre obtenues par une m'ethode de d'erivation num'erique. Nous avons
retenu tout simplement
X(p) = ? f
j [f + hè,c)p) ) -- f
(èOp) --
hè,c,p))
[BB(P)
0=00,Z=Zj 1
2h (
è,?
Z=Zj
avec hè(p) 0tres
petit, de l'ordre de
è0p).10-4
en pratique. D'autres m'ethodes existent, celle-ci 'etant la plus simple et la
plus 'econome en calculs.
Avec ces notations et d'apres les 'equations
(3.2) et (3.3) qui permettent d''ecrire
f(Zj, è0) = Y0j
-- îj -- u0j
nous pouvons poser, en n'egligeant o
[(èi --
è0)'(èi
-- è0)] :
|
Yij -- Y0j =
|
XP
p=1
|
X(p)
j
· â(p)
i + oij (3.4)
|
o`u oij = uij -- u0j.
Ainsi,E(oij) = 0, Var(oij) =
2ó2 u,Cov(oij,
oi'j') = 0, mais Cov(oij, oi'j) =
ó2 u.
Si nous disposons de I g'enotypes et de
J environnements, nous pouvons poser le modele suivant :
Y -- (Y0 1I) = X
·
â + o (3.5)
Le vecteur Y repr'esente le rendement de tous
les g'enotypes dans tous les envi-
ronnements, rang'e par environnement et
par g'enotype. Si tous les g'enotypes
ont eteobserves une fois dans chaque environnement, ce vecteur
est de lon-
gueur IJ. Puis
Y'0 = (Y01 · · ·
Y0J) et 1I est un vecteur formede 1,
de longueur
I.
Le symbole ? designe le produit de Kronecker.
Le vecteur o est un vecteur d'erreur aleatoire. Sa matrice de
covariance est de la forme
ó2uÙ
avec
|
|
?
? ? ? ? ? ? ? ? ?
|
ù1
|
..
|
.
|
|
|
0
|
1
|
|
Ù=
|
|
|
|
ùj
|
|
|
|
o`u
|
|
0
|
|
|
|
...
|
ùJ
|
(
ùj =
Les matrices Ù et ùj sont
carrees de nombre de lignes, respectivement le nombre d'observations de tous
les environnements et le nombre d'observations de l'environnement
j. La matrice Ù est bloc diagonale tandis que
ùj est une matrice form'ee de 1 partout sauf des 2 sur
la diagonale. Dans le cas o`u tous les genotypes ont etevus une seule fois dans
chaque environnement, Ù est de dimension IJ
× IJ et ùj de dimension I ×
I.
Ensuite,
X = [ X(1) ? II · ·
· X(p) ? II ·
· · X(P) ?
II
o`u
X(p)' = [
X1p) ...
XSp) ]
de longueur J, est le vecteur des derivees
des sorties de notre modele de simulation de cultures, SarraH, dans chacun des
environnements par rapport au pe parametre du
vecteur de parametres du temoin.
La matrice II est la matrice
identited'ordre I, la matrice
X(p) 0II
s'ecrit alors de cette facon :
|
X(p)
1
...
X(p)
(
J )
|
0
|
(
|
1
.
0
|
0
..
1
|
)
|
=
|
X(p)
1
?
0
? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?
X(p)
J
0
|
...
...
0
?
X(p)
1
...
0
? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?
X(p)
J
|
C'est une matrice a` IJ lignes et
I colonnes. Par consequent, la matrice X est de
dimension IJ x PI.
Enfin
â = [ â(1)'
...â(P)' 1'
avec
â(p)' = [
â1.p) ...
â(p) ]
Nous avons proposed'appeler cette methode par l'acronyme APLAT
: Ap-
proximation Par Linearisation Autour d'un Temoin. Elle consiste a`
appro-
cher, localement, le rendement predit par un modele de simulation de
cultures,
par s'erie de Taylor a` l'ordre 1 au voisinage du vecteur de
paramètres d'un g'enotype de r'ef'erence. Cette lin'earisation permet,
par r'egression lin'eaire, l'estimation des paramètres de ces
g'enotypes. Par la suite, la pr'ediction de l''ecart entre le rendement de ces
g'enotypes et celui du g'enotype de r'ef'erence dans des environnements
nouveaux, c'est a` dire o`u ils ne sont pas encore test'es, pourra se faire si
le climat de ces derniers est connu.
Il y a en g'en'eral beaucoup de paramètres dans un
modèle de simulation de cultures et peu d'environnements dans un essai
multienvironnement, ce qui rend souvent PI grand par rapport
a` IJ.
Pour notre exemple, nous avons utilis'e SarraH comme
modèle de simulation de cultures. Ce modèle dispose de 61
paramètres fonction du g'enotype. Avec un tel nombre de pr'edicteurs,
l'estimation de â s'est faite par r'egression PLS. Ceci permet de
r'eduire l'espace des r'egresseurs de rang de X a` k
dimensions.
La r'egression PLS, voir section 3.1, s'effectue selon le
principe de l'algorithme NIPALS, Nonlinear estimation by Iterative
Partial Least Squares, (Tenenhaus, 2001) o`u un ensemble de
r'egressions successives par moindres carr'es ordinaires est effectu'e, en
même temps que le calcul des composantes.
Ici, la matrice de covariance de o est 'egale a`
ó2
uÙ et non a` ó2
uIIJ. La solution serait d'effectuer toutes les r'egressions
partielles par moindres carr'es g'en'eralis'es. Mais cette matrice de
covariance est inconnue. Elle s''ecrit tout de même a` une constante
multiplicative près en fonction de Ù qui elle
est connue. La matrice Ù 'etant sym'etrique et
semi-d'efinie positive, par d'ecomposition de Cholesky, il existe une matrice
ç tel que ç'ç =
Ù-1.
Si nous multiplions a` gauche tous les termes du modèle
3.5 par ç, nous obtenons le modèle 3.6 dont les erreurs sont
ind'ependantes.
çY - ç(Y0
? 1I) = çX · â +
ço (3.6)
En effet, la variance de l'erreur ço s''ecrit
uçç-1(ç')-1ç'
= ó2
E(çoo'ç')
=
çI(oo')ç'
= ó2
uçÙç' =
ó2
uç(ç'ç)-1ç'
= ó2 uIIJ
â peut alors être estim'e a` l'aide des moindres
carr'es ordinaires, appelons âPLS
son estimateur.
Le nombre de composantes a` retenir est d'etermin'e par le PRESS,
Prediction Error Sum of Squares (Tenenhaus, 2001).
3.2.2 Illustration avec les données de l'essai
pluriannuel
Les données
Les donn'ees sont les r'esultats de l'essai pluriannuel
(tableau 1.1, page 13). Rappelons que le g'enotype de r'ef'erence est le
55-437, un g'enotype de 90 jours. C'est une vari'et'e
largement cultiv'ee au S'en'egal, dans le bassin arachidier, a` ce titre elle a
'et'e pr'esente comme t'emoin dans tous les essais et c'est elle aussi qui a
servi pour le param'etrage du modèle SarraH.
Dans ce milieu a` forte variabilit'e des pluies dans l'espace
et même dans le temps pour un même lieu, nous avons consid'er'e
chacune des 5 ann'ees d'exp'erimentation comme un environnement (figure
3.1).
SarraH a 'et'e utilis'e pour calculer X.
Compte tenu du nombre de donn'ees disponibles, seuls deux paramètres
(P = 2) ont 'et'e consid'er'es parmi les 61 de SarraH. Le
premier paramètre est en fait un coefficient multiplicateur qui agit sur
5 paramètres de SarraH : Coefficient moyen d'angle des feuilles,
Coefficient de conversion en assimilat, Coefficient d'efficience d'assimilation
des feuilles a` la phase v'eg'etative juv'enile, Coefficient d'efficience
d'assimilation
FIG. 3.1 - R'epartition des pluies sur la station de
Bambey au S'en'egal de 1994 a` 1998.
MIT
0 50 100 150 200 250
1994 1995 1996 1997 1998
Juin Juillet Août Septembre Octobre
des feuilles a` la première phase de maturation - phase
sensible de remplissage des grains - et Coefficient d'efficience d'assimilation
des feuilles a` la deuxième phase de maturation - phase non sensible -.
Le deuxième paramètre est le poids moyen des gousses.
Validation croisée
Pour valider notre modèle, nous avons r'eserv'e
successivement chacune des ann'ees et estim'e les paramètres des
g'enotypes sur les ann'ees restantes. Pour chacune des cinq ann'ees, nous avons
identifi'e un modèle par la m'ethode APLAT et les rendements observ'es
ont 'et'e compar'es a` ceux ainsi pr'edits. Les rendements sont exprim'es en
kilogrammes de gousses par hectare.
L''evaluation de la qualit'e de chaque modèle propos'e
est faite avec l'erreur
quadratique moyenne de pr'ediction MSEP,
Mean Squared Error of Prediction
(Wallach et Goffinet, 1987). La MSEP est utilis'ee comme
critère pour comparer diff'erents modèles dont le mod`ele moyen
(Colson, Wallach, Bouniols, Denis et Jones, 1995) d'efini pour nos donn'ees par
:
Yij = m + gi + Ej + äij (3.7)
o`u m est la moyenne de la population
et gi l'effet g'enotype. L'effet Ej de
l'environnement j est suppos'e al'eatoire, d'esp'erance nulle
et de variance
ó2E. Les erreurs
äij sont ind'ependantes, d'esp'erance nulle et de
variance ó2ä. De
plus, Ej et äij sont suppos'es
ind'ependants.
Le modèle moyen n'est rien d'autre que le modèle
lin'eaire mixte (2.1) o`u l'interaction al'eatoire G×E,
impr'evissible, est fusionn'ee avec le terme d'erreur qui porte les
màemes indices i pour le g'enotype et
j pour l'environnement.
Nous avons calcul'e les intervalles de confiance des
coefficients estim'es par la m'ethode bootstrap (Efron, 1979).
Cette technique permet d'estimer la loi inconnue d'un estimateur par une loi
empirique obtenue a` partir d'une proc'edure de r'e'echantillonnage fond'ee sur
des tirages al'eatoires avec remise des donn'ees. Les intervalles de confiance
construits sont de type percentile-t (Aji, Tavolaro, Lantz, et
Faraj, 2003).
Soit z(p)?b
i,PLS la variable al'eatoire d'efinie par :
(p)?b
zi,PLS
=
(3.8)
ei?LbS)
â(p)?b
i,PLS- 7A,S
s?(
o`u
â(p)i,PLS est le
(p.i)e 'el'ement de
eâPLS, il s'agit du pe
paramètre de la ie vari'et'e
estim'e par
la m'ethode PLS. gL?bS est obtenu
au be tirage avec b =
1,
·
·
· , B.
|
de la matrice de variance de
|
â(p)?b
i,PLS.
|
Soit bFB la fonction
de répartition empirique des z(pP
)?b Le fractile d'ordre á,
i LS'
1
B
XB
b=1
FB (á) est
estimépar la valeur
bt(á) telle que
1n = á
z(p)?b
Alors un intervalle de confiance percentile-t
pour le (p.i)e élément de
13 peut s'écrire :
hs(â(p)i,PLS)
t(1 - á) , /R1,3P)LS -
s(â(p)i,PLS) ·
Rá) ] (3.9)
R'esultats
Pour les modeles sans les données respectivement de
1994, 1995 et 1997, le PRESS minimal est atteint avec 6 composantes. Pour les
deux autres modeles, le PRESS est minimal avec 9 composantes, mais nous avons
réduit leur espace a` 5 dimensions car le PRESS n'y est pas trop
différent de ses valeurs minimales (figure 3.2).
Les coefficients des régressions PLS et les intervalles de
confiance qui leur sont associés, sont représent'es a` la figure
3.3.
Les MSEP estimées pour les modeles APLAT, sauf celle
sans les données de l'année 1998, sont inférieures aux
MSEP des modeles moyens correspondants (tableau 4.2). Ce qui signifie que pour
ces modeles, prédire le rendement par la méthode APLAT est
meilleur que par la moyenne des rendements du passé. Ainsi, 4 fois sur
5, la méthode APLAT s'est révélée meilleure que le
modele moyen. Toutefois, cette étude souffre de la faible taille de
notre échantillon.
Evolution du PRESS en fonction du nombre de composantes.
Le FIG. 3.2 - modèle (-1994) utilise les données
sauf celles de l'année 1994 et ainsi de suite.
PRESS
0.0 0.5 1.0 1.5 2.0 2.5
PRESS
Modèle (-1994) : Dim. opt. 6,
PRESS=0.051
5 10 15 20
Dim. Mod.
Modèle (-1996) : Dim. opt. 9,
PRESS=0.037
5 10 15 20
Dim. Mod.
Modèle (-1995) : Dim. opt. 6,
PRESS=0.061
5 10 15 20
Dim. Mod.
Modèle (-1997) : Dim. opt. 6,
PRESS=0.034
5 10 15 20
Dim. Mod.
Modèle (-1998) : Dim. opt. 9,
PRESS=0.046
5 10 15 20
PRESS
0.0 0.5 1.0 1.5
PRESS
0.0 0.5 1.0 1.5
PRESS
Dim. Mod.
Intervalle de confiance percentile-t a` 95% des
coefficients es-
timés. Le modèle (-1994) est
sans les données de 1994 et ainsi
FIG. 3.3 - de suite. Sur l'axe des abscisses, les valeurs
de chaque paramètre sont rangées par ordre
alphabétique de génotype. Le symbole
représente l'estimation ponctuelle des coefficients.
0.5 1.0 1.5 2.0 2.5
3
Paramètre 1 Paramètre 2
Modèle (-1994)
Paramètre 1 Paramètre 2
010 20 30
Modèle (-1996)
Modèle (-1995)
0 10 20 30
Modèle (-1997)

0 10 20 30 50 0 10 20 30
-0.5 0.0 0.5 1.0 1.5 2.0
Modèle (-1998)
Paramètre 1 Paramètre 2
50
40
30
0 10 20
|
APLAT
|
Modèle moyen
|
|
Modèle (-1994)
|
24
|
687,3
|
64
|
651,6
|
|
Modèle (-1995)
|
5
|
915,0
|
7
|
160,6
|
|
Modèle (-1996)
|
35
|
446,1
|
37
|
814,8
|
|
Modèle (-1997)
|
10
|
038,3
|
18
|
201,1
|
|
Modèle (-1998)
|
118
|
304,9
|
84
|
963,6
|
MSEP des différents modèles APLAT et
modèles moyens corres-
TAB. 3.1 - pondants de l'essai pluriannuel. Le
modèle (-1994) est sans les données de 1994 et ainsi de
suite.
3.2.3 Illustration avec les données de l'essai
multilocal
Les donn'ees sont les r'esultats de l'essai multilocal
(tableau 1.2, page 15). Chaque lieu est r'eserv'e et l'estimation des
paramètres effectu'ee sur les lieux restants.
Le modèle SarraH a aussi 'et'e utilis'e pour simuler le
rendement de la vari'et'e de r'ef'erence dans les diff'erents lieux. Les deux
paramètres a` r'eestimer restent les mêmes qu'àla section
pr'ec'edente.
Les MSEP estim'es pour les modèles APLAT sont
inf'erieures aux MSEP des modèles moyens correspondants, sauf celle sans
les donn'ees de Wenthiwy (tableau 3.2). Ainsi, pour cet essai multilocal, la
m'ethode APLAT s'est montr'ee meilleure que le modèle moyen 10 fois sur
11 (c''etait 4 fois sur 5 avec l'essai pluriannuel).
3.2.4 Conclusion
Au Sahel, l'interaction G×E est
largement due aux al'eas climatiques, dont la probabilit'e peut être
estim'ee a` l'aide de longues chroniques de relev'es m'et'eorologiques au sol.
Cependant, relier l'interaction G×E et la pluviom'etrie
a` l'aide d'un modèle de simulation de cultures n'est habituellement
possible que pour des vari'et'es dont on a estim'e les paramètres, au
prix d'une
|
APLAT
|
Modèle moyen
|
|
Modèle (sans Bambey)
|
10
|
289,4
|
45
|
374,1
|
|
Modèle (sans Gatte)
|
6
|
457,1
|
102
|
784,7
|
|
Modèle (sans Keur Fary)
|
3
|
474,2
|
44
|
457,9
|
|
Modèle (sans Ndangalma)
|
7
|
445,7
|
74
|
477,6
|
|
Modèle (sans Ndiadiane)
|
12
|
824,9
|
99
|
194,2
|
|
Modèle (sans Nioro)
|
47
|
015,4
|
157
|
989,6
|
|
Modèle (sans Nioro Sud)
|
234
|
506,4
|
738
|
389,6
|
|
Modèle (sans Pasokoto)
|
76
|
987,8
|
559
|
545,3
|
|
Modèle (sans Keur Samseun)
|
1
|
677,8
|
120
|
068,3
|
|
Modèle (sans Sinthiou Thiabala)
|
11
|
041,1
|
20
|
998,8
|
|
Modèle (sans Wenthiwy)
|
80
|
887,3
|
48
|
687,1
|
TAB. 3.2 - MSEP des différents modèles
APLAT et modèles moyens correspondants de l'essai
multilocal.
expérimentation spécifique. Le modèle
APLAT permet de prédire cette interaction avec les seules données
d'une expérimentation multilocale classique, sans autre instrumentation
que des stations météorologiques simples.
La méthode APLAT peut être vue ainsi comme un
outil d'aide a` la décision pour la sélection au Sahel. Dans
l'exemple o`u un sélectionneur doit tester plusieurs génotypes
dans un nouvel environnement, cette méthode lui permettra
d'écarter d'emblée certains génotypes qui donneront une
production faible. En lieu et place de longs essais multilocaux ou pluriannuels
ou d'une tentative de paramétrisation d'un modèle de simulation
de cultures qui implique un coàut élevé. Son attention
sera portée par la suite sur l'ensemble restreint des génotypes
retenus avec APLAT o`u il pourra appliquer les schémas classiques de
sélection.
Cette méthode évite ainsi de recourir soit a` de
longs essais multilocaux et pluriannuels, soit a` la paramétrisation
coàuteuse d'un modèle de simulation de culture. De plus, elle
permet d'assumer le choix des lieux comme devant expliquer largement la
diversitédu milieu plutôt que de la représenter a` travers
un sondage équiprobable. En effet, avec APLAT, seuls les résidus
de
la modélisation de l'interaction G×E
sont aléatoires, et non pas l'interaction toute entière.
Chapitre 4
La méthode APLAT-mixte
Dans ce chapitre, nous partirons de la m'ethode APLAT, qui
nous le rappelons, consiste a` lin'eariser autour du vecteur de
paramètres d'un g'enotype de r'ef'erence, la r'eponse de g'enotypes
pr'edite par un modèle de simulation de cultures.
Notre hypothèse pour cette m'ethode, 'etait qu'un
modèle de simulation de cultures, qui est une fonction des
paramètres des g'enotypes et des caract'eristiques des environnements
o`u nous voulons faire la pr'ediction, permet de capter la majeure partie de
l'effet al'eatoire de l'environnement. Ce qui autorise la r'eduction des
interactions al'eatoires G×E, et par là, facilite
la s'election vari'etale.
Dans cette partie, nous revenons sur cette hypothèse
faite en première approximation et consid'erons que certes, si l'al'ea
de l'environnement peut être pris en compte par un modèle de
simulation de cultures, il ne l'est toutefois pas totalement. Ainsi, si nous
pensons toujours pouvoir mod'eliser les variations de la r'eponse d'un
g'enotype dans un environnement par le biais de tels modèles de
simulation, il subsistera n'eanmoins de l'al'ea environnemental,
responsable d''eventuelles interactions
G×E, dont il conviendra d'estimer sa variance, le cas
'ech'eant.
Ce chapitre traite alors de notre deuxième m'ethode
mise au point pour estimer les paramètres fixes issus de la
lin'earisation du rendement des g'enotypes par le modèle SarraH et les
composantes de variance de l'al'ea environnemental restant. Avec les nombreux
paramètres de SarraH, ce qui est le cas pour la plupart des
modèles de simulation de cultures, nous devons identifier un
modèle o`u un nombre important de r'egresseurs et des composantes de
variance coexistent. Nous avons a` cet effet, propos'e une m'ethode combin'ee
de r'egression PLS et de modèle mixte pour l'estimation des
paramètres inconnus. Il s'agit d'une extension de la m'ethode APLAT
propos'ee au pr'ec'edent chapitre o`u cette fois-ci, il est not'e la pr'esence
d'effets al'eatoires additionnels dont nous nous attacherons a` estimer les
variances. Cette m'ethode a 'et'e d'enomm'ee APLAT-Mixte.
Nous d'ebutons ce chapitre par la Section 4.1 o`u nous faisons
un retour sur le modèle mixte. Si les composantes de variance 'etaient
connues, c'est-àdire s'il y avait uniquement une contrainte a` savoir le
nombre important de r'egresseurs par rapport aux observations, une extension de
la m'ethode PLS devrait suffire pour r'esoudre le problème. Une telle
proc'edure est alors d'etaill'ee a` la Section 4.2. Comme ces composantes sont
est en fait souvent inconnues, une m'ethode combin'ee de PLS et d'algorithme EM
est pr'esent'ee a` la Section 4.3 pour estimer les paramètres inconnus.
Les d'eveloppements de cette m'ethode appel'ee PLS-Mixte, sont fond'es dans un
premier temps, sur un modèle mixte o`u des effets al'eatoires sont
suppos'es simplement de variance
ó2I. Pour l''eprouver, nous
nous sommes 'eloign'es de nos donn'ees qui ne s'y prêtent pas et avons eu
recours a` des donn'ees de NIRS. Ensuite, puisque nos donn'ees d'interaction
G×E peuvent être appr'ehend'ees a` travers un
modèle mixte o`u les effets al'eatoires sont suppos'es de variance
ó2Ä, nous avons
adapt'e la m'ethode PLS-Mixte a` ce type de modèle.
4.1 Le modèle mixte
Consid'erons un modèle lin'eaire mixte comme d'ecrit
par McCulloch et Searle (2001). Le vecteur des observations Y
de dimension n × 1 est suppos'e suivre le
modèle suivant :
Y = Xâ + Zu + e (4.1)
o`u â d'ordre p est le vecteur de
paramètres des effets fixes, u d'ordre q
le vecteur de paramètres des effets al'eatoires, X
et Z deux matrices d'incidence connues, et e
le vecteur d'erreur al'eatoire.
Dans ce modèle, si nous avons r effets
al'eatoires, Zu peut être d'ecompos'e comme suit :
Zu = [ Z1 · · ·
Zr ]
ur
o`u uk d'ordre qk, est le
vecteur des effets al'eatoires pour le facteur k avec les
suppositions i(uk) = 0, Var(uk) = ó2
kIqk ? k, et Cov(uk,u' k')
= 0 pour k =6 k',
q= Xr qk
k=1
Aussi i(e) = 0, Var(e) =
ó2 eIn et Cov(uk,
e') = 0 ? k. La fonction i(·)
indique l'esp'erance.
En posant u0 = e,
q0 = n et Z0 =
In comme dans la pr'esentation de Rao et Kleffe (1988),
l''equation (4.1) devient
Y = Xâ + Xr Zkuk
(4.2)
k=0
et V = Xr ZkZ'
ió2 k
k=0
L'estimation des paramètres â et
ó2 k peut se faire concomitamment au
moyen des méthodes de vraisemblance ML, Maximum likelihood
ou REML, Restricted or residual maximum likelihood.
Pour chacune de ces méthodes, une fonction log-vraisemblance est
maximisée par rapport aux paramètres inconnus. La fonction
log-vraisemblance pour la méthode ML (REML étant une variation de
ML) s'écrit,
l = (-1/2)[log |V| + (Y -
Xâ)'V-1(Y
- Xâ) + n log(2ð)]
En dérivant la fonction l par rapport a`
â et a` chacun des ó2 k et en
annulant ces dérivées, nous obtenons r + 1
équations pour ó2 k et
une équation pour â.
|
? ?
?
|
?l/?â =
2X'V-1Y -
2X'V-1Xâ
[ ]
?l/?ó2 k = -(1/2)
tr(V-1V' k) - (Y -
Xâ)'V-1V'
kV-1(Y - Xâ)
|
oiV' k = ?V/?ó2
k
Les solutions de ce système d'équations ne sont
généralement pas obtenues de façon explicite. Pour
résoudre ce problème d'optimisation, l'on a recours a` des
algorithmes itératifs tels que celui de Newton-Raphson ou l'algorithme
EM, Expectation maximization.
Ces deux méthodes d'itérations requièrent
des valeurs initiales pour les pa-
ramètres inconnus. L'algorithme EM
permet de s'approcher de la région de
l'optimum plus rapidement mais la progression vers l'optimum
est lente. Au contraire, celui de Newton-Raphson, malgréqu'il soit
instable loin de l'optimum, permet une convergence vers celui-ci beaucoup plus
rapidement une fois dans sa région.
L'algorithme de Newton-Raphson utilise un développement
de premier ordre de la fonction score c'est-à-dire du gradient de la
fonction log-vraisemblance autour de l'estimation du paramètre a` la
me itération pour fournir l'estimation a` la
(m + 1)e itération. Chaque étape dans
l'algorithme nécessite le calcul de la fonction score et de sa
dérivée, la matrice hessienne de la logvraisemblance.
L'algorithme EM (Meng and van Dyk, 1997) permet l'estimation
de paramètres dans des modèles avec des données
incomplètes. L'argumentaire de l'utilisation de cet algorithme dans le
cadre du modèle mixte est fourni en détail par Searle, Cassella
et McCulloch (1992, pp. 297-303). Ainsi, les effets aléatoires sont-ils
vus comme des données non observées. Searle et al.
considèrent alors que si ces effets aléatoires étaient
connus, l'estimation des paramètres inconnus pourrait facilement se
faire. En effet, il suffirait d'adopter une démarche a` deux
étape.
D'abord, estimer la variance de chaque effet aléatoire
par
bó2 k = (1/qk) Xqk (uk
- uk)2 = (1/qk) Xqk u2
k = u' kuk/qk k=1 k=1
o`u uk d'ordre qk est
supposégaussien d'espérance nulle et de variance
ó2 k.
Ensuite, déduire ces effets aléatoires
supposés connus du vecteur des données Y et
appliquer une régression OLS, Ordinary least squares,
sur le modèle suivant
Y - Xr Zkuk ~ N(Xâ,
ó2 0In) k=1
Mais comme ces effets al'eatoires ne sont pas connus en
r'ealit'e, l'algorithme EM permet de calculer les valeurs conditionnelles
de u'iui a` utiliser a` la place
de u'iui et les valeurs
conditionnelles de ui a` la place de ui.
Selon la terminologie de l'algorithme EM, dans le cas du
mod`ele mixte, les donn'ees observ'ees Y sont appel'ees
donn'ees incompl`etes et Y en plus des effets al'eatoires non
observ'es u1, . . . , ur
constituent les donn'ees compl`etes. Nous rappelons ci-dessous cet algorithme
pour l'estimation des param`etres fond'ee sur une variation de ML publi'ee par
Laird (1982). Les valeurs calcul'ees ók
2(m) de óz sont
obtenues apr`es la me it'eration et sont utilis'ees
pour la mise a` jour de la variance
V-1(m).
'Etape 0 Poser m = 0 et choisir
des valeurs initiales 2(0)
ók
'Etape 1 ('Etape-E) Calculer
Q(ó2 |
ó2(m))
=Eó2(m)(u'kuk
| Y)
= qkók
· ak
2(m) + 4(m)
[Y'P(m)ZkZ'kP(m)Y
-
tr(Z'kV-1(m)Zk)1
o`u P(m) =
V-1(m) -
V-1(m)X(X'V-1(m)X~-X'V-1(m)
'Etape 2 ('Etape-M) Determiner
ó2k(m+1) qui maximise
Q(ó2 |
ó2(m)) c'est-`a-dire,
tel que
Q(ó2(m+1) |
ó2(m))
Q(ó2 |
ó2(m)). Alors,
ók2(m+1)
= Eó2(m)(ukuk |
Y)/qk for k = 0, 1,···, r
'Etape 3 A la convergence c`ad
L(ó2k (m+1) | Y)
-L(ó2k (m)
|Y) ç o`u
ç est une quantit'e arbitrairement petite
et L la fonction de vraisemblance, prendre
bó2k =
ók2(m+1) et alors calculer
Xbâ =
X(X'V-1(m+1)X)-X'V-1(m+1)Y
sinon ajouter 1 a` m et
retourner a` l''Etape 1.
A l''Etape-E, calculer l'esp'erance de la
vraisemblance conditionnelle fond'ee sur
fY,u1,··· ,ur,la
fonction de densit'e des donn'ees compl`etes sachant les donn'ees incompl`etes
est 'equivalent a`
calculerEó2(m)(u'kuk
| Y).
4.2 La r'egression PLS sur un modèle de variance
connue
La régression PLS est une méthode d'estimation
particulière utilisée pour les modèles linéaires
avec l'éventualitén < p. Pour ce type
d'analyse, l'objectif est de prédire Y par des
combinaisons linéaires des colonnes de X
appelées variables latentes. Il est habituellement mis en
oeuvre a` l'aide de l'algorithme NIPALS, Nonlinear estimation by
iterative partial least squares (Wold 1966; de Jong 1993) o`u le
calcul des variables latentes est effectuésimultanément avec un
ensemble de régressions par OLS. Cependant, ces régressions sont
adéquates seulement dans le cas des erreurs sur Y
iid.
L'algorithme PLS appliquéa` un modèle de
variance connue est effectuéen remplaçant les régressions
OLS sur les variables latentes par des régressions GLS, General
least squares, dont voici la description.
1. Centrer et éventuellement réduire X
et Y : x0 = X,
y0 = Y
2. Pour h = 1,··· ,H avec
1 H rang(X)
(a) Calculer les p-vecteurs wh =
[w1 h · · · wp
h]' V/X
o`u wp h = Cov(xp h, yh)/
Cov2(xp h, yh) et
xp h la pe colonne de
xh
p
(b) Normer wh : wh = wh/ wh
II
(c) Calculer les variables latentes PLS th =
xh-1wh
(d) Calculer ch par régression GLS de
yh-1 sur th
yh-1 = thch + yh o`u
Var(yh-1) = V ch =(t'
hV-1th)-t'
hV-1yh-1
(e) Calculer ph par régression de
xh-1 sur th
)-1t'
xh-1 = thp'
h + xh d'o`u p'
h =(t' hth hxh-1
(f) Calculer les résidus xh et yh
(g) Alors Y = t1c1 +
·
·
· + thch + yh
Ainsi, le seul changement par rapport a` l'algorithme PLS
classique est le remplacement de la régression OLS par la
régression GLS au point 2.(d).
4.3 La méthode PLS-Mixte
Les méthodes de vraisemblances, ML ou REML, comme
techniques pour estimer les paramètres fixes et les composantes de
variance dans un modèle linéaire mixte, ne sont applicables que
dans le cas classique o`u le nombre de régresseurs est faible devant le
nombre d'observations, c'est-à-dire n > p. Dans ce
cas, comme nous l'avons vu, un algorithme itératif tel que l'algorithme
EM est nécessaire pour obtenir l'estimation des paramètres
inconnus.
Pour traiter du cas n < p, nous proposons
d'imbriquer une méthode de réduction de dimension telle que la
régression PLS dans l'algorithme EM. Puisqu'il s'agira d'estimer des
composantes de variance dans un contexte de réduction de dimension, nous
avons appelécette technique PLS-Mixte.
Avant cette méthode proposée, dans les
modèles o`u il y avait plus de régresseurs que d'observations et
plusieurs sources de variation, l'estimation des paramètres inconnus se
faisait simplement par régression PLS, c'est-à-dire sans
tenir compte précisément des sources de variation.
Aussi, avons-nous comparél'estimation faite par simple
régression PLS a` celle faite par notre méthode en
utilisant le critère MSEP, Mean square error of
prediction, dans les différentes applications de ce chapitre.
La question de la convergence sera abordée plus loin.
4.3.1 La méthode PLS-Mixte sur un modèle a`
effets aléatoires indépendants de variances homogènes
Nous nous plaçons dans le cadre du modèle 4.2 et
considérons que n < p. Nous proposons la
méthode PLS-Mixte qui consiste donc a` imbriquer une méthode de
réduction de dimension telle que la régression PLS dans
l'algorithme EM. Cette méthode d'estimation est fondée sur ML et
ses variantes. L'estimation par ML est réalisée en maximisant la
vraisemblance de Y par rapport aux paramètres
inconnus.
Suivant l'algorithme EM, nous prenons des valeurs de
départ pour les paramètres inconnus. Ces valeurs permettent de
calculer les variables latentes obtenues a` partir du modèle (X,
Y) vu que la variance V(0) est connue, les
valeurs initiales ó2(0)
k étant choisies. Sur ces variables
latentes, sont calculées les composantes de
varianceó2(1)
k comme dans le cas classique de
l'algorithme
EM. Avec les valeurs ó2(1)
k , ces étapes sont
répétées avec les composantes
ó2(0)
k
remplacées par les estimations courantes
ó2(1)
k . Ce processus itératif est alors
continuéjusqu'àconvergence. Nous décrivons
ci-dessous l'algorithme de la méthode proposée avant de le
détailler plus loin.
'Etape 1 ('Etape-E) Centrer et reduire
X et Y
'Etape 1.1 Reduire la dimension de l'espace des
regresseurs en
determinant les h variables latentes
T(m) =
[t(m)
1 · · ·
t(h
:m)]
vu que la variance
V(m) est connue 'Etape
1.2 Calculer
Q(ó2 |
ó2(m))
=Eó2(m)(u'iuk
| Y)
= qkó2(m) k +
ó4(m)
[Y'P(m)ZkZ'kP(m)Y
- tr(ZkV-1(m)Zk)1
o`u P(m) =
V-1(m)-V-1(m)T(m)(T(m)'V-1(m)T(m))-T(m)'V-1(m)
'Etape 2 ('Etape-M) Determiner
ó2k(m+1) qui maximise
Eó2(m)(u'iuk
| Y) c`ad, tel
que
QT(m)(ó2(m+1)
| ó2(m)) %
QT(m)(ó2
| ó2(m))
ók2(m+1)
=
Eó2(m)(u'iuk
| Y)/qk pour k = 0, 1, · ··, r
'Etape 3 Si convergence, prendre
bó2k =
ók2(m+1) et alors calculer
X-â =
X(X'V-1(m+1)X)-X'V-1(m+1)Y
sinon ajouter 1 a` m et
retourner a` 'Etape 1.
Les variables latentes sont ainsi recalculees au debut de
chaque iteration dans l'algorithme EM avec les composantes de variance mises a`
jour. Cet algorithme de la methode PLS-Mixte fondee sur une variation de ML est
detaillede la facon suivante.
'Etape 1 Centrer et reduire X
et Y : x0 = X,
y0 = Y
'Etape 1.1 Pour h = 1,2, ... ,
rang(X)
(a) Calculer les p-vector wh =
[w1h · ·
· wph]'
o`u wph =
Cov(xph,yh)/ E
Cov2(xph,
yh) et xph la
pe
p
colonne de xh
(b) Normer wh : wh = wh/ II wh
II
(c) Calculer les variables latentes
t(m)
h = xh-1wh
(d) Calculer ch par regression GLS de
yh-1 sur t(m)
h
|
yh-1 =
t(h
m)ch + yh o`u
Var(yh-1) = V(m)
=
|
Xr
k=0
|
ZkZ'kók2(m)
|
ch =~t(m)'
h
V-1(m)t(m)
~-t(m)'
h
V-1(m)yh-1
h
(e) Calculer ph par regression de
xh-1 sur t(m)
h
~-1t(m)'
xh-1 =
t(m)
h p' h + xh d'o`u
p' h =~t(m)'
h t(m) h
xh-1
h
(f) Calculer les residus xh and yh
(g)
Xr
i=0
Ziui
Finalement Y =
T(m)C +
o`u T(m)
=[t(m)
1 ···t(h
m)] et C =
[c1 · · · ch]'
'Etape 1.2 Calculer
2m+1)
ók( = C r k
2(m) + (ó
k(m) / qk)
[Y'P(m)
Z
kZ'kP(m)Y
- tr (Z'kV -1(m)
Zk)1
o`u P(m) =
V-1(m)-V-1(m)T(m)(T(m)'V-1(m)T(m))-T(m)'V-1(m)
'Etape 2 Si convergence, prendre
bó2k =
ó2k(m+1) ; sinon ajouter
1 a` m
et retourner a` l''Etape 1.1
La procedure REML, quant a` elle, maximise la vraisemblance de
certaines combinaisons lineaires des elements de Y par rapport
aux param`etres inconnus (McCulloch and Searle, 2001). Notre methode fond'ee
sur cette procedure REML est effectuee en remplacant l''Etape 1.2
par
2(m+1)
ói =
ó(m) +
(ó4(m) /qi)
[Y'P(m)ZiZ'iP(m)Y
-
tr(Z'iP(m)Zi)]
o`u P(m) reste
defini comme ci-dessus.
Convergence de l'algorithme de la m'ethode
PLS-Mixte :
Les proprietes de convergence de l'algorithme EM restent
valides avec cette methode PLS-Mixte, en particulier sa convergence monotone.
Par exemple, pour la monotonocitede l'algorithme EM, nous devons montrer que la
fonction de vraisemblance, L(ó2 |
Y) ne decroit pas apr`es une iteration, c'est-`adire,
LT(m)(órm+1)
| Y) LT(m-1)(ói
2(m)| Y)
(4.3)
o`u la valeur de la vraisemblance
LT(m)(ó2(m+1) | Y)
est calculee a` la (m + 1)e
i
iteration en utilisant les valeurs actuelles de
T(m) comme regresseurs et la valeur de la
vraisemblance LT(m-1)(ói 2(m)
| Y) calculee a` la me iteration avec les
valeurs de T(m-1)
Pour l'algorithme EM classique, le fait que la vraisemblance
augmente a` chaque iteration est un resultat bien connu qui a etemontrepar
Dempster, Laird et Rubin (1977) et discuteplus tard par Wu (1983) et par
McLachlan et Krishnan (1997).
Nous nous sommes fond'es sur ce resultat pour montrer
l'inegalite(4.3). La fonction de densiteconditionnelle des donnees
compl`etes [Y, u'] o`u
u' = [u'
1,· · ·
,u'r] sachant
les donnees incompl`etes Y est egale a`
k([Y,u'] | Y,
ó2) =
f[Y,u']([Y,u']
| ó2)
f(Y | ó2)
(4.4)
Alors la fonction log-vraisemblance des donnees incompl`etes peut
s'ecrire comme suit :
l(ó2 | Y) = ln (f(Y |
ó2))
= ln
(f[Y,u']([Y,
u'] | ó2)/k([Y,
u'] | Y,
ó2))
= ln
(f[Y,u']([Y,
u'] | ó2)) -- ln
(k([Y, u'] | Y,
ó2)) =
l[Y,u'](ó2
| [Y, u']) --
ln(k([Y,u'] | Y,
ó2))
L'esperance de cette equation est prise par rapport a` la
distribution conditionnelle des donnees compl`etes sachant les donnees
incompl`etes, et en utilisant
ó2(m) a` la place de
ó2. Ainsi,
Eó2[l(ó2
| Y) | Y1 = lT(m-1)(ó2 | Y)
(4.5)
=
IEó2(m)
[l[Y,u'](ó2
| [Y,u']) | Y1
--Eó2(m)
[ln(kT(m-1)([Y,u'] | Y,
ó2)) | Y1
Nous avons, a` l''Etape-E de l'algorithme du mod`ele PLS-Mixte
avec une variance connue (section 2.2), la relation suivante :
Eó2(m)
[l[Y,u'](ó2
| [Y,u']) | Y1
=Eó2(u'u
| Y)
= QT(m-1)(ó2 |
ó2(m))
et en posant
GT(m-1)(ó2 |
ó2(m)) =
[Eó2(m)
[ln(kT(m-1)([Y,u'] |
Y,ó2)) | Y1
l'equation (4.5) devient
lT(m-1)(ó2 | Y) =
QT(m-1)(ó2 |
ó2(m)) --
GT(m-1)(ó2 |
ó2(m))
Nous pouvons maintenant calculer
lT(m)(ó2(m+1)
| Y) - lT(m-1)(ó2(m) | Y) =
QT(m)(ó2(m+1) |
ó2(m))
-
GT(m)(ó2(m+1) |
ó2(m))
-
QT(m-1)(ó2(m) |
ó2(m))
+GT(m-1)(ó2(m) |
ó2(m))
La quantit'e
QT(m)(ó2(m+1) |
ó2(m))-QT(m-1)(ó2(m)
| ó2(m)) est positive
car ó2(m+1)
est choisie tel que
QT(m)(ó2(m+1)
| ó2(m)) =
QT(m-1)(ó2 |
ó2(m)) ? ó2
Et, ? ó2
GT(m-1)(ó2 |
ó2(m)) -
GT(m-1)(ó2(m) |
ó2(m)) =
Eó2(m)
[ln(kT(m-1)([Y,u'] | Y,
ó2)) | Y]
-Eó2(m)
[ln(kT(m-1)([Y,u'] |
Y,ó2(m))) | Y]
= ln(1)
= 0
Ce r'esultat est montr'e par McLachlan et Krishnan (1997) pour
l'algorithme
EM classique o`u les r'egresseurs sont consid'er'es comme
fixes. Ici, la quantit'e
GT(m-1)(ó2 |
ó2(m)) -
GT(m-1)(ó2(m) |
ó2(m))
est prise, ? ó2, par rapport
aux màemes r'egresseurs
T(m-1). Ce qui ne change donc
pas le r'esultat.
Nous avons alors
lT(m)(ó2(m+1)
| Y) - lT(m-1)(ó2(m) | Y) ~
0
La m'ethode PLS-Mixte, utilise entre autres, la technique de
r'eduction de dimension. Et dans ce sens, il faudra d'eterminer la dimension du
modèle retenue. Pour cela, nous choisissons un nombre maximum h
(h < rang(X)) de variables latentes au d'epart. La m'ethode
it'erative d'ecrite plus haut permettra de calculer et d'actualiser tour a`
tour ces h variables latentes et les composantes de variance.
Avec ces composantes de variance estim'ees au final sur le modèle a`
h variables latentes, les PRESS, Prediction error sum
of squares (Stone 1974), des h sous-modèles
avec respectivement 1, 2,
·
·
·,
h r'egresseurs sont calcul'es. La dimension retenue est celle
du sous-modèle a` plus faible PRESS.
Illustration avec des données de NIRS
Les algorithmes pr'esent'es ci-dessus sont test'es et
appliqu'es a` titre illustratif sur un jeu de donn'ees de Rami, Dufour,
Trouche, Fliedel, Mestres, Davrieux, Blanchard et Hamon (1998).
Il s'agit d'une population de lign'ees recombinantes de sorgho
'etudi'ee a` la station exp'erimentale de l'INERA au Burkina Faso. Cette
population est obtenue par une m'ethode »modified single-seed
descent» avec le g'enotype IS 2807 (collection de l'ICRISAT) consid'er'e
comme femelle crois'ee avec le g'enotype 249 (collection du CIRAD) consid'er'e
comme màale. Cette population de 90 individus fut r'ecolt'ee en 1995 a`
la g'en'eration F7, a` partir d'un dispositif exp'erimental en lattice
9x10 avec trois r'ep'etitions.
Nous avons seulement retenu la teneur en prot'eine parmi un
ensemble de mesures biochimiques effectu'ees sur les lign'ees, pour nous
conformer aux conditions de notre m'ethode; c'est-à-dire qu'il n'est
consid'er'e qu'une seule variable a` expliquer.
Pour cette teneur en prot'eine, il a 'et'e utilis'e une
m'ethode traditionnelle de mesure, fiable mais longue a` effectuer; les valeurs
mesur'ees sur toutes les lign'ees constituent le vecteur Y.
Parallèlement a` cette m'ethode de mesure, la technique
NIRS, Near infrared reflectance spectroscopy, beaucoup plus
rapide a 'et'e effectu'ee; les spectres d'absorption obtenus pour 1050
longueurs d'onde constituent la matrice X. La s'erie de
longueurs d'onde consid'er'ee est constitu'ee d'une s'equence de 400 a` 1098
par 2 (figure 4.1).
FIG. 4.1 - Spectres d'absorption proches de
l'infrarouge pour une population de lignées de sorgho
récoltées en 1995 a` la Station expérimentale de l'INERA
au Burkina Faso.
500 1000 1500 2000 2500
absorbance
0.1 02 0.3 0.4 0.5 0.6 0.7
longueurs d'onde
Au delàde notre souci d'utiliser ces donn'ees pour
tester et 'eprouver notre m'ethode, l'int'erêt de cette 'etude chez le
praticien, pourrait être de calibrer la technique NIRS sur le sorgho pour
la teneur en prot'eine dans le but d'obtenir une m'ethode de mesure rapide en
tenant compte de la structure particulière de la covariance des erreurs
induite par le dispositif exp'erimental.
Pour la mise en oeuvre de la m'ethode PLS-Mixte sur ces
donn'ees, il existe cependant des donn'ees manquantes a` savoir la
troisième r'ep'etition et trois lign'ees des deux autres r'ep'etitions.
Eu 'egard au nombre important de longueurs d'onde (1050) et la corr'elation
massive habituellement constat'ee sur les donn'ees de NIRS, nous avons d'ecid'e
de conserver syst'ematiquement une longueur d'onde toutes les cinq.
Le modèle consid'er'e pour ces donn'ees s''ecrit comme
suit :
Y = Xâ +
Z1u1 +
Z2u2 + e
(4.6)
o`u u1 d'ordre 2 est l'effet al'eatoire de la
r'ep'etition et u2 d'ordre 20 l'effet al'eatoire du bloc
hi'erarchis'e dans la r'ep'etition.
Finalement, la dimension de Y est
174×1 et la dimension de X est
174×210. Aussi, Z1 est une matrice
174×2 et u1 un vecteur
2×1 tandis que Z2 est une matrice
174×20 et u2 un vecteur
20×1.
Les hypothèses pour ce modèle sont par
cons'equent
|
? ????
????
|
u1 ~
N(0,ó21I2)
u2 ~ N(0, ó2
2I20)
u0 ~ N(0,ó2
0I174) avec u0 = e
|
Nous avons donc pour ce modèle, plus de r'egresseurs
que d'observations (210 contre 174) et trois composantes de variance a` estimer
(ó21 pour l'effet al'eatoire de
la r'ep'etition, ó2 2 pour l'effet al'eatoire du bloc
et ó2 0 pour l'erreur r'esiduelle al'eatoire). Nous
allons donc avoir recours a` la m'ethode PLS-Mixte fond'ee sur ML et sur REML
pour estimer les quatres paramètres â,
ó2 1, ó2 2 et
ó2 0.
Pour le calcul du PRESS, nous avons, comme annonc'e plus haut,
choisi un
nombre maximum de 10 variables latentes. Avec un modèle de
dimension 10,
nous avons utilis'e la m'ethode PLS-Mixte pour estimer les
trois composantes de variance. Par la suite, pour chacun des
sous-modèles de dimension 1 a` 10, nous avons enlev'e successivement
chaque lign'ee (dans les deux r'ep'etitions) dont nous avons pr'edit sa teneur
en prot'eine par les lign'ees restantes en utilisant une r'egression GLS avec
les composantes de variance estim'ees sur le modèle de dimension 10.
Le PRESS d'ecroàýt continuellement avec le
nombre de variables latentes pour les deux modèles fond'es sur ML et sur
REML quand le nombre de composantes maximum avait initialement 'et'e fix'e a`
10. Cependant, nous avons d'ecid'e pour ces modèles de r'eduire leur
espace a` 7 dimensions 'etant donn'e que le PRESS n'y est pas trop diff'erent
de ses valeurs minimales (figure 4.2).
FIG. 4.2 - 'Evolution du PRESS selon le nombre de
variables latentes quand le nombre maximal de variables latentes avait
initialement étéfixéa` dix pour les données de
NIRS.
ML REML
PRESS
20 25 30 35
2 4 6 8 10
2 4 6 8 10
20 25 30 35
PRESS
Par rapport a` l'objectif de cette 'etude, il serait question
de s'electionner les r'egresseurs influents. Nous pouvons alors pour cela,
consid'erer les coefficients les plus grands en valeur absolue des r'egresseurs
pour le modèle s'electionn'e. Nous notons ainsi que les bandes
d'absorption localis'ees autour de 2210, 1940, 1690 et 1450 ont les plus grands
coefficients (figure 4.3).
FIG. 4.3 - 'Evolution des
coefficientsâb
(modèle (4.6)) selon les différentes longueurs d'onde pour les
modèles a` 7 variables latentes fondés sur ML et REML des
données de NIRS.
ML REML
.9 02
02
0.0 0.1
-0 -02 .1 -0
-0.9
02
02
00 0.1
-02 -0.1
500 1000 1500 2000 2500 500 1000 1500 2000 2500
wavelengths wavelengths
Les estimations de ó k pour
ML et pour REML sont montr'ees au tableau 4.1. Nous notons que la variance de
l'effet bloc hi'erarchis'e dans la r'ep'etition est deux fois plus faible que
celle de l'effet r'ep'etition.
ML REML
ó 1 0,11273
0,11276
ó 0,06248 0,06309
ó
0 0,46839 0,48917
Estimation des composantes de variance pour les
modèles a` 7 va-
TAB. 4.1 - riables latentes fondés sur ML et REML
des données de NIRS.
Au tableau 4.2, nous avons le MSEP du modèle 4.6
analys'e par r'egression PLS et le MSEP du même modèle analys'e
par la m'ethode PLS-Mixte fond'ee sur ML et sur REML. La pr'ediction par la
m'ethode PLS-Mixte, comme nous nous y attendons, de la teneur en prot'eine du
sorgho par les bandes d'absorption, quand il y a plusieurs sources de
variation, est meilleure que celle faite par simple r'egression PLS.
MSEP
ML 0,64637
PLS-Mixte REML 0,64639
PLS 0,91479
TAB. 4.2 - MSEP des modèles de la méthode
PLS-Mixte et de la régression PLS pour les données de
NIRS.
Toutefois, la détermination de la dimension optimale a`
travers le calcul du PRESS est ici liée a` h, le nombre
maximum de variables latentes choisi au départ. En effet, les
composantes de variance sont estimées avec les h
variables latentes et utilisées par la suite respectivement
pour chaque sous-modèle a` 1, 2, ..., h
variables latentes pour estimer â et finalement pour calculer
les PRESS. Cependant, le choix de h semble ne pas avoir un
impact important sur le calcul des PRESS dans notre example. A la figure 4.4,
nous avons présentéles valeurs des PRESS calculées pour un
nombre maximum de variables latentes choisi initialement pour être
égal respectivement a` 10, 20, 30, 40 et 50. L'évolution du PRESS
jusqu'àla dixième variable latente n'est quasiment pas
affectée par le choix initial du nombre maximum de variables
latentes.
4.3.2 La méthode PLS-Mixte sur un modèle a`
effets aléatoires corrélés de variances
hétérogènes
La méthode PLS-Mixte, présentée ci-dessus
et appliquée aux données de NIRS, a
étéécrite avec les hypothèses classiques de
normalitédes effets aléatoires et surtout de la forme
particulière des matrices de variance de ces effets aléatoires.
En effet, il a fallu supposer que cette variance pour chaque effet
aléatoire Uk était de la forme
ó2 kIqk.
Ici, nous allons légèrement relâcher cette
hypothèse et considérer que la va-
riance pour chaque effet
aléatoire pourrait être de la forme ó2
kAk o`u Ak est
Variation du PRESS selon les dix premières
variables latentes
FIG. 4.4 quand le nombre maximum de variables a
initialement étéfixéde façon successive a` 10, 20,
30, 40 et 50 pour le modèle fondésur
ML pour les données de NIRS. Le modèle
fondésur REML donne des résultats voisins de ceux
présentés ici.
ML
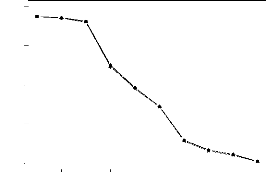
20 25 30 35 40
PRESS
2 4 6 8 10
une matrice quelconque symétrique connue. Nous allons
alors voir comment la méthode PLS-Mixte s'écrira avec ces
nouvelles données du problème.
Nous nous plaçons toujours dans le cadre du modèle
(4.2) que nous allons rappeler.
Y = Xâ + Xr ZkUk
(4.7)
k=0
Nous supposons cette fois-ci donc que Uk ~ N(0,
ó2 kÄk), d'o`u V = Xr
ZkÄkZ' kó2
k.
k=0
Avant de présenter notre méthode avec un
modèle o`u n < p et la variance
V
des observations écrite comme ci-dessus, nous traiterons dans
un premier
temps, du cas plus simple n > p o`u nous
verrons comment s'effectuent les estimations des paramètres inconnus
â et ó2 k a` l'aide de
l'algorithme EM fondésur ML.
Le modèle mixte a` effets aléatoires
corrélés
L'algorithme EM, nous le rappelons, permet de calculer
l'espérance condi-
tionnelle des effets aléatoires sachant les
données incomplètes Y. Pour cela, il
est
nécessaire d'avoir la loi jointe de Y et u = [
u'1 u' 2 ... u' r
]'. Nous avons
Cov(Y, u' k') = Cov(Xâ
+ Xr Zkuk,u' k') =
Zk'Cov(uk',u' k') = ó2
k'Zk'Äk' (4.8)
k=0
car Var(uk) = ó2 kÄk V
k, et Cov(uk,u'k') = 0
pour k =6 k'.
Ainsi, avec les suppositions du modèle
(4.7), la loi jointe de Y et u
est gaussienne de la forme .IV(12,Ó)
o`u
|
12 =
|
Xâ
0
...
0
|
?
? ? ? ? et Ó =
?
|
" #
V Cov(Y,u')
Cov(u,Y') Var(u)
|
(4.9)
|
avec Cov(Y,u') = [
ó2 1Z1Ä1 . . .
ó2 rZrÄr ]
?ó21Ä1Z'1
?
Cov(u,Y') = ? ..
? .
ó2
rÄrZ' r
ó2 1Ä1
et Var(u) = 0 ... 0
ó2 rÄr
Ce qui implique la fonction de densitésuivante
Pr
fY,u1,···
,ur(Y, u1, · · · ,
ur) = (2ð)-
1 k=0 qk|Ó|-
1
o`u
2 2 exp(-1 2Q) (4.10)
|
[ ]
Q = (Y - Xâ)'
u' 1 · · · u'
Ó-1
r
|
Y -
Xâ
u1
...
|
(4.11)
|
ur
Nous allons donc avec les développements qui vont
suivre, trouver les valeurs de |Ó| et de
Ó-1 dans le but de simplifier la
densité(4.10). Il s'agira par la suite, de
dériver tout simplement cette densité, pour avoir les estimations
des paramètres.
Pour calculer |Ó|, nous faisons appel au
résultat déjàétabli (Searle et alp 453, 1992) : Si
A, B, C et D
sont quatre matrices avec les bonnes dimensions, alors
|
~~~~~
|
A B
C D
|
~~~~~
|
= |D||A - BD-1C|
|
|
Ainsi,
|Ó| =
|
~~~~~
|
V Cov(Y,u')
Cov(u,Y') Var(u)
|
~~~~~
|
= |Var(u)||V - Cov(Y,
u')(Var(u))-1Cov(u,
Y')|
|
|
|Var(u)| =
|
~~~~~~~~
|
ó21Ä1
0 ... 0
ó2 rÄr
|
~~~~~~~~
|
=
|ó21Ä1| ×
··· × |ó2
rÄr| =
|
Lr
k=1
|
(ó2
k)qk|Äk|
|
Or,
car chaque matrice Äk est carree de nombre
de lignes qk D'autre part,
Cov(Y,
u')(Var(u))-1Cov(u,
Y')
|
= [
ó21Z1Ä1
··· ó2 rZrÄr ]
|
ó1
2Ä1-1
0
·
·
·
0
ó-2
r Ä
|
-1 r
|
ó2
1Ä1Z'
1
·
·
·
ó2rÄrZ'r
|
|
=
|
Xr
k=1
|
ó2kZkÄkZ'
k
|
Or,
(Var(u))-1Cov(u,
Y') =
0
·
·
·
0
ó-2
r
Ä
ó21Ä1Z'1
·
·
·
ó2rÄrZ'r
-1
r
ó-2
1 Ä1-1
=
Z'1
·
·
·
Z'r
D'o`u
|Ó| = Yr
(ó2k)qk|Äk||V -
Xr ó2
kZkÄkZ' k|
=
Yr
k=1
k=1 k=1
(ó2k)qk|Äk||ó20Ä0|
Pour calculer Ó-1, nous
utilisons les resultats des inverses generalisees (Searle et al p 450, 1992) et
etablissons que
" #
=
0 0
0 (Var(u))-1
"+
# I
ó,:72Ä:7,
1 [ I -Cov(Y,
u')(Var(u))-1
-(Var(u))-1Cov(u,
Y')
Par ailleurs,
Cov(Y,
u')(Var(u))-1 = [
Z1 · · ·Zr
Par cons'equent,
Ó-1 = Ó1
-1 + Ó-1
2
o`u Ó1 1 =
ó-2
1 Ä-1
1
?
m ?0
... 0
ó-2
r Ä-1
r
I
|
et Ó2 -1 =
|
-
|
Z'1
...
|
h h i i
ó-2
0 Ä-1 I - Z1
· · · Zr
0
|
Z' r
Nous allons donc remplacer Ó-1
par Ó-1
1 +Ó-1
2 dans (4.11) pour avoir la valeur
de Q.
Ainsi,
Q = Q1 + Q2
|
o`u Q1 = [ (Y -
Xâ)'
u'1 · · · u'
Ó-1
r 1
|
Y - Xi3
u1
...
|
ur
|
et Q2 = [ (Y -
Xâ)'
u'1 · · ·
u'Ó-1
r 2
|
Y - Xi3
u1
...
ur
|
m 0
m ?0
?
? ó-2
1 Ä-1
1
... 0
r 2Är-1
ó
Q1 = [ (Y -
Xâ)'
uc. · · ·
u'r ]
Y - Xi3
u1
...
ur
= I 0 [ u'1
· · ·
u'r ]
|
ó-2
1 Ä-1
1
0
|
...
0
ór
2Är-1
|
Y - Xi3
u1
...
ur
|
|
= [ u'1 ·
· · u'r
]
|
ó-2
1 Ä-1
1
0
|
...
0
ór
2Är-1
|
u1
...
ur
|
u'kÄk
1uk
=
ó2
k
Er
k=1
Pour calculer Q2, trouvons d'abord une formule
simplifiée de Ó-1
2
I
Ó-1
2 =
|
-
|
Z'1
...
Z'r
|
ó
0 2Ä,Y1 [ I - [
Z1 ··· Zr ] ]
|
|
Ä01
-Ä,y1 [ Z1
·
1
|
·· Zr ]
|
|
= ó-2
0
|
-
|
Z'1
...
Z'r
|
? ?
Ä-1 ?
0 ?
|
Z'1
...
Z'r
|
Ä01 [ Z1 ·
· · Zr ]
|
|
Ainsi Q2 = ó02
[ Q21 Q22 ]
|
Y - Xi3
u1
...
ur
|
|
?
?
avec Q21 = (Y -
Xâ)'Ä-1
0 - [ u' 1 · · ·
u' r ] ? ?
Ä-1
0 [ Z1 · · · Zr
]
Z' r
Z' 1
...
? ?
et Q22 =
-(Y-Xâ)'Ä-1
0 [ Z1 · · ·
Zr ]+[ u' 1 · · · u'
r ] ? ?
D'o`u,
Z'1
...
Z'r
? ?
Q2 = ó-2
? r ]
0 ?(Y -
Xâ)'Ä-1
0 - [ u' 1 · · ·
u'
? ?
? ?
? Ä-1 ? (Y -
Xâ) ? 0 ?
Z'1
...
Z'r
?
? ? ?
- ó-2
0
? ?
(Y -
Xâ)'Ä-1
0 [ Z1 · · ·
Zr ] - [ u' 1 · · · u'
r ] ? ?
?
? ?Ä-1
0 [ Z1 · · · Zr
]
?
u1
- 2 (Y -
Xâ)'Ä-1
0 -
Xr
k=1
!
u' kZ'
kÄ-1 (Y -
Xâ)
ó0
=
0
- ó-2
0
|
(Y -
Xâ)'Ä-1
0
|
Xr
k=1
|
Zkuk -
|
Er
k=1
|
u'kZ'kÄ0-1
|
Xr
k=1
|
!Zkuk
|
|
!
Xr Xr
Zkuk - ó-2
u' kZ'
kÄ-1 Y - Xâ -
Zkuk
0 0
k=1 k=1
- 2 (Y -
Xâ)'Ä-1
Y - Xâ -
0
Er
k=1
ó
=
0
! Xr
u' kZ'
kÄ-1 Y - Xâ -
Zkuk
0
k=1
- 2 (Y -
Xâ)'Ä-1
0 -
Xr
k=1
= ó 0
|
= ó-2 Y -
Xâ -
0
|
Xr
k=1
|
!' Xr
Zkuk Ä-1 Y - Xâ
-
0
k=
|
1
|
Zkuk)
|
u' kÄ-1
k uk + ó-2 Y -
Xâ -
ó2 0
k
) !
X
Zkuk Ä-1 Y - Xâ
- Zkuk (4.12)
0
k=1
Q =
Xr
k=1
Er
k=1
' r
En ajoutant Q1 et Q2, nous
obtenons
En remplaçant (4.11) dans
(4.9) et en passant au logarithme, nous avons
|
l = -1
2
|
X r
k=0
|
log27r --
) k -
k=0 k=1
1 Er (gkiegOlc +
loglAkl) -- 1 `-r uk'
Ä-1u1 qk '
2
2 L--1
ó2
k
|
Zkuk) Ä0 1 Y
- Xâ-
-2 ó0
2
Y - Xâ -
Xr
k=1
EZkuk)
k=
1

1
-
2
X r
k=0
) 1(qklogó
r r 1.1'
Ä-1uk
qk log27r --
Nlc + log|Äk |) - V k k
2 1---/ 2 L--/ ,.,.2
"
k=0 k=0 k
car Y - X0 - Xr Zkuk = e =
u0
k=1
De ce fait, il suffira de d'eriver la fonction l
ci-dessus par rapport a` chacun des param`etres inconnus du mod`ele
(4.7) pour trouver les estimateurs suivants
= ukÄk
1uk/qk, k = 0,1,· · · r (4.13)
et
XS =
X(X'Ä,y1X)-X'Ä,y1
Y -
k=1 Zkuk) ) (4.14)
Ainsi, comme les uk ne sont pas connus, nous
avons besoin des esp'erances conditionnelles de
u'k k
1uk
et de
r
Y - Zkuk
k=1
sachant les donn'ees incompl`etes Y.
Pour cela, nous pouvons utiliser le r'esultat suivant : si
I # I #
I
x1 u1 V11
V12
~ N ,
x2 u2 V21
V22
alors la loi conditionnelle de x1 sachant
x2 est
x1|x2 ~ N[u1
+ V12V-1
22 (x2 - u2),
V11 - V12V-1
22 V21]
Avec uk ~ N(0,ó2
kÄk) et Y ~
N(Xâ,V), nous avons
I # I #
I #!
uk 0 ó2 kÄk Cov(uk,
Y')
~ N ,
Y Xâ
Cov(Y,u' k) V
D'apr`es (4.8), il vient
uk|Y ~ N (ó2 )
kÄkZ'
kV-1(Y - Xâ),
ó2 kÄk - ó2
kÄkZ'
kV-1ó2 kZkÄk
D'o`u
kV-1(Y -
Xâ)
[(uk|Y) = ó2
kÄkZ' (4.15)
Ensuite, en utilisant le th'eor`eme suivant (Searle p 231,
1987)
Théorème 1
si x ~ N(u, V)
alors[(x'Ax) = tr(AV) +
u'Au ? A
et d'apr`es ce qui pr'ec`ede, nous avons
kÄ-1
kÄ-1
E(u' k uk|Y) =
tr(ó2 kÄ-1
k Äk - ó4 k
ÄkZ'
kV-1ZkÄk)
+ó4k(Y -
Xâ)'V-1ZkÄ;cÄk
1ÄkZ'kV-1(Y
- Xâ)
D'o`u
E(u'kÄ-1
k uk|Y) = tr(ó2kIqk -
ó4kZ'kV-1ZkÄk)
+ó4k(Y -
Xâ)'V-1ZkÄ'kZ'kV-1(Y
- Xâ) (4.16)
Nous pouvons alors établir l'algorithme EM dans le cas
du modèle linéaire mixte o`u chaque effet aléatoire est de
variance
ó2kÄk.
L'estimation, fondée sur ML, s'effectue en plusieurs itérations.
Des valeurs initiales ók 2(0) et
â(0) sont choisies au départ. A la
me itération de l''Etape-E, sont calculées
les espérances conditionnelles suivantes, a` partir de
(4.16),
g(k m) =
E(ukÄk
1uk|Y)|0=0(0) et
óZ=óZ(m) (4.17)
=
tr(ók(m)Iqk)
- ó4(m)
k tr(Z'
kV-1(m)ZkÄk)
+ók
4(m) (Y -
Xâ(m))'V-1(m)ZkÄ'kZ'kV-1(m)(Y
- Xâ(m)) =
qkók + (ó4(m)
2(m) k )
× [ (Y -
Xâ(m))'V-1(m)ZkÄ'kZ'kV-1(m)
(Y - Xâ(m)) -
tr(Z'kV-1(m)ZkÄk)]
Et, a` partir de (4.15)
|
b(m)
=E(Y -
|
Xr
k=1
|
Zkuk|Y)0=0(0) et ó2
(4.18)
k=ó2(m)
k
|
= Y - Xr
ZkÄkZ;có2k
(m)V-1(m)(Y
- Xâ(m))
k=1
= Y - (V(m)
-
ó,!,(m)Ä0)V-1(m)(Y
- Xâ(m))
= Y -
V(m)V-1(m)(Y
- Xâ(m)) +
ó02(m)Ä0V-1(m)(Y
- Xâ(m)) =
Xâ(m) +
ó0
2(m)Ä0V-1(m)(Y
- Xâ(m))
A l''Etape-M, pour trouver les estimateurs, il suffit de
prendre
|
ók
2(m+1)
|
.ik m)/qk
(4.19)
|
= 4,(m) +
(4(m)/qk)
× [(Y -
Xâ(m))'V-1(m)ZkÄ;,Z'kV-1(m)(Y
- Xâ(m)) -
tr(Z'kV-1(m)ZkÄk)]
Et, a` partir de (4.14)
Xâ( 1)
=
X(X'Ä,y1X)-X'Ä,y1
(Xâ(m) +
ó0
2(m)Ä0V-1(m)(Y
- Xâ(m)) )
(4.20)
=
X(X'Ä-1
0
X)-X'Ä-1
0
Xâ(m) +
ó0(m)X(X'Ä,y1X)-X'ÄC,1Ä0V-1(m)(Y
- Xâ(m)) =
Xâ(m) +
4(m)X(X'Ä1X)-X'V-1(m)(Y
- Xâ(m))
A la place d'itérer pour obtenir a` la fois les valeurs
de â et de
ó2k, Laird (1982)
suggère de trouver les valeurs de
ó2k et seulement, a` la fin des
itérations, de calculer la valeur de â. En effet, a` la place de
calculer V-1(m)(Y -
Xâ(m)) dans
l'équation (4.19), il est
calculéP(m)Y
o`u
P(m) =
V-1(m) -
V-1(m)X(X'V-1(m)X)-X'V-1(m)
ne depend pas de â.
En effet, si
Xâ(m) etait l'estimation de
Xâ, c'est-`a-dire
Xâ(m) =
X(X'V-1(m)X)-X'V-1(m)Y
alors
V-1(m)(Y
- Xâ(m)) =
V-1(m)Y -
V-1(m)Xâ(m)
= V-1(m)Y -
V-1(m)X(X'V-1(m)X)-X'V-1(m)Y
= (V-1(m) -
V-1(m)X(X'V-1(m)X)-X'V-1(m))
Y
=
P(m)Y
De ce fait, il ne sera pas necessaire de disposer de
â(m) dans les iterations et a`
l''Etape-E.
Il ne sera alors plus calculequ'une seule esperance conditionnelle
|
ó2(m+1)
k =
|
bs(m)
k /qk (4.21)
|
= ó2k
(m) +
(4(m)/qk) [
Y'P(m)ZkÄ'kZ'kP(m)Y
-
tr(Z'kV-1(m)ZkÄk)]
Nous presentons ci-dessous l'algorithme EM fondesur ML
appliqueaux effets aleatoires corr'el'es du mod`ele mixte.
'Etape 0 Mettre m = 0 et
choisir des valeurs initiales
ó2k(0)
'Etape 1 ('Etape-E) Calculer
Q(ó2 |
ó2(m))
= Eó2(m) (ukÄk
1uk | Y)
= qkók (Irk2(m)
+ 4(m)
[Y'P(m)Zk k
Ä' Z'
P(m)Y --
tr(Z'kV-1(m)ZkÄk)1
o`u P(m) =
V-1(m) --
V-1(m)X(X'V-1(m)X~-X'V-1(m)
'Etape 2 ('Etape-M) Determiner
ó2k(m+1) qui maximise
Q(ó2 |
ó2(m)) c'est-`a-dire,
tel que
Q(ó2(m+1) |
ó2(m)) ?.
Q(ó2 |
ó2(m)). Alors,
ók2(m+1)
= E 2(m)
(ukÄi;1uk | Y)/qk
for k = 0,1,
·
·
· , r
'Etape 3 A la convergence c`ad
L(ó2k (m+1) | Y) --
L(ó2k (m) | Y) ,....
ç o`u
ç est une quantitearbitrairement petite
et L la fonction de vraisemblance, prendre
bó2k =
ók2(m+1) et alors calculer
X-â =
X(X'V-1(m+1)X)-X'V-1(m+1)Y
sinon ajouter 1 a` m et
retourner a` l''Etape 1.
Simulations pour la convergence de l'algorithme EM
appliqu'e aux effets al'eatoires corr'el'es du mod`ele mixte :
Pour tester la convergence de cet algorithme et verifier la
qualitedes estima-
tions, nous avons effectuedes simulations numeriques.
Nous avons considerea` cet effet le modele simple suivant :
Y = X/3 +
Z1u1 + Z2u2
+ Z0u0 (4.22)
o`u la dimension du vecteur des reponses Y
est 120x1, celle de la matrice des observations
X est 120x5 et le vecteur 13
des parametres fixes est de longueur 5. Aussi, avons-nous suppose
|
? ????
????
|
u1 ~
N(0,ó21Ä1)
u2 ~ N (0, ó2
2Ä2)
u0 ~ N (0,
ó2 0Ä0)
|
|
?
o`u Ä1 =
???????
|
2 1 1 1 1
|
1 1 1 1 1
|
1
1
2
1
1
|
1
1
1
2
1
|
1 1 1 1 1
|
?
,
???????
|
?
? ? ? ? ? ? ? ? ? ?
Ä2 =
|
3 1 1 1 1 1
|
1
2 1 1 1 1
|
1 1 1 1 1 1
|
1
1
1
3
1
1
|
1 1 1 1
3
1
|
1 1 1 1
1
2
|
?
et Ä0 = I120.
??????????
|
Nous avons choisi un dispositif équilibréavec le
facteur 2 (6 niveaux) hiérarchisédans le facteur 1 (5 niveaux).
Les matrices Z1 et Z2 sont donc les
matrices
d'incidence correspondantes a` ces deux facteurs tandis que la
matrice Z0 est l'identitéd'ordre 120.
Nous présentons au Tableau 4.3 les résultats des
simulations obtenues avec le modèle 4.22 sur 300 jeux de
données.
|
Valeurs simulées
|
Valeurs estimées
|
|
0,5402
0,8948
|
Moyenne
0,5380
0,8974
|
'Ecart-type
0,0937
0,0931
|
|
â
|
0,6476
|
0,6423
|
0,0912
|
|
0,9513
|
0,9526
|
0,0921
|
|
0,0772
|
0,0748
|
0,0933
|
|
ó21
|
0,4653
|
0,4241
|
0,4416
|
|
ó2 2
|
0,2458
|
0,2471
|
0,3324
|
|
ó2 0
|
0,9226
|
0,8874
|
0,1233
|
TAB. 4.3 - Résultats des simulations pour un
modèle mixte a` effets aléatoires corrélés
effectuées sur 300 jeux de données.
Au vu de ces résultats, les valeurs estimées pour
les paramètres ne sont pas, en moyenne, éloignées des
valeurs simulées.
Le modèle PLS-Mixte a` effets aléatoires
corrélés
Nous venons ainsi de voir, a` la section pr'ec'edente,
l'estimation des paramètres
fixes et des composantes de variance dans
un modèle lin'eaire mixte, a` l'aide
de l'algorithme EM, o`u chaque
effet al'eatoire est de variance ó2 kÄk
et o`u n > p.
Le cas n < p avec cette même
structure de variance des effets al'eatoires, n'ecessite, comme au chapitre
pr'ec'edent, d'imbriquer la r'egression PLS, en tant que m'ethode de r'eduction
de dimension, a` l'algorithme EM. Nous proposons l'algorithme PLS-Mixte
suivant, fond'e sur la variation de ML propos'ee par Laird
'Etape 0 Mettre m = 0 et
choisir des valeurs de depart
ó2k(0)
'Etape 1 Centrer et reduire X
et Y : x0 = X,
y0 = Y
'Etape 1.1 For h = 1, 2, ... ,
rang(X)
(a) Calculer les p-vector wh =
[w1h · ·
· wph]' o`u
wph = Cov(xph, yh)/ E
Cov2(xph, yh)
et xph la
pe
p
colonne de xh
(b) Normer wh : wh = wh/ II wh
II
(c) Calculer les variables latentes
t(m)
h = xh-1wh
(d) Calculer ch par regression GLS de
yh-1 sur t(m)
h
|
yh-1 =
t(h
m)ch + yh o`u
Var(yh-1) = V(m)
=
|
Er
k=0
|
ZkÄk
Z'kó2k m)
|
ch =(t(h
m)'V-1(m)t(hm))-t(h
m)'V-1(m)yh-1
(e) Calculer ph par regression de
xh-1 sur t(m)
h
xh-1 =
t(h
m)p'h+
xh p'h
=(t(h
m)'t(hm)
)-1t(h
m)'xh-1
(f) Calculer les residus xh and yh
(g)
Er
i=0
Ziui
Finalement Y =
T(m)C +
o`u T(m)
=[t(m)
1 ···t(h
m)] et C =
[c1 · · · ch]'
'Etape 1.2 Calculer
2m+1)
ók( =
ó2k (m)
(ók (m) / qk)
[YP(m) Z k
Ä'kZ'kP(m)Y
- tr(Z'kV -1(m)
Zk k)1
o`u P(m) =
V-1(m)-V-1(m)T(m)(T(m)'V-1(m)T(m))-T(m)'V-1(m)
'Etape 2 Si convergence, prendre
bó2k =
ó2k(m+1) ; sinon ajouter
1 a` m
et retourner a` 'Etape 1.1
Bien evidemment, le changement par rapport a` l'algorithme de
la section precedente concerne l''Etape 1.1 (d) et l''Etape 1.2 o`u
l'estimation des
ó2k tient compte de la
variance ó2kÄk des
effets aleatoires uk.
En resume, les estimations dans le mod`ele PLS-Mixte a` effets
aleatoires
corr'el'es ont ete'ecrites de mani`ere explicite. Neanmoins, pour
l'algorithme
itératif proposéa` cet effet, la convergence n'a
pas étéétablie et est restée locale.
Chapitre 5
Conclusion générale
Nous nous 'etions fix'es comme objectif principal de
mod'eliser les interactions G×E en fonction de
covariables notamment climatiques observ'ees sur le milieu, avec comme
application sa pr'ediction dans une exp'erimentation multienvironnements
sah'elienne.
Nous avons commenc'e par exposer les principales m'ethodes
existantes dans la litt'erature et montrer leurs limites. Ces m'ethodes ne sont
adapt'ees dans le contexte sah'elien que par la prise en compte de la grande
variabilit'e climatique du milieu d'une ann'ee sur l'autre. Or, la plupart de
ces m'ethodes ne considèrent pas justement les caract'eristiques du
milieu pour y pr'edire la performance des g'enotypes. La seule m'ethode a`
notre connaissance qui y tient compte - la r'egression factorielle - ne permet
pas de g'erer les nombreuses variables climatiques ordinairement mesur'ees sur
ces lieux.
Les modèles de simulation de culture ont aussi 'et'e
pr'esent'es comme une m'ethode alternative pour 'evaluer le comportement des
g'enotypes selon les environnements. Cependant, ces modèles tout comme
le modèle SarraH que nous avons utilis'e pour cette 'etude, sont
rarement param'etr'es pour plus d'une vari'et'e. Or, pour pouvoir rendre compte
des diff'erences de production
des g'enotypes, il est essentiel que les paramètres de ces
modèles puissent changer selon les g'enotypes.
Nous proposons en cons'equence la m'ethode APLAT,
Approximation par lin'earisation autour d'un t'emoin. Cette m'ethode consiste
a` lin'eariser la r'eponse pr'edite par un modèle de simulation de
culture de tout g'enotype d'un environnement donn'e autour du vecteur de
paramètres connu d'un g'enotype de r'ef'erence. Après cela,
certains des paramètres du modèle de simulation de culture, qui
peuvent être choisis aux dires d'experts, peuvent être r'eestim'es
a` l'aide de cette lin'earisation locale. L'avantage de cette m'ethode est de
ne pas n'ecessiter pour tout g'enotype, l'exp'erimentation sp'ecifique
indispensable a` la param'etrisation du t'emoin. A l'ensemble de ces
exp'erimentations instrument'ees requises pour toute nouvelle vari'et'e
d'int'erêt, est substitu'e un essai multilocal classique qu'on aura pris
soin de munir de stations m'et'eorologiques simples.
La m'ethode APLAT a 'et'e valid'ee avec les donn'ees
d'arachide d'un essai pluriannuel men'e a` la station exp'erimentale du CERAAS
au S'en'egal de 1994 a` 1998. Pour ces donn'ees, chacune des ann'ees a 'et'e
successivement r'eserv'e et le rendement des g'enotypes pr'edit a` l'aide des
ann'ees restantes, d'abord a` l'aide de la m'ethode APLAT, ensuite a` l'aide du
modèle moyen. Le modèle moyen est utilis'e pour estimer la
performance des g'enotypes pour une ann'ee donn'ee par la moyenne des autres
ann'ees. Un modèle qui pr'edit moins bien en moyenne que le
modèle moyen n'est pas acceptable. Nous avons trouv'e que quatre fois
sur cinq, la m'ethode APLAT s''etait montr'ee meilleure que le modèle
moyen.
Ensuite sur les donn'ees de 11 sites d'un essai multilocal
men'e durant la saison des pluies de 2005 au S'en'egal, la m'ethode APLAT a
fourni une meilleure pr'ediction moyenne du rendement des g'enotypes que le
modèle moyen, 10 fois sur 11.
Par la suite, la m'ethode APLAT a 'et'e 'etendue au cas
d'essais a` plusieurs composantes de variance, qui devient APLAT-Mixte. Nous
avons adjoint au modèle SarraH que nous avions utilis'e pour rendre
compte du comportement diff'erenci'e des g'enotypes dans cette situation de
variabilit'e environnementale accrue, un effet al'eatoire de l'environnement
responsable d'interactions G×E dont il a fallu estimer la
variance. Cette m'ethode s'appuie sur la technique PLS-Mixte que nous avons
propos'e pour l'estimation des composantes de variance dans le cas o`u il y a
plus de r'egresseurs que d'observations. Cette technique PLS-Mixte consiste a`
imbriquer la r'egression PLS dans l'algorithme EM et constitue un algorithme
it'eratif d'estimation des paramètres inconnus dans le cas du
modèle mixte dans un contexte de r'eduction de dimension.
Cette technique PLS-Mixte a 'et'e illustr'ee avec des donn'ees
de NIRS obtenues a` partir d'un dispositif exp'erimental en lattice
9×10 o`u l'effet du bloc et l'effet de la r'ep'etition
ont 'et'e suppos'es al'eatoires. Sur ces donn'ees, nous avons estim'e les
paramètres fixes et les composantes de variance du modèle avec
cette m'ethode PLS-Mixte fond'ee sur ML et sur REML. Nous avons compar'e ce
type d'estimation a` celui de la r'egression PLS qui 'etait habituellement
utilis'ee dans ce genre de situation c'est-à-dire, quand il y avait plus
de r'egresseurs que d'observations. Il a alors 'et'e trouv'e que les MSEP de la
technique PLS-Mixte fond'ee sur ML et sur REML 'etaient plus faibles que le
MSEP de la r'egression PLS faite simplement sans tenir compte des diff'erentes
sources de variation.
Nous avons voulu adapt'e la technique PLS-Mixte au cas
d'effets al'eatoires corr'el'es et h'et'erogènes. Pour cela, il a
d'abord fallu se placer uniquement dans le cadre du modèle mixte avec de
tels effets al'eatoires et surseoir au cas de r'eduction de dimension. Ainsi,
les estimations des composantes de variance pour ces effets al'eatoires et
celles des paramètres ont 'et'e 'ecrits. Sur les simulations effectu'ees
pour tester de la convergence et de la qualit'e des ces estimations, nous avons
retrouv'e en moyenne des valeurs très proches de
ceux des paramètres inconnus simul'es. Cependant,
l'algorithme 'ecrit pour le cas PLS-Mixte avec des effets al'eatoires
corr'el'es et h'et'erogènes s'est heurt'e a` des difficult'es de
convergence.
Il nous parait n'ecessaire d'attirer l'attention sur les
limites de notre travail qui sont multiples. Tout d'abord, la m'ethode APLAT
s'appuie essentiellement sur un modèle de simulation de culture. Et a`
ce titre, l'efficacit'e de cette m'ethode a` bien g'erer le comportement
vari'etal selon les environnements est intrinsèquement li'e a` la
capacit'e du modèle de simulation utilis'e a` bien exprimer les
interactions G×E.
Ensuite, dans ce travail, il est propos'e de r'eestimer les
paramètres des g'enotypes utilis'es dans le cadre d'un modèle de
simulation de culture pour pouvoir mieux pr'edire leurs rendements. Or, de tels
modèles de simulation disposent g'en'eralement d'un ensemble important
de paramètres qui ont 'et'e estim'es seulement pour un t'emoin, au moyen
d'exp'erimentations sp'ecifiques. Pour appr'ecier la diff'erence de
productivit'e des vari'et'es selon les environnements, il serait n'ecessaire de
r'eestimer tous les paramètres pour tout nouveau g'enotype avant
l'utilisation de ces modèles. Ce qui peut être complexe et
fastidieux. Mais comme tous les paramètres n'agissent pas de la
même façon sur la performance des vari'et'es, nous avons propos'e
d'en r'eestimer seulement quelques uns. Pour cela, nous avons effectu'e ce tri
s'electif par dires d'experts pour gagner du temps et aussi parce qu'en
g'en'eral les promoteurs d'un modèle de simulation de culture ont une
assez bonne vue du comportement g'en'eral et individuel des paramètres
vari'etaux. N'eanmoins, cette prise en compte des connaissances a
priori sur les paramètres peut être coupl'ee a` une
analyse de sensibilit'e qui permet d'avoir l'impact quantitatif de ces
paramètres sur la production des g'enotypes. Une autre voie est
d'attacher une loi de probabilit'e aux paramètres, qui permet
d'int'egrer les connaissances a priori des experts sur les
vari'et'es. Cette m'ethode, non loin de celle que nous avons utilis'e dans le
cadre de ce travail, se place dans le cadre bay'esien. Les dires d'experts,
pour chaque paramètre, peuvent ainsi être
consid'er'es comme une r'ealisation d'une variable al'eatoire dont on
connaàýt la loi a priori. Il s'agira par la
suite de prendre comme estimation de chaque paramètre vari'etal
l'esp'erance de la loi a posteriori obtenue avec les
donn'ees.
Enfin, nous avons utilis'e dans le cadre de cette 'etude des
donn'ees d'essais multienvironnements. La courte s'erie de cinq ann'ees de
l'essai pluriannuel et le nombre important d'abandons de g'enotypes au cours de
cet essai n'autorisent pas de conclure directement quant a` l'efficience de la
m'ethode APLAT. A l'essai multilocal, la s'erie 'etait plus longue, 11 lieux,
mais ce qui gêne en r'ealit'e au Sahel, c'est moins la variabilit'e
climatique entre les lieux que celle d'une ann'ee sur l'autre pour un
même lieu. Pour cela, ce qu'il aurait fallu faire si le temps l'avait
permis, c''etait de r'ep'eter l'essai multilocal sur deux a` cinq ans tout en
diminuant le nombre de lieux. Ce qui est pr'ef'erable a` mener le même
nombre d'essais sur plusieurs lieux en une seule ann'ee (Talbot, 1997).
En somme, nous avons propos'e de pr'edire les interactions
G×E par lin'earisation d'un modèle de simulation
de culture. Dès lors qu'il est possible, pour tout g'enotype, de
r'eestimer les paramètres n'ecessaires a` un modèle de simulation
de culture, cette m'ethode permet la comparaison de vari'et'es dans des
environnements o`u elles n'ont pas 'et'e observ'ees. Bien entendu, pour cela
faudrait-il disposer de longues s'eries de donn'ees climatiques pour les lieux.
Ces donn'ees climatiques pourront servir alors comme entr'ees d'un
modèle de simulation des pluies journalières a` l'exemple du
modèle stochastique des chroniques pluviom'etriques d'evelopp'e par
Goz'e (1990).
Permettant de mesurer l'impact du changement climatique, cet
outil est destin'e en premier lieu aux s'electionneurs du Sahel qui pourront
dans des programmes de s'election de courte dur'ee (deux a` cinq ans) r'eduire
l'incertitude de la pr'ediction des performances des nouvelles vari'et'es.
Adapt'e en outre a` l''echelle de la r'egion et coupl'e a` une m'ethode
d'estimation des surfaces
cultivées, il peut être utilisédans le
cadre de la prévention des crises alimentaires en Afrique de lOuest, et
particulièrement au Sahel. Ainsi, pour chaque année, le
rapprochement entre les productions et les besoins prévisionnels peut
alimenter le dispositif de veille déjàmis en place par le CILSS
avec l'ensemble de ses partenaires techniques dans le cadre de l'alerte
précoce, afin de faciliter la prise de décision dans
l'élaboration des stratégies alimentaires au Sahel.
R'ef'erences cit'ees
[1] AASTVEIT A.H. et MARTENS H. ANOVA interactions interpreted
by partial least squares regression. Biometrics, 1986, 42, 829-844.
[2] AFFHOLDER F. Empirically modelling the interaction
between intensification and climatic risk in semi arid regions. Field Crops
Research; 1997, 52, 79-93.
[3] AJI S., TAVOLARO S., LANTZ F., FARAJ A. Apport du
bootstrap a` la r'egression PLS : application a` la pr'ediction de la qualit'e
des gazoles, Oil & Gas Science andTechnology-Rev. IFP, 2003, 58,
599-608.
[4] ANNEROSE D. et DIAGNE M. Caract'erisation de la
s'echeresse agronomique en zone semi-aride. Pr'esentation d'un modèle
simple d''evaluation appliqu'e au cas de l'arachide cultiv'ee au S'en'egal.
Ol'eagineux, 1990, 45, 12, 547-554.
[5] ANNEROSE D. et DIAGNE M. Les modèles de cultures :
des outils de la recherche et du d'eveloppement agricole. Arachide Infos, 1994,
5, 5-11.
[6] ANNICCHIARICO, P. Genotype x environment interaction -
challenge and opportunities for plant breeding and cultivar recommendations.
FAO, Rome, 150 p.
[7] BAKER R.J. Tests for crossover genotype-environmental
interactions. Canadian Journal of Plant Science, 1988, 68, 405-410.
[8] BARIL, C.P. Etude de la stabilit'e du rendement chez le bl'e
tendre d'hiver.- 170 p. Th : Agronomie : Paris-Sud centre d'Orsay : 1992.
[9] BARON C. Modèle de bilan hydrique et de croissance
des plantes cereals: Mil, Sorgho et Arachide. CIRAD, 2002.
[10] BATTERBURY S., WARREN A. The African Sahel 25 years
after the great drought : assessing progress and moving towards new agendas and
approaches. Global Environmental Change, 2001, 11, 1-8.
[11] BECKER H.C. Correlations among some statistical measures of
phenotypic stability. Euphytica, 1981, 30, 835-840.
[12] BECKER H.C. et L'EON J. Stability analysis in plant
breeding. Plant Breeding, 1988, 101, 1-23.
[13] BELHASSEN E., THIS D. et MONNEVEUX P. L'adaptation
génétique face aux contraintes de sécheresse. Cahiers
agricultures, 1995, 4, 251-261.
[14] BRADU D. et GABRIEL K.R. The biplot as a diagnostic tool
for models of two-way tables. Technometrics, 1978, 20, 47-68.
[15] BRANCOURT-HULMEL M., BIARNES-DUMOULIN V. et DENIS J.B.
Points de repères dans l'analyse de la stabilitéet de
l'interaction génotype-milieu en amélioration des plantes.
Agronomie, 1997, 17, 219- 246.
[16] CALINSKI T., CZAJKA S. et KACZMAREK, Z. A model for the
analysis of a series of experiments repeated at several places over a period of
years. I. Theory. Biuletyn Oceny Odmian, 1987, 17/18, 7-33.
[17] CILLS. CSSA, Cadre stratégique de
sécuritéalimentaire durable dans une perspective de lutte contre
la pauvretéau sahel. CILSS, Ougadougou, 2000, 88 p.
[18] COLSON J., WALLACH D., BOUNIOLS A., DENIS J., JONES J.
Mean Squared Error of Yield Prediction by SOYGRO. Agronomy Journal, 1995, 87 ,
397-407.
[19] CORNELIUS P.L. Functionc approximating Mandel's tables
for the means and standard deviations of the first three roots of a Wishart
matrix. Technometrics, 1980, 22, 613-616.
[20] CORNELIUS P.L. Statistical tests and retention of terms
in the additive main effects and multiplicative interaction model for cultivar
trials. Crop Science, 1993, 33, 1186-1193.
[21] CORNELIUS P.L., SEYEDSADR M.S. et CROSSA J. Using the
shifted multiplicative model to search for »separability» in crop
cultivar trials. Theoretical and Applied Genetics, 1992, 84, 161-172.
[22] CROSSA J. Statistical analyses of multilocation trials.
Advances in agronomy, 1990, 44, 55-85.
[23] CROSSA J., CORNELIUS P.L., SAYRE K. et ORTIZ-MONASTERIO
R.J. A shifted multiplicative model fusion method for grouping environments
without cultivar rank change. Crop Science, 1995, 35, 54-62.
[24] CROSSA J., CORNELIUS P.L., SEYEDSADR M.S. et BYRNE, P. A
shifted multiplicative model cluster analysis for grouping environments without
genotypic rank range. Theoretical and Applied Genetics, 1993, 85, 577-586.
[25] de JONG S. SIMPLS : An alternative approach to partial
least squares regression. Chemometrics and Intelligent Laboratory Systems,
1993, 18, 251-263.
[26] DEMPSTER A.P., LAIRD N.M. et RUBIN, D.B. Maximum
Likelihood from Incomplete Data via the EM Algorithm. Journal of the Royal
Statistical Society B, 1977, 39, 1-38.
[27] . DENIS J.B. et VINCOURT P. Panorama des méthodes
statistiques d'analyse des intéractions génotype x milieu.
Agronomie, 1982, 2, 3, 219- 230
[28] DENIS J.B. L'analyse de régression factorielle.
Biométrie Praximétrie, 1980, 10, 1-34.
[29] DENIS J.B. Two-way analysis using covariates. Statistics,
1988, 19, 123- 132.
[30] DENIS J.B., PIEPHO H.P. et van EEUWIJK F.A. Modelling
expectation and variance for genotype by environment data. Heredity, 1997, 79,
162-171.
[31] DIENG I. Pr'ediction de l'interaction g'enotype x
environnement a` partir d'indices vari'etaux de sensibilit'e a` la s'echeresse
et de bilan hydrique a` l'aide d'un modèle de r'egression factorielle
dans des essais d'arachide au S'en'egal.- 60 p. M'emoire de DEA :
Biostatistique : Montpellier II : 2003.
[32] DIENG I.; GOZE E., SABATIER R. Lin'earisation autour
d'un t'emoin pour pr'edire la r'eponse des cultures. Comptes Rendus de
l'Acad'emie des Sciences Biologies, 2006, 329, 148-155.
[33] DOORENBOS J. et JASSAM A. H. Yield response to water. In
Irrigation and drainage Paper 33. FAO, Rome, 1979, pp 193.
[34] EAGLEMAN J.R. An experimentally derived model for actual
evapotranspiration. Agricultural meteorology, 1971, 8, 4-3, 385-94.
[35] EBDON J.S. et GAUCH, H.G. Jr. Additive Main Effect and
Multiplicative Interaction Analysis of National Turfgrass Performance Trials :
I. Interpretation of Genotype x Environment Interaction. Crop Science, 2002a,
42, 489-496.
[36] EBDON J.S. et GAUCH H.G. Jr. Additive Main Effect and
Multiplicative Interaction Analysis of National Turfgrass Performance Trials :
II. Cultivar Recommendations. Crop Science, 2002b, 42, 497-506.
[37] EBERHART S.A. et RUSSELL W.A. Stability parameters for
comparing varieties. Crop Science, 1966, 6, 36-40.
[38] EFRON B. Bootstrap Methods : Another Look at the Jacknife.
Annals of statistics, 1979, 7, 1-26.
[39] FAO. IRSIS, Irrigation scheduling information system. FAO,
Rome, 1987.
[40] FINLAY K.W. et WILKINSON G.N. The analysis of adaptation
in a plant-breeding programme. Australian Journal of Agricultural Research,
1963, 14, 742-754.
[41] FOREST F. et CLOPES A., 1991. Contribution a`
l'explication de la variabilitédes rendements d'une culture de
ma·ýs plus ou moins intensifiée a` l'aide d'un
modèle de bila hydrique amélioré. In Bilan hydrique
agricole et sécheresse en Afrique tropicale. France : John Libbey
Eurotext, 1991.- pp 3-15.
[42] FOREST F. et CORTIER B. Le diagnostic hydrique des
cultures et la prévision du rendement régional du mil
cultivédans les pays du CILSS. IRAT/CIRAD/AGRHYMET. IAHS, 1990,
544-557.
[43] FRANQUIN P. et FOREST F. Des programmes pour
l'évaluation et l'analyse frequentielle des termes du bilan hydrique.
Agronomie Tropicale, Janvier-Mars 1977, XXXII-1, 7-11.
[44] FREEMAN G. H. et PERKINS J. M. Environmental and
genotype-environmental components of variability. VIII. Relations between
genotypes grown in different environments and measures of these environments.
Heredity, 1971, 27, 15-23.
[45] GAUCH H.G. et ZOBEL R.W. 1996. AMMI analysis of yield
trials. In Genotype-by-environment interaction. Boca Raton, Floride : M.S. Kang
& H.G. Gauch, 1996.- pp 85-122.
[46] GAUCH H.G. et ZOBEL R.W. Predictive and postdictive
success of statistical analyses of yield trials. Theoretical and Applied
Genetics, 1988, 76, 1-10.
[47] GAUCH H.G. Jr. Statistical Analysis of Yield Trials by AMMI
and GGE. Crop Science, 2006, 46 :1488-1500.
[48] GAUCH H.G. Statistical analysis of regional yield trials :
AMMI analysis of factorial designs. Amsterdam : Elsevier, 1992.
[49] GAUCH, H.G. Full and reduced models for yield trials.
Theoretical and Applied Genetics, 1990, 80, 153-160.
[50] GOLLOB H.F. A statistical model which combines features
of factor analytic and analysis of variance techniques. Psychometrika, 1968,
33, 73-115.
[51] GOZE E. Modèle stochastique de la
pluviométrie au Sahel - Application a` l'agronomie- Th :
UniversitéMontpellier II : 1990.
[52] KEMPTON R.A. The use of biplots in interpreting variety by
environment interactions. Journal of Agricultural Science, 1984, 103,
123-135.
[53] LAIRD N. M. Computation of variance components using the
EM algorithm. Journal of Statistical Computation and Simulation, 1982, 14,
295-303.
[54] LIN C.S., BINNS M.R. et LEFKOVITCH L.P. Stability Analysis
: Where Do We Stand? Crop Science, 1986, 26, 894-900.
[55] MANDEL J. A new analysis of variance model for non-additive
data. Technometrics, 1971, 13, 1-18.
[56] MANDEL J. Non-additivity in two-way analysis of variance.
Journal of American Statistical Association, 1961, 56, 878-888.
[57] MCCULLOCH C. E. et SEARLE S. R. Generalized, Linear, and
Mixed Models. New York: John Wiley & Sons, 2001.
[58] MCLACHLAN G.J. et KRISHNAN T. The EM Algorithm and
Extensions. New Jersey : Wiley, 1997.
[59] MENG X.L. et van DYK D.A. Fast EM-type implementations
for mixed effects models. Journal of the Royal Statistical Society B, 1998, 60,
559- 578.
[60] MORENO-GONZALEZ J., CROSSA J. et CORNELIUS P.L. Additive
Main Effects and Multiplicative Interaction Model : I. Theory on Variance
Components for Predicting Cell Means. Crop Science, 2003a, 43, 1967- 1975.
[61] MORENO-GONZALEZ J., CROSSA J. et CORNELIUS P.L. Additive
Main Effects and Multiplicative Interaction Model: II. Theory on Shrinkage
Factors for Predicting Cell Means. Crop Science, 2003b, 43 :1976- 1982
[62] PERKINS J.M. et JINKS J.L. Environmental and
genotype-environmental components of variability. III. Multiple lines and
crosses. Heredity, 1968, 23, 339-356.
[63] PIEPHO H.P. Best Linear Unbiased Prediction (BLUP) for
regional yield trials : a comparison to additive main effects and
multiplicative interaction (AMMI) analysis. Theoretical and Applied Genetics,
1989, 89, 647-654.
[64] PIEPHO H.P. Methods for Comparing the Yield Stability of
cropping Systems - A Review. Journal of Agronomy and Crop Science, 1998, 180,
193-213.
[65] PIEPHO H.P., Denis J.B. et van Eeuwijk F.A. Predicting
cultivar differences using covariates. Journal of Agricultural, Biological, and
Environmental Statistics, 1998, 3, 151-162.
[66] PIEPHO, H.P. Robustness of statistical tests for
multiplicative terms in the additive main effects and multiplicative
interaction model for cultivar trials. Theoretical and Applied Genetics, 1995,
90, 438-443.
[67] RAMI J. F., DUFOUR P., TROUCHE G., FLIEDEL G., MESTRES
C., DAVRIEUX F., BLANCHARD P., et HAMON P. Quantitative trait loci for grain
quality, productivity, morphological and agronomical traits in Sorghum
(Sorghum bicolor L. Moench). Theorical Applied genetics, 1998,
97, 605-616.
[68] RAO C. R. et KLEFFE J. Estimation of variance components
and applications. Amsterdam : North Holland series in statistics and
probability, Elsevier, 1988.
[69] SEARLE S. R. Linear models for unbalanced data. New York :
John Wiley & Sons, 1987.
[70] SEARLE S. R., CASELLA G. et MCCULLOCH C. E. Variance
Components. New York : John Wiley & Sons, 1992.
[71] SEIF E., EVANS J.C. et BALAAM L.N. A multivariate
procedure for classifying environments according to their interaction with
genotypes. Australian Journal of Agricultural Research, 1979, 30, 1021-1026.
[72] STONE, M. Cross-validatory choice and assessment of
statistical predictions. Journal of the Royal Statistical Society B, 1974, 36,
111-147.
[73] TALBOT M. et WHEELWRIGHT A.V. The analysis of genotype x
environment interactions by partial least squares regression. Biuletyn Oceny
Odmian, 1989, 21/22, 19-25.
[74] . TALBOT M. Resource allocation for selection systems.
In : Statistical methods for plant variety evaluation. London : KEMPTON RA, FOX
PN, Chapman et Hall, 1997.- pp 162-174.
[75] TENENHAUS M. La Régression PLS : théorie et
pratique. Parsi : Tech-nip, 1998.- 254 p.
[76] . THEOBALD C.M., TALBOT M. et NABUGOOMU F. A Bayesian
approach to regional and local-area prediction from crop variety trials.
Journal of Agricultural, Biological, and Environmental Statistics, 2002, 7,
12-28.
[77] TUCKER C.J., DREGNE H.E., NEWCOMB W.W. Expansion and
contraction of the Sahara Desert from 1980 to 1990. Science, 1991, 253,
299-301.
[78] TUKEY J.W. One degree of freedom for non additivity.
Biometrics, 1949, 5, 232-242.
[79] TURNER N. C., WRIGHT G. C. et SIDDIQUE K. H. M.
Adaptation of grain legumes (pulses) to water-limited environments. Advances in
agronomy, 2002, 71, 193-231.
[80] van EEUWIJK F.A. Interpreting genotype-by-environment
interaction using redundancy analysis. Theoretical and Applied Genetics, 1992,
85, 89-100.
[81] van EEUWIJK F.A. Linear and bilinear models for the
analysis of multi-environment trials. I. An inventory of models. Euphytica,
1995, 84, 1-7.
[82] van EEUWIJK F.A., DENIS J.B. et KANG M.S. 1996.
Incorporating additional information on genotypes and environments in models
for two-way genotype by environment tables. In Genotype-by-environment
interaction. Boca Raton, Floride : M.S. Kang & H.G. Gauch, 1996.- pp
15-49.
[83] VARGAS M., CROSSA J., SAYRE K., REYNOLDS M., RAM`IREZ
M.E. et TALBOT M. Interpreting genotype x environment interaction in wheat by
partial least squares regression. Crop Science, 1998, 38, 679-689.
[84] VARGAS M., CROSSA J., van EEUWIJK F.A., RAM`IREZ M.E. et
SAYRE K. Using partial least squares regression, factorial regression, and AMMI
models for interpreting genotype x environment interaction. Crop Science, 1999,
39, 955-967.
[85] WALLACH D., GOFFINET B. Mean Squared Error of Prediction
in Models for Studying Ecological and Agronomic Systems. Biometrics, 1987, 43,
561-573.
[86] WILLIAMS E.J. The interpretation of interactions in
factorial experiments. Biometrika, 1952, 39, 65-81.
[87] WOLD H. Estimation of principal components and related
models by iterative least squares. In Multivariate Analysis. New York :
Krishnaiah P.R., Academic Press, 1966, pp 391-420.
[88] WOLD H. Modelling in Complex Situations with Soft
Information. Third World Congress of Econometric Society. Toronto, Canada, 1975
August 21-26.
[89] WOLD S., ALBANO C., DUNN W.J., ESBENSEN K., HELLBERG S.,
JOHANSSON E., SJOSTROM M. Pattern recognition: Finding and
using patterns in Multivariate Data. In : Food Research and
Data Analy-
sis. London : Applied Science: Martens H, Russwurm H Jr, 1983
;147-188.
[90] WOOD J. T. The use of environmental variables in the
interpretation of genotype-environment interaction. Heredity, 1976, 37, 1-7.
[91] WU C. F. On the convergence properties of the EM algorithm.
The Annals of Statistics, 1983, 11, 95-103.
[92] YAN W. et HUNT W.A. Interpretation of Genotype x
Environment Interaction for Winter Wheat Yield in Ontario. Crop Science, 2001,
41, 19-25.
[93] YATES F. et COCHRAN W.G. The analysis of groups of
experiments. Journal of Agricultural Science, 1938, 28, 556-580.
[94] ZOBEL R.W., WRIGHT M.J. et GAUCH H.G. Statistical analysis
of a yield trial. Agronomy Journal, 1988, 80, 388-393.
Annexe A : Mod`ele de
R'egression factorielle
On dispose de trois matrices : X de
dimension I x p est la matrice des co-variables associees au
premier facteur (facteur varietepar exemple), Z de
dimension J x q est la matrice des covariables associees au
deuxieme facteur (facteur lieu) et Y de dimension I x
J est la matrice des observations. Les matrices
X, Y et Z s'ecrivent ainsi :
|
x11
?
x21
X=
? ? ? ? ? ...
xI1
|
x12
x22
...
xI2
|
'' '
' ' '
'' '
|
x1p
x2p
...
xIp )
|
z11
?
z21
Z=
? ? ? ? ? ...
zJ1
|
z12
z22
...
zJ2
|
' ''
' ' '
' ''
|
z1q
z2q
...
zJq )
|
|
y11
?
y21
Y=
? ? ? ? ? ...
yI1
|
y12
y22
...
yI2
|
' '
' '
' '
|
'
'
'
|
y1J
y2J
...
yIJ )
|
L'idee est d'utiliser les deux matrices de covariables
X et Z attachees aux deux facteurs varieteet
environnement pour expliquer la matrice Y.
Pour cela, une premiere regression de Y
sur X est faite : Y =
Xa bYX =
X(X/X)-1X'Y
et RX = Y -
avec II repr'esentant la matrice identit'e
d'ordre I et RX la matrice des r'esidus de la
r'egression de Y sur X qui est de
dimension I × J.
Une deuxième r'egression de àY'
X sur Z est faite.
àY'X
= Zâ àY'XZ =
Z(Z'Z)-1Z'
àY'X
Ainsi, àYXZ =
àYXZ(Z'Z)-1Z'
=
X(X'X)-1X'YZ(Z'Z)-1Z'
D'o`u
RàYx =
àYX - àYXZ =
X(X'X)-1X'Y
-
X(X'X)-1X'YZ'(ZZ')-1Z
RàYx =
X(X'X)-1X'Y(IJ
-
Z'(ZZ')-1Z)
Enfin, une dernière r'egression de RX
sur Z est faite.
R'X
= Zy àR'XZ =
Z(Z'Z)-1Z'R'X
àRXZ =
RXZ(Z'Z)-1Z'
d'o`u àRXZ = (IJ -
X(X'X)-1X')YZ(Z'Z)-1Z'
Ainsi, Y = àYXZ +
RàYx +
àRXZ + R'esidus,
Posons
àYXZ =
X(X'X)-1X'YZ(Z'Z)-1Z'
= XMZ' (B.1)
RàYx =
X(X'X)-1X'Y(IJ
-
Z(Z'Z)-1Z')
= Xâ' (B.2)
àYXZ = (IJ -
X(X'X)-1X')YZ(Z'Z)-1Z'
= áZ' (B.3)
X et Z peuvent àetre mis
sous la forme X = ( 1I Xÿ ) Z = ( 1J Zÿ
)
1' 1'
avec ./ ) et Z' = ÿI
) vec Xÿ se d'eduisant de la matrice
X par
X' Z'
centrage des colonnes (de màeme pour
ÿZ)
! !
1' I 1' I
Xÿ
ÿX'X
= I ( 1I Xÿ ) =
ÿX'
ÿX'1I ÿX'
Xÿ
! !
IJ 1'
1 1
IY1J I 1' IY
ÿZ( ÿZ'
ÿZ)-1 m m'
2
=
J ( ÿX'
ÿX)-1
ÿX'Y1J (
ÿX'
ÿX)-1
ÿX'Y
ÿZ( ÿZ'
ÿZ)-1
1 m1 Mÿ
M=
!-1
A B = A-1 +
FE-1F'
--FE-1
Or
B' D
--E-1F' E-1
avec E = D --
B'A-1B
et F = A-1B
Ici A = I , B = 1'I
Xÿ = B' =
ÿX'1I et D =
ÿX' Xÿ D'o`u
A-1 = 1I , F = I
1 1' I Xÿ = 0 car
Xÿ centre
et E = ÿX' Xÿ =
E-1 =
(ÿX' Xÿ) 1.
!-1 1
ÿX'1I ÿX'
Xÿ
0
( ÿX'
ÿX)
-1
De màeme (
ÿZ'Z)-1
=
0 ( ÿZ'
ÿZ)-1
1 J 0
D'après l'equation (B.1) M =
(X'X)-1X'YZ(Z'Z)-1
I 1' I Xÿ 0
Ainsi (
ÿX'X)-1
= I
=
M=
M=
M= D'o`u
! ! 1 !
1 0 1' 0
I I Y ( 1J Zÿ ) J
0 ( ÿX'
ÿX)-1
ÿX' 0 (
ÿZ'
ÿZ)-1
! 1 !
I 1'
1 IY 0
( 1J Zÿ ) J
( ÿX'
ÿX)-1
ÿX'Y 0 (
ÿZ'
ÿZ)-1
I 1'IY1
J I 11'IY Zÿ
)
1 0
J
( ÿX'
ÿX)-1
ÿX'Y1J (
ÿX'
ÿX)-1
ÿX'Y Zÿ 0 (
ÿZ'
ÿZ)-1
D'après l'equation (B.2) â' =
(X'X)-1X'Y(IJ
--
Z(Z'Z)-1Z')
!
I 1'
( ÿX'
ÿX)-1
ÿX'Y =
1 IY
( ÿX'
ÿX)-1
ÿX'Y
1!
0
Z(Z'Z)-1
= ( 1J Zÿ ) J = ( 1 J 1J ÿZ(
ÿZ'
ÿZ)-1 )
0 ( ÿZ'
ÿZ)-1
!
1'
Donc
Z(Z'Z)-1Z'
= ( 1 J 1J ÿZ( ÿZ'
ÿZ)-1 ) J = 1
J 1J1' J + ÿZ(
ÿZ' ÿZ)-1
ÿZ'
ÿZ'
!I11'IY (IJ- 1
J1J1' J-
ÿZ(ÿZ'ÿZ)-1ÿZ')
D'o`u â' =
( ÿX'
ÿX)-1
ÿX'Y
! !
I 1'
1 IY(IJ-
1J1J1'J -
ÿZ(ÿZ'
ÿZ)-1ÿZ') = b'
( ÿX'
ÿX)-1
ÿX'Y(IJ-
1J1J1'J -
ÿZ(ÿZ'ÿZ)-1ÿZ')
B'
â' =
D'après l'équation (B.3) á = (II -
X(X'X)-1X')YZ(Z'Z)-1
X(X'X)-1X'
= ( 1I Xÿ )
!
I 1' 1
I= I 1 1I1'
I + ÿX(
ÿX' ÿX)-1
ÿX' (
ÿX' ÿX)-1
ÿX'
D'o`u a = (II -
1I1I1'I -
ÿX(ÿX'ÿX)-1ÿX')(
1J Y1J
YÿZ(ÿZ'ÿZ)-1
)
a = ( (II -
I11I1'I
-
ÿX(ÿX'ÿX)-1ÿX')
1JY1J (II -
1I1I1'I -
ÿX(ÿX'ÿX)-1
ÿX')YÿZ(ÿZ'
ÿZ)-1 )
= ( a A
)
Ainsi Y = àYxZ +
RàYX + àRxZ + R'esidus =
XMZ' + Xâ' +
áZ' + R'esidus
|
! !
mD'o`uY= ( 1IX) m'
2 J + ( 1I Xÿ )
m1 Mÿ Zÿ
B'
|
!
1' J
+ ( a A ) Zÿ
|
!
1'
Yà = ( 1Im + ÿXm1
1Im' 2 + Xÿ Mÿ ) J +
1Ib' +
ÿXB' + a1'
J + A ÿZ'
ÿZ'
D'o`u
Yà =
1Im1'J +
ÿXm11'
J+ 1Im' 2 ÿZ' +
Xÿ Mÿ ÿZ' + 1Ib'
+ ÿXB' +
a1'J + A
ÿZ'
Annexe B : Article au C.R.
Biologie
|
|
|
|
C. R. Biologies 329 (2006) 148-155
|
http://france.elsevier.com/direct/CRASS3/
Biomodélisation / Biological modelling
Linéarisation autour d'un témoin pour
prédire la réponse de cultures
Ibnou Dieng a,*, Éric Gozé
b, Robert Sabatierc
a Centre d'étude régional pour
l'amélioration de l'adaptation à la sécheresse, BP 3320,
Thiès-Escale, Thiès, Sénégal
b Centre
de coopération internationale en recherche agronomique pour le
développement, TA 70/09, avenue d'Agropolis,
34398 Montpellier cedex
5, France
c Laboratoire de physique moléculaire et
structurale, faculté de pharmacie, 15, avenue Charles-Flahault, 34060
Montpellier, France
Reçu le 18 avril 2005; accepté le 17 janvier
2006
Disponible sur Internet le 9 février
2006
Présenté par Michel Thellier
Résumé
Une nouvelle méthode pour modéliser les
interactions génotype x environnement : APLAT. Le rendement de
génotypes prédit par un modèle de simulation de cultures
est développé en série de Taylor à l'ordre 1 au
voisinage du vecteur de paramètres d'un génotype de
référence. À l'aide de cette linéarisation locale,
l'estimation des paramètres de ces génotypes se fait par
régression linéaire des rendements observés sur la
sensibilité des sorties du modèle de simulation de cultures par
rapport aux paramètres. Pour citer cet article :I. Dieng et
al., C. R. Biologies 329 (2006).
(c) 2006 Académie des sciences. Publié par Elsevier
SAS. Tous droits réservés.
Abstract
Prediction of crop response by linearisation about
control approximation. A new method for modelling genotype x
environment interaction: APLAT. The yield predicted by a crop-simulation model
is developed as a Taylor series in the neighbourhood of a parameter vector of a
control genotype. With this local linearisation, these genotype parameters can
be estimated by a linear regression of the observed yield on the derivatives of
the crop-simulation model predictions with respect to its parameters.
To cite this article: I. Dieng et al., C. R. Biologies 329
(2006).
(c) 2006 Académie des sciences. Publié par Elsevier
SAS. Tous droits réservés.
Mots-clés: Linéarisation;
Prédiction de la réponse de cultures ; Témoin; Interaction
genotype x environnement Keywords: Linearization; Predict responses
culture; Control; Genotype x environment interaction
Abridged English version
In Sahel, genotype x environment interactions are often large:
this is the justification behind multilocation
* Auteur correspondant.
Adresses
e-mail:
ibnou.dieng@ceraas.org,
dieng_ibnou@yahoo.fr (I.
Dieng),
eric.goze@cirad.fr (É.
Gozé),
sabatier@univ-montp1.fr (R.
Sabatier).
and pluriannual trials. Because of these sizeable environment
effects and interactions, the prediction of an expected yield with a linear
mixed model is generally imprecise.
Improving this prediction can be achieved by modelling the
environment effect. It is then partly shifted from the random part to the fixed
part of a mixed model, by the use of a crop-simulation model like DHC, IRSIS,
SarraH... This could not be possible with the empirical
genotype × environment interactions analysis methods like
AMMI and joint regression, which do not make use of environmental variables.
The factorial regression method does make use of environmental variables;
however, it requires their effect on the production to be linear, which might
not be the case.
Unfortunately, most crop-simulation models bear a number of
parameters, the estimation of which requires a specific and costly experiment.
As a consequence, these parameters are usually known, but for a small set of
reference genotypes. It would not be sensible to invest in a parameter
estimation experiment for every new genotype that is proposed for selection.
To overcome this problem, one can notice that multisite
experiments usually share a control variety for which parameters have already
been estimated. In this paper, we propose to develop as a Taylor series the
modelled response about the parameters of this control genotype. The other
genotypes' parameters can then be estimated by a linear regression of the
observed yields on the sensitivity to parameters, i.e., on the derivatives of
the response with respect to the parameters. With this estimation, one can
predict the new genotype responses in environments where they have not been
tested. In a given location, this estimation can benefit from the available
historic climatic records to estimate a distribution of probable yields.
Let f
(Zj,èi) denote the yield
of a genotype i predicted by a crop simulation in an environment
j and Yij the observed yield. We can write:
Yij = f (Zj
,èi) + îj + uij
where Zj is the vector of the
jth environment regressors and
èi the P -vector of the ith
genotype parameters. The bias îj is that of the crop-simulation
model. We suppose that it depends only on environment and is the same for all
genotypes in a same environment. The error term uij is supposed random
with expectation 0 and variance
ó2u.
Let us consider a control genotype, i.e., whose parameters are
known or at least already estimated. Let è0 be
the vector of parameters of this control genotype and let us suppose that
f is a C1 class function in a neighbourhood
of è0 and f' derivable in this
neighbourhood. Moreover, let us suppose
èi in the neighbourhood of
è0. Then, a Taylor series expansion yields:
f (Zj,èi)
= f(Zj,è0)
+ P_1 aè(p)
è=è0,Z=Zj
EP [ ?f
× (èi (p) -
è0p))+
?[(èi -
è0)'(è
- è0)]
with èi (p) the pth component of the
parameters vector
of the ith genotype, è(p)
0 that of the control genotype.
(p) ?
Let Xj = [ ? fo(p)
|è=è0,Z=Zj
] and âi (p) = èi (p) -
è0(p)
for p = 1, . . . , P. As f is not
known in closed form, one
has to estimate its derivatives by numerical
approxima-
tion. The function X(p) jis a
function of environment j,
while â(p)
i is a function of genotype i. Then, the
local
linearization yields:
Yij - Y0j = ~P
p=1
X(p)
j ·âi (p) +
~ij
where Y0j is the control response
in the environment j and eij = uij
- u0j. So, E(eij) = 0,
V(eij) = 2ó2u,
Cov(eij , ~i~j~) = 0, but
Cov(eij, ei,j) =
ó2u.
This equation can be put in the form of a linear model with
correlated errors:
Y - (Y0 ?
1I) = X ·
â + e
In this equation, Y is the vector of
responses of the I genotypes in the J environments,
Y'0 = (Y01 · · ·
Y0J), 1I is unit vector of size
I × 1. The symbol ? indicates the Kronecker product and
E is a random error vector. Its covariance matrix is
ó2u?
where:
|
?
? ? ? ? ? ?
|
ù1
|
...
|
|
? =
|
0
|
|
and
?
?
0 ? ?
ùj ? ?
..?
.
ùJ
1
2
?
?
ùj = (21
. . .
The number of columns of the square matrices
? and ùj are
respectively the number of observations for all the environments and the number
of observations for environment j.
Also, X = [
X(1) ?
II · · ·
X(P) ? II ]
where X(p)' = [X(p)
1 ··· X(p)
J ] is a J × 1 vector and
II is the I × I unit matrix.
The dimension of X is then IJ × PI.
inally, )=
[â(1)' ·
· · â("]
where â(p), = [
â Pit
1
We call this method APLAT for Approximation Par
Linéarisation Autour d'un Témoin.
Because of the large number of columns of X,
some dimension reduction method like Partial Least Squares regression is
necessary. The dimension of the space spanned by the regressors is then reduced
from rank of X to k. The PLS regression is usually
carried out
with the NIPALS (Nonlinear estimation by Iterative Partial
Least Squares) algorithm, where the calculation of the components is performed
simultaneously with a set of regressions by ordinary least squares. Here, the
error covariance matrix is
ó2u?,
not ó2uIIJ,
generalized least squares should be used instead. As
? is symmetric and positive semi-definite, a work around consists
in factorizing its inverse, finding a matrix ç
such that ç~ç
= ?-1.
Then, estimating â by PLS with
regressions by generalized least squares is equivalent to consider the
model:
çY -
ç(Y0 ?
1I) = çX ·
â + çE
where âPLS is the
estimation with regressions made by ordinary least squares.
The number of components is chosen to minimize
the PRESS
(Prediction Error Sum of Squares) criterion.
To calculate the confidence
interval of the coeffi-
cients, we used a bootstrap technique. Let z(p)~b
i,PLS be the
random variable defined by:
(p)*b
zi, PLS =
â(p)~b
i,PLS -â(p)
i,PLS
s*(
â(p)~b i,PLS)
where
â(p)
i,PLS is the (p · i)th element
of
âPLS,â(p)~b
i,PLS
is obtained at the bth draw with b = 1, .
. . , B and
s( àâ(p)~b
i,PLS) is the standard error of
â~b PLS. Let ~FB be
the empirical distribution function of z(p)~b
i,PLS. The frac-
tile FB 1(á) is estimated by
àt(á) such that #{z(p)~b
i,PLS
~ àt(á)} = áB.
A percentile-t confidence interval for the (p
· i)th element of â is in the
following form:
~ â(p)
i,PLS -sOilitS ) ·
àt(1 - á), sOilltS) ·
àt(á)]
To evaluate the quality of the new model, we compared its MSEP
(Mean Squared Error of Prediction) with that of the average model defined for
our data as follows:
Yij = m + gi + Ej +
äij
where m is the population mean and gi the
genotype effect. The term Ej is the year effect and it is assumed
random with expectation 0 and variance
ó2E. Errors äij are
distributed independently with expectation 0 and variance
ó2ä . The terms Ej
and äij are assumed to be mutually independent.
The data set consists of plant yields of 26 groundnut
genotypes. The experiments have been carried out at Bambey (14?42N
and 16?28W) in Senegal, over a period of five years from 1994 to
1998. The data of each year were kept in turn as a test sample. Yields are
expressed in kilograms of pods per hectare.
We used SarraH, a crop simulation model developed by CIRAD in
collaboration with CERAAS, to calculate X. Taking into account
the available number of data, we estimated two of its varietal parameters.
The PRESS is minimal with six components for models adjusted
without the data of 1994, 1995 and 1997. For each of the others, the PRESS is
minimal with nine components. However, we decided to keep only five components,
as the PRESS was not very different from its minimum value.
The APLAT MSEPs are lower than the average model MSEP, except
for prediction of 1998 data. Then the prediction of yield for these models by
APLAT was better than that made with the average model four times out of
five.
With the APLAT method, the prediction of a genotype in a new
environment comes at a relatively low price, using mostly available data,
except for the environmental data, which has to be recorded for every site of
the experiment, according to the crop-simulation model needs. This method seems
promising, but requires additional studies with more numerous data.
1. Introduction
Au Sahel, les interactions genotype × environnement
constatées lors des essais multilocaux et pluriannuels sont
généralement importantes. Sur les réponses moyennes par
variété et par environnement, le modèle linéaire
généralement adopté s'écrit :
Yij = m + gi + Ej +
(gE)ij + eij (1)
où Yij est la réponse du
génotype i de l'environnement j, m la moyenne
générale et gi l'effet fixe du génotype
i. L'effet Ej de l'environnement j et l'interaction
(gE)ij peuvent être fixes ou aléatoires. Pour l'objectif de
prédiction des réponses de génotypes dans l'ensemble des
environnements potentiels auxquels ils sont destinés, l'optique
aléatoire est plus pertinente. Ainsi, supposons ces deux effets et le
terme d'erreur eij aléatoires, iid et indépendants les
uns des autres avec E(Ej) =
E[(gE)ij] = E(eij) = 0 et
V(Ej) = ó2 E,
V[(gE)ij] = ó2gE et
V(eij) = ó2e
où E(·) et
V(·) désignent l'espérance et la
variance.
Choisir un génotype i dans un
environnement j suppose d'estimer l'espérance de sa performance
dans j. La précision de cette estimation est fonction
de ó2E, ó2gE et de
ó2e . Dans cette zone du Sahel,
l'environnement est variable, c'est-à-dire que
ó2E et
ó2gE sont grands, ce qui
dégrade cette précision. Pour
l'améliorer, une solution est de modéliser les variations
de Yij en fonction de l'environnement par l'utilisation de
modèles de simulation de cultures tels que DHC [1], IRSIS [2], SarraH
[3], etc. De ce fait, une partie de l'effet aléatoire de l'environnement
est reportée dans la partie fixe du modèle. Cette approche n'est
pas possible avec les modèles classiques de l'interaction
génotype × environnement. En effet, la méthode AMMI,
Additive Main effects and Multiplicative Interactions [4] ainsi que la
régression conjointe [5,6] ne tiennent pas compte des nouveaux
environnements pour y prédire les réponses des génotypes.
La régression factorielle [4,5] en tient compte, mais suppose que
l'action des variables des environnements sur la production est
linéaire, ce qui n'est pas certain.
Cependant, les paramètres des modèles de
simulation de cultures ne sont pour la plupart connus que pour un petit nombre
de génotypes, car leur évaluation demande une
expérimentation spécifique et des mesures coûteuses.
L'objectif de cette étude se pose alors en ces termes :
comment prédire le comportement de génotypes dans de nouveaux
environnements en tenant compte de ces derniers, sans coût excessif ?
2. Le modèle proposé
Si nous partons du modèle de simulation de cultures,
chacune des sorties de ce modèle, le rendement potentiel par exemple,
peut s'interpréter comme la réponse d'un génotype
i dans un environnement j :
Yij = f
(Zj,èi) +
îj + uij (2)
où Zj est le vecteur des
variables telles que la pluie, la température, etc., mesurées sur
l'environnement j et èi le
vecteur de longueur P des paramètres du génotype
i. L'erreur îj est le biais du modèle de simulation
de cultures ; nous supposons qu'elle ne dépend que de
l'environnement j : elle est donc la même pour tous les
génotypes d'un même environnement. Le terme uij est pris
aléatoire, avec E(uij) = 0 et
V(uij) = ó2 u .
Comme on l'a dit précédemment, les
paramètres des modèles de simulation de cultures ne sont
généralement connus que pour un petit nombre de génotypes.
Considérons un modèle de simulation de cultures et un
génotype de référence dont les paramètres sont
connus et appelons è0 le vecteur de ses
paramètres. Alors, supposons f de classe C1
dans un voisinage de è0 et f'
dérivable sur ce voisinage. De plus supposons
èi au voisinage de
è0. En pratique, les génotypes dont nous
chercherons à estimer leurs paramètres seront choisis de telle
sorte qu'ils ne soient pas trop éloignés du génotype de
référence. Alors, un développement en série de
Taylor à l'ordre 1 nous donne :
f (Zj,èi)
= f (Zj,è0)
+
~P ~?f ~
?è(p))
p=1
è=è0,Z=Zji
× (èi (p) - è(p)
0 ) + ?[(èi
-
è0)'(èi
- è0)]
(3)
(p
avec èi (p) et è0
) la pe composante du vecteur de paramètres
respectivement du génotype i et du génotype de
référence.
Posons X(p) j=
[ ?f
?è(p)
]è=è0,Z=Zj
: c'est une fonction de l'environnement j et
â(p)
i = è(p) i-
è(p)
0 une fonc-
tion du génotype i. La fonction X(p)
j est la dérivée partielle de la sortie
du modèle de simulation de cultures pour l'environnement j par
rapport à la pe composante du vecteur de
paramètres de la variété de référence. Comme
la fonction f n'est pas généralement connue
analytiquement, ces sensibilités peuvent être obtenues par une
méthode de dérivation numérique. Nous avons retenu tout
simplement :
X(p) = L?è(p)
jè=è0,Z=Zj
~f
(è(p)
0 + hè(p)
0 ) - f
(è(p)
0 - hè(p)
0 ) ~
~
2hè(p)
Z=Zj
0
(p
avec hè0 (p) très petit, de
l'ordre de è0 ) × 10-4 en pra-
tique. D'autres méthodes existent, celle-ci étant
la plus simple et économe en calculs.
Avec ces notations et d'après l'Éq. (2), qui
permet d'écrire f
(Zj,è0) =
Y0j - îj - u0j, nous pouvons
écrire, en négligeant ?[(èi
-
è0)'(èi
- è0)] :
Yij - Y0j =
~P
p=1
X(p) j · â(p)
i + ~ij (4)
oùfi eiji = uiji ---
u0j.. Ainsi, E(eij)) = 0,
V(eij)) =
2ó2u,,
Cov(eij ,, Eijj~) == 0, mais
Cov(eij , ~i~j)) =
ó2u..
Si nous disposons de I génotypess et de
J environnements, nous pouvons poser le modèle suivant :
Y - (Y0 ?
1I) = X · â
+ ee (5)
Le vecteur Y représente le rendement
de tous les génotypess dans tous les environnements ; ilt est de
longueur IJ,, Y'0 = (Y01l
·
· ·
· ·
· Y0J)) et
1I/ est un vecteur formé de 1, de longueur
I. Le symbol ? désigne le produit de Kronecker. Le
vecteur E est un vecteur d'erreurr aléa-
toire. Sa matrice de covariance est de la forme
ó2u?,
avec :
où
?
?
. 0 ? ?
ùj ? ?
..?
.
ùJ
2 1
ù j = (
..?
.
1 2
Les matrices ? et
ùj sont carrées de nombre de lignes,
respectivement le nombre d'observations de tous les environnements et le nombre
d'observations de l'environnement j.
Ensuite, X =
[X(1) ?
II · · ·
X(P) ? II ]
où X(p)' = [
X(p) 1· · · X(J
p) ] est de longueur J et II
est la matrice identité d'ordre I. La matrice
X est donc de dimension IJ × PI.
Enfin, â~ = [
â(1)~ · · ·
â(P )~ ] avec
â(p)~ =
[ â(p)
1 · · · â(p)
I ].
Nous proposons d'appeler cette méthode par l'acronyme
APLAT : Approximation Par Linéarisation Au-tour d'un Témoin. Elle
consiste à approcher, localement, le rendement prédit par un
modèle de simulation de cultures, par série de Taylor à
l'ordre 1 au voisinage du vecteur de paramètres d'un génotype de
référence. Cette linéarisation permet, par
régression linéaire, l'estimation des paramètres de ces
génotypes. Par la suite, la prédiction de l'écart entre le
rendement de ces génotypes et celui du génotype de
référence dans des environnements nouveaux, c'est-à-dire
où ils ne sont pas encore testés, pourra se faire si le climat de
ces derniers est connu.
3. Estimation des paramètres et validation du
modèle
Il y a en général beaucoup de paramètres
dans un modèle de simulation de cultures et peu d'environnements dans un
essai multienvironnement, ce qui rend souvent PI grand par rapport
à IJ. Pour notre exemple, nous avons utilisé SarraH
comme modèle de simulation de cultures. Ce modèle dispose de 61
paramètres, qui sont fonction du génotype. Avec un tel nombre de
prédicteurs, l'estimation de â s'est
faite par régression PLS, Partial Least Squares [7]. Il s'agit
donc pour nous d'écrire un modèle linéaire de
prédiction des rendements des génotypes pour de nouveaux
environnements par les sensibilités par rapport aux paramètres
des génotypes des sorties d'un modèle de simulation de cultures,
fondé sur la construction de composantes orthogonales dans l'image
de X. Ceci permet de réduire l'espace des
régresseurs de rang de X à k
dimensions. La régression PLS s'effectue selon le principe de
l'algorithme NI-PALS, Nonlinear estimation by Iterative Partial Least
Squares [7], où un ensemble de régressions partielles par
moindres carrés ordinaires est effectué, en même temps que
le calcul des composantes. Ici, la matrice de covariance de
E est égale à
ó2u? et
non à
ó2uIIJ .
La solution serait d'effectuer toutes les régressions partielles par
moindres carrés généralisés. Mais cette matrice de
covariance est inconnue. Elle s'écrit tout de même, à une
constante multiplicative près, en fonction de
?, qui elle est connue. La
matrice ? étant symétrique et
semi-définie positive, par décomposition de Cholesky, il existe
une matrice ç tel que
ç~ç
= ?-1.
Ainsi, estimer â par PLS avec
les régressions partielles par moindres carrés
généralisés consiste à poser le modèle
suivant :
çY -
ç(Y0 ?
1I) = çX
· â + çe
(6)
où âPLS est
l'estimation avec les régressions partielles effectuées par
moindres carrés ordinaires.
Dans ce cas, la variance de l'erreur
çc s'écrit :
E(ç~E~ç~)
=
çE(ce)ç'
=
ó2uç?ç'
=
ó2uç(ç'ç)-1ç~
2
=
óuçç
-1(çr)-1ç/
= ó2uIIJ
Le nombre de composantes à retenir est
déterminé par le PRESS, Prediction Error Sum of Squares
[7].
Nous avons calculé les intervalles de confiance des
coefficients estimés par la méthode bootstrap [8]. Cette
technique permet d'estimer la loi inconnue d'un estimateur par une loi
empirique obtenue à partir d'une procédure de
rééchantillonnage fondée sur des tirages aléatoires
avec remise des données. Les intervalles de confiance construits sont de
type percentile-t [9]. Soit
(p)*b
zi,PLS la variable aléatoire définie par
:
(p)*b zi,PLS =
(7)
â(p)~b
i,PLS -
â(p)
i,PLS
s*(
â(p)~b i,PLS)
où âi(,pPlS est le (p
· i)e élément de
âPLS,
âi(,PL p)*S obtenu
au be tirage avec b = 1, . . .
, B et s( â(p)~b
i,PLS) l'écart-type
estimé de
â~bPLS. Soit 'FB la fonction de
répartition empi-
rique des z(p)~b
i,PLS. Le fractile d'ordre á,
%1 (á) est estimé
par la valeur àt(á) telle que :
1
B
~B
b=1
(p)*bà= á
{zi,PLS t(á)}
Donc un intervalle de confiance percentile-t pour le
(p.i)e élément de â
peut s'écrire :
[ â(p)
i,PLS - s~ â(p) ~
· àt(1 - á),
â(p)
i,PLS - s( â(p) )
· àt(á)]
i,PLS i,PLS
(8)
L'évaluation de la qualité du modèle
proposé est faite avec l'erreur quadratique moyenne de prédiction
MSEP, Mean Squared Error of Prediction [10]. La MSEP est
utilisée comme critère pour comparer différents
modèles dont le modèle moyen [11], défini pour nos
donnés par:
Yij = m + gi + Ej +
äij (9)
où m est la moyenne de la population et gi
l'effet génotype. L'effet Ej de l'environnement j
est supposé aléatoire, d'espérance nulle et de
variance ó2 E. Les erreurs
äij sont indépendantes, d'espérance
nulle et de variance ó2 ä . De plus, Ej
et äij sont supposés
indépendants.
Le logiciel R [12] a été utilisé la
fonction qui a servi pour les régression est de J.-F. Durand [13].
4. Les données utiisées
Nous avons des résultats d'essais agronomiques
d'arachide menés de 1994 à 1998 sur la station
expérimentale du Ceraas, située à Bambey
(14?42N et 16?28O), au Sénégal. Ces essais
pluriannuels ont concerné au total 26 génotypes à cycle de
développement de 90 jours et répondaient à l'objectif de
recherche de génotypes physiologiquement adaptés à la
sécheresse.
La variété de référence choisie
est la 55-437, c'est une variété hâtive de 90 jours; elle a
donc une longueur de cycle proche de celle des autres variétés
utilisées. Elle a été choisie parce que ses données
étaient disponibles.
Dans ce milieu à forte variabilité des pluies
dans l'espace et même dans le temps pour un même lieu, nous avons
considéré chacune des cinq années d'expérimentation
comme un environnement (Fig. 1).
Pour valider notre modèle, nous avons
réservé successivement chacune des années et estimé
les paramètres des génotypes sur les années restantes.
Pour chaque année, les rendements observés ont été
comparés à ceux prédits par la méthode APLAT. Les
rendements sont exprimés en kilogrammes de gousses par hectare.
SarraH a été utilisé pour calculer
X. Compte tenu du nombre de données disponibles, seuls
deux paramètres (P = 2) ont été
considérés parmi les 61 de SarraH. Le premier paramètre
est en fait un coefficient multiplicateur qui agit sur cinq paramètres
de SarraH : coefficient
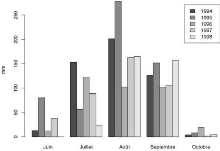
Fig. 1. Répartition des pluies sur la station de Bambey,
au Sénégal, de 1994 à 1998.
moyen d'angle des feuilles, coefficient de conversion en
assimilat, coefficient d'efficience d'assimilation des feuilles à la
phase végétative juvénile, coefficient d'efficience
d'assimilation des feuilles à la première phase de maturation,
phase sensible de remplissage des grains et coefficient d'efficience
d'assimilation des feuilles à la deuxième phase de maturation,
phase non sensible. Le deuxième paramètre est le poids moyen des
gousses.
5. Résultats
Au Sahel, l'interaction G×E est largement due aux
aléas climatiques, dont la probabilité peut être
estimée à l'aide de longues chroniques de relevés
météo au sol. Cependant, relier l'interaction G×E et la
pluviométrie à l'aide d'un modèle de simulation de
cultures n'est habituellement possible que pour des variétés dont
on a estimé les paramètres, au prix d'une expérimentation
spécifique. Le modèle APLAT permet de prédire cette
interaction avec les seules données d'une expérimentation
multilocale classique, sans autre instrumentation que des stations
météo simples.
Pour les modèles sans les données respectivement
de 1994, 1995 et 1997, le PRESS minimal est atteint avec six composantes. Pour
les deux autres modèles, le PRESS est minimal avec neuf composantes,
mais nous avons réduit leur espace à cinq dimensions, car le
PRESS n'y est pas trop différent de ses valeurs mini-males (Fig. 2).
Les coefficients des régressions PLS et les intervalles
de confiance qui leur sont associés sont représentés sur
la Fig. 3.
Les MSEP estimées pour les modèles APLAT, sauf
celle sans les données de l'année 1998, sont inférieures
aux MSEP des modèles moyens correspondants (Tableau 1). Ce qui signifie
que, pour ces modèles, pré-
Fig. 2. Evolution du PRESS en fonction du nombre de composantes.
Le modèle (-1994) utilise les données, sauf celles de
l'année 1994, et ainsi de suite.
Fig. 3. Intervalle de confiance percentile-t à
95 % des coefficients estimés. Le modèle (-1994) utilise les
données, sauf celles de l'année 1994, et ainsi de suite. Sur
l'axe des abscisses figurent les génotypes par ordre alphabétique
pour chacun des deux paramètres. Le symbole représente
l'estimation des coefficients.
Tableau 1
MSEP des différents modèles APLAT et
modèles moyens correspon-
dants. Le modèle (-1994) utilise les
données, sauf celles de l'année
|
1994, et ainsi de suite
|
|
|
|
APLAT
|
Modèle moyen
|
|
Modèle (-1994)
|
24687,3
|
64651,6
|
|
Modèle (-1995)
|
5915,0
|
7160,6
|
|
Modèle (-1996)
|
35446,1
|
37814,8
|
|
Modèle (-1997)
|
10038,3
|
18201,1
|
|
Modèle (-1998)
|
118304,9
|
84963,6
|
dire le rendement par la méthode APLAT est meilleur que
par la moyenne des rendements du passé. Ainsi, quatre fois sur cinq, la
méthode APLAT s'est révélée meilleure que le
modèle moyen. Toutefois, cette étude souffre de la faible taille
de notre échantillon.
6. Conclusion
La méthode APLAT peut être vue comme un outil
d'aide à la décision pour la sélection au Sahel. Dans
l'exemple où un sélectionneur doit tester plusieurs
génotypes dans un nouvel environnement, cette méthode lui
permettra d'écarter d'emblée certains génotypes qui
donneront une production faible, en lieu et place d'essais multilocaux ou
pluriannuels dans ces environnements contrastés ou d'une tentative de
paramétrisation d'un modèle de simulation de cultures qui
implique un coût élevé. Son attention sera portée
par la suite sur l'ensemble restreint des génotypes retenus avec APLAT,
où il pourra appliquer les schémas classiques de
sélection.
Cette nouvelle approche semble prometteuse, mais il faut des
études supplémentaires. Notamment disposer de données
agronomiques plus conséquentes pour l'éprouver.
Remerciements
Nous remercions Danièle Clavel pour les données
de l'étude et Jean-Claude Combres pour toutes les discussions autour du
modèle SarraH.
Références
[1] AGRHYMET, Bulletins décadaires et mensuels de suivi
de la campagne agricole pluviale, Niamey, 1991.
[2] FAO, IRSIS, Irrigation scheduling information system, Rome,
1987.
[3] C. Baron, Modèle de bilan hydrique et de croissance
des plantes céréales : Mil Sorgho et Arachide, Cirad, 2002.
[4] M. Vargas, J. Crossa, F.v. Eeuwijk, K.D. Sayre, M.P.
Reynolds, Interpreting treatment × environment interaction in agronomy
trials, Agron. J. 93 (2001) 949-960.
[5] J.-B. Denis, P. Vincourt, Panorama des méthodes
statistiques d'analyse des interactions génotype × milieu,
Agronomie 2 (1982) 219-230.
[6] S.A. Eberhart, W.A. Russel, Stability parameters for
comparing varieties, Crop Sci. 6 (1966) 36-40.
[7] M. Tenenhaus, La Régression PLS : théorie et
pratique, Technip, Paris, 1998.
[8] B. Efron, Bootstrap methods: another look at the jackknife,
Ann. Stat. 7 (1979) 1-26.
[9] S. Aji, S. Tavolaro, F. Lantz, A. Faraj, Apport du
bootstrap à la régression PLS : application à la
prédiction de la qualité des gazoles, Oil Gas Sci. Technol.-Rev.
IFP 58 (2003) 599-608.
[10] D. Wallach, B. Goffinet, Mean squared error of
prediction in models for studying ecological and agronomic systems, Biometrics
43 (1987) 561-573.
[11] J. Colson, D. Wallach, A. Bouniols, J. Denis, J. Jones,
Mean squared error of yield prediction by SOYGRO, Agron. J. 87 (1995)
397-407.
[12] R Development Core Team, R: A language and environment
for statistical computing, R Foundation for Statistical Computing, Vienna,
Austria, ISBN 3-900051-07-0, 2004, URL
http://www.R-project.org.
[13] J.-F. Durand, Calcul matriciel et analyse factorielle des
données, université Montpellier-2, Montpellier, France, 2002.
| 


