|

1 íØÓ
ÓÇÈÚ ÊÇÍÑ
ÉÚãÇÌ
Univesité Ferhat Abbas Sétif
1
Faculté des Sciences de la
Nature et de la Vie
Éíáß
ÉÇíÍáÇ
J
ÉÚíÈØáÇ
ãæáÚ
DEPARTEMENT DE MICROBIOLOGIE N°
/SNV/2017
MEMOIRE
Présenté par:
REFOUFI KHALIL ABDALLAH
Pour l'obtention du
diplôme de
MASTER EN BIOLOGIE
Filière: Biologie
Spécialité: Microbiologie
Appliquée
THèME
Soutenue publiquement le : 01/07/2017
DEvANT LE JURY
Président : Dr. Silini Allaoua MCA UFA SETIF1
Promoteur : Dr. Silini-Cherif Hasfa MCA UFA SETIF1
Co-promoteur : Pr. Sahli Farida Pr CHU/UFA SETIF1
Examinateur : Dr. Yahiaoui Bilal MCA UFA SETIF1
Laboratoire de Microbiologie Appliquée
2016/2017
DEDICACE
Je dédie ce modeste travail à celle qui m'a
donné la vie le symbole de tendresse qui s'est sacrifiée
pour mon bonheur et ma réussite, à la
lumière de ma vie íã
A ma cher grand mère Malika
A mes frères Mohsen et Ayoub que J'aime
beaucoup
A mes soeurs Hadjer et Hafsa que j'aime autant
A ma cher époue Aicha qui m'a aidé tout ce
parcours de Master
A mes deux petites puces Ritedj et Djenat
A mes oncles TOUFIK, SEDDIK, LAZHER, AZZEDINE, AHMES et
FETHEDDINE
A mes tentes NABILA, HOURIA, SAMIA, KHAOULA, LAMIA, et
HANANE
A tout mes cousins et cousines
A mes meilleurs amis KAMAL et FOUZI
A Moumeni A/Krim et Zerroug Imad qui mon soutenue
A Radjai Issam prof. d'Anglais qui ma beaucoup
aidé
A mes collégues de ces deux années de Master
surtout Kebaili Oussama, Boutaghane, Méziane
Amine, Marouani Kaouther et Soltani Nassima qui mon
aidés a intégré A ma famille REFOUFI et GHODBANE
Je souhaite remercie aussi toute l'équipe du
Laboratoire surtout HARBOUCHE Wided, KHABCHACHE Hasna, BOULAHYA Zineb,
GUETTOUCHE Houda, AYADI Hayet, DIABI
REMERCIEMENT
Avant tout, je remercie le bon Dieu tout puissant qui m'a
donné la force et de m'avoir permis d'arriver à ce
stade-là.
Ce travail a été effectué au sein de deux
laboratoires : Laboratoire de « Microbiologie »,
Hôpital « SAADNA Mohamed Abdennour » de
Sétif, Algérie ; et Laboratoire de recherche à
l'Université FERHAT Abbas, Faculté des Sciences Nature et Vie, El
BAZ.
Mes remerciements s'adressent d'abord, à mon directeur
de thèse, le Docteur SILINI-CHERIF Hafsa. Je la remercie pour son
encadrement, pour ses encouragements et sa rigueur scientifique au quotidien
durant ce modeste travail. Merci de m'avoir fait confiance et m'a
encouragé pour la réalisation de mon mémoire de fin
d'étude.
Je remercie également, ma co-directrice de
thèse, Madame le Professeur SAHLI Farida pour ses aides et ses
orientations.
C'est avec un grand plaisir que je remercie le Docteur SILINI
Allaoua, pour l'honneur qu'il m'a fait en acceptant de présider le jury
de cette thèse, que ce travail constitue le témoignage de notre
respect.
Je tiens également à exprimer mes plus profonds
remerciements au Docteur YAHIAOUI Bilal, pour sa disponibilité, et pour
l'honneur qu'il m'a fait en acceptant d'examiner ce travail. Je le remercie
pour ses précieux conseils et ses encouragements.
Je remercie le Docteur MESSAI Chafik Redha pour l'aide et les
conseils qu'il ma donné. Je voudrais désormais remercier Mr BIAZ
Mustapha, le chef de service du laboratoire de
Microbiologie, CHU de Sétif ; pour m'avoir accueilli au
sein de son laboratoire, pour ses discussions scientifiques et son ouverture
d'esprit. Je le remercie pour sa confiance, son soutien, ses encouragements, et
son affection.
Mes vifs remerciements s'adressent également a Mme
YAHIA-CHERIF-BOUNECHADA Nora de m'avoir aidé tout au long de ma
présence au Laboratoire de Sétif.
Nardjes, BOUDJLLEL Naziha, CHERRAD Hasna, Marwa. Je remercie
aussi tous mes collègues et mes enseignants, ainsi que les personnels
administratifs de l'université FERHAT Abbas.
Merci enfin à l'ensemble des personnes qui m'ont
aidé de prés ou de loin dans l'élaboration de ma
thèse.

SOMMAIRE
Listes de tableaux Listes des figures
Liste des abreviations
Résumé
Introduction 1
CHAPITRE 1 : SYNTHESE BIBLIOGRAPHIQUE
1-Les entérobactéries 3
1-1-Définition 3
1-2-Classification 3
1-3-Caractères bactériologiques 3
1-4-Caractères biochimiques 4
1-5-Caractères antigéniques 4
1-6-Pouvoir Pathogène 4
2-Les f3-lactamines 7
2-1-Classification des f3-lactamines 7
2-1-1-Les pénicillines (noyau péname) 8
2-1-2-Les céphalosporines (noyau céphème)
8
2-1-3-Les pénèmes (Carbapénèmes)
10
2-1-4-Les monobactames 10
2-1-5-Les inhibiteurs des f3-lactamases 10
2-2-Mode d'action des f3-lactamines 12
3-Résistance des entérobactéries aux
f3-lactamines 12
3-1-Types de résistance 12
3-1-1-La résistance naturelle 13
3-1-2-La résistance acquise 13
3-2-Supports génétiques de la résistance
14
3-2-1-Le chromosome 14
3-2-2-Les éléments génétiques
mobiles 14
a) Plasmides 15
b) Séquences d'insertion et transposons 16
c) Les intégrons 16
3-3-Mécanismes de résistance aux f3-lactamines
17
3-3-1-Diminution de la perméabilité 17
3-3-2-Modification de la cible 17
3-3-3-Hyperproduction du système d'efflux 17
3-3-4-Production d'enzymes 18
4-Les f3-lactamases 18
4-1-Classification des f3-lactamases 19
4-1-1-Classification d'Ambler 19
4-1-2-Classification de Bush-Jacoby-Medeiros 20
5-Les f3-lactamases à spectre élargi BLSE 22
5-1-Différents types de BLSE 22
5-1-1-BLSE de type TEM (Temoneira - nom du patient) 22
5-1-2-BLSE de type SHV : (Sulfhydryl variable) 24
5-1-3-BLSE de type CTX-M (Cefotaximase-Munich) 25
5-1-4-Autres types de BLSE 25
5-1-5-Les Carbapénèmases 26
a)Classification des carbapénèmases 26
6-Epidémiologie mondiale des
entérobactéries BLSE 29
6-1-Les BLSE classiques 29
6-1-1-En Europe 29
6-1-2-En Afrique 29
6-1-3-En Asie 30
6-1-4-En Amérique 31
6-2-Les BLSE de type carbapénèmases 32
7-Facteurs de risque d'acquérir une BLSE 33
CHAPITRE 2 : MATERIEL ET METHODES
1-Prélèvement et identification 34
1-1-La mini galerie (galerie classique) 34
1-2-La galerie Api 20E 35
1-3-Le systeme MicroScan WalkAways SIEMENS 35
2-Antibiogramme 36
3-Détection phénotypique des EBLSE 37
3-1-Test de synergie 38
3-2-Test du double disque 38
3-3-Test à la cloxacilline 39
4-Détection génotypique des EBLSE 40
4-1-Extraction d'ADN plasmidique par la lyse alcaline 40
a) Extraction d'ADN 40
b) Extraction de l'ADN chromosomique par Boiling 41
4-2-Amplification par PCR (Polymerase Chain Reaction) 41
4-2-1-Principe 41
4-3-Electrophorèse sur gel d'agarose 43
CHAPITRE 3 : RESULTATS ET DISCUSSION
Résultats 44
1-Entérobactéries 44
1-1-Répartition des entérobactéries par
espèces 44
1-2-Répartition des entérobactéries selon
l'origine de l'infection 45
1-3-Pourcentage des entérobactéries selon le
sexe 45
1-4-Répartition des entérobactéries selon
les tranches d'âge 46
1-5-Profil de résistance des
entérobactéries aux antibiotiques 47
1-6-Profil de résistance des
entérobactéries aux antibiotiques par année 47
a) Résistance aux f3-lactamines 47
b) Résistance aux autres antibiotiques 48
2-Les EBLSE 49
2-1-Fréquence d'isolement des EBLSE 49
2-2-Répartition des EBLSE selon l'espèce 50
2-3-Répartition des EBLSE selon l'origine de
l'infection 50
2-4-Répartition des EBLSE selon le service 51
2-5-Répartition des EBLSE selon la nature du
prélèvement 52
2-6-Répartition des EBLSE selon le sexe 52
2-7-Profil de résistance des EBLSE 53
2-7-1-Résistance aux f3-lactamines 53
2-7-2-Résistance aux aminosides 54
2-7-3-Résistance aux quinolones 54
2-7-4-Résistance aux autres antibiotiques 54
3-Support génétique de la résistance
55
4-Caractérisation des BLSE par la PCR-multiplex 58
4-1-BLSE de type CTX-M 58
4-2-BLSE de type SHV 60
4-3-BLSE de type TEM 60
4-4-Résistances multiples 60
Discussion 63
Conclusions et Perspectives 69
Références bibliographique
72
Annexe
Liste des tableaux
Tableau 1. Classification des f3-lactamines
11
Tableau 2. Classification global des BLSE
21
Tableau 3. Principales
carbapénémases des entérobactéries 27
Tableau 4. Antibiotiques testés 37
Tableau 5. Composition des différents
tampons d'extraction 40
Tableau 6. Amorces utilisées 41
Tableau 7. Etapes de la PCR-Multiplex 42
Tableau 8. Différents types de
plasmides extraits des EBLSE 56
Tableau 9. Caractéristiques
générales et moléculaires des souches
d'entérobactéries
productrices de f3-lactamases à spectre étendu
isolées au CHU de Sétif 61-62
Liste des figures
Figure 1. Structures chimiques des principales
f3-lactamines et inhibiteurs de f3-lactamases... 8
Figure 2. Structure chimique de la ceftaroline
10
Figure 3. Modes de transmission du matériel
génétique : la conjugaison, la transformation et
la transduction chez les bactéries 15
Figure 4. Mode d'action des f3-lactamases 18
Figure 5. Les BLSE dérivées de TEM
23
Figure 6. Les BLSE dérivées de SHV
24
Figure 7. Disposition des disques d'antibiotique
pour le test de synergie 38
Figure 8. Schéma de détection de
BLSE par le test du double disque 39
Figure 9. Photo du thermocycleur TC-4000 42
Figure 10. Photo d'une migration d'ADN par
électrophorèse 43
Figure 11. Photo du transilluminateur
Fisher-Bioblock UV 43
Figure 12. Répartition des
entérobactéries selon l'espèce 44
Figure 13. Pourcentages des
entérobactéries selon l'origine de l'infection 45
Figure 14. Pourcentages des souches
entériques selon le sexe 46
Figure 15. Répartition des
entérobactéries selon les tranches d'âge 46
Figure 16. Pourcentages de résistance des
entérobactéries aux antibiotiques 47
Figure 17. Pourcentages de résistance des
entérobactéries aux f3-lactamines par année 48
Figure 18. Pourcentages de résistance des
entérobactéries aux autres antibotiques par année 49
Figure 19. Fréquence des EBLSE 49
Figure 20. Répartition des EBLSE par
espèce 50
Figure 21. Répartition des EBLSE selon
l'origine de l'infection 51
Figure 22. Répartition des EBLSE selon
le service 51
Figure 23. Répartition des EBLSE selon
la nature du prélèvement 52
Figure 24. Répartition des EBLSE selon
le sexe 52
Figure 25. Test de synergie 53
Figure 26. Test de synergie vu dans un
antibiogramme d'une souche Klebsiella pneumoniae
BLSE+ 54
Figure 27. Profil de résistance des
EBLSE aux antibiotiques 55
Figure 28. Profil plasmidique des 56 souches
d'EBLSE 57
Figure 29. Amplifications par PCR-Multiplex
des gènes blaCTX-M, blaSHV et blaTEM 59
Figure 30. Prévalence des gènes
de résistance chez les souches amplifiées 60
Figure 31. Stratégies et les cibles
bactériennes utilisées pour lutter contre la résistance
aux. 71
Liste des abréviations: ATB :
Antibiogramme.
BCP : Bromo-Créso-Phénol.
bla : bêta-lactamse.
BLSE : 3-Lactamase à Spectre Elargi.
C1/2/3/4/5G : Céphalosporine de
1ére /2éme /3éme
/4éme /5éme Génération.
Cfr : Citrobacter freundii.
CHU : Centre Hospitalo-Universitaire.
CMI : Concentration Minimale Inhibitrice.
CTX-M : Céfotaximase-Munich.
DAEC : E. coli à
Adhérence Diffuse.
Eae : Enterobacter
aerogenes.
EAEC : E. coli
Entéroagrégatives.
EBLSE : Entérobactéries
3-Lactamase à Spectre Elargi.
Ecl: Enterobacter cloacae.
Eco: Escherichia coli.
EHEC : E. coli
Entérohémorragiques.
EIEC : E. coli
Entéroinvasives.
Enz : Enzyme.
EPEC : E. coli
Entéropathogènes.
ETEC : E. coli
Entérotoxinogènes.
IR : Intégrons de
Résistance.
kpb : kilos paire de base.
KPC : Klebsiella pneumoniae
Carbapénémase.
Kpn: Klebsiella
pneumoniae.
KT : cathéter.
LCR : Liquide Céphalo-Rachidien.
LP : Liquide Pleural.
McF : McFarland.
MH: Muler-Hinton.
Mmo: Morganella morganii
MYSTIC: Meropenem Yearly Susceptibility Test
Information Collection.
MâL :
Métallo-â-Lactamase.
OMS : Organisation Mondiale de la
Santé.
ONPG : OrthoNitrophényl
b-D-GalactoPyranoside.
pb : paire de base.
PB : Prélèvement Buccal.
PBP: Penicillin Binding Proteins.
PCR: Polymerase Chain Reaction.
PLP : Protéines de Liaison aux
Pénicillines.
PR : Prélèvement Rectal.
PV : Prélèvement Vaginal.
RM : Rouge Méthyle.
Ser : Sérine.
SHV: Sulfhydryl Variable.
SI : Séquence d'Insertion.
Sma : Serratia
marcescens.
TEM: Temoneira.
TRI : TEM Résistantes aux
Inhibiteurs.
TSI : Triple Sugar Iron.
VP : Voges-Proskauer.
WHO: World Health Organization.
Mots clés :
Entérobactéries, BLSE, plasmides, PCR-multiplex.
Résumé
Résumé
L'évaluation de la résistance aux antibiotiques
des entérobactéries provenant de malades externes ou
hospitalisés dans les différents services du CHU de Sétif,
durant la période de Janvier 2013 à Avril 2017 porte sur les
données du logiciel de sauvegarde «Whonet 5.6 » utilisé
au laboratoire.2421 souches d'entérobactéries isolées sont
identifiées. E. coli est la souche la plus
fréquente (n= 1160), suivie par K. pneumoniae (n=479) et
P.mirabilis (n=216). Ces souches proviennent majoritairement du milieu
hospitalier (57.71 %) et semblent coloniser les adultes plutôt que les
enfants où le sexe féminin représente un facteur de
risque. Le profil de résistance de ces entérobactéries
atteste d'un taux très élevé de résistance aux
bêtalactamines. Les valeurs maximales sont notées pour
l'amoxiclline où 98.40% des
souches sont résistantes et les plus faibles
résistances sont notées pour les
carbapénèmes
(l'imipénème et l'ertapénème) et la cefoxitine. Les
56 entérobactéries productrices de BLSE isolées durant
l'année 2017 représentent 35% de la totalité des
entérobactéries isolées pendant cette période.
Elles sont réparties comme suit : 23 E. coli, 22 K.
pneumoniae, 4 E. cloacae, 3 M. morganii, 2 S.
marcescens, 1 E. aerogenes et 1 C. freundii.
Elles sont plutôt issues d'infections nosocomiales (75%)
que communautaires (25%). Les services les plus concernés sont
l'infectieux et la néphrologie et les prélèvements les
plus incriminés sont les urines et le pus.
Ces souches EBLSE ont des résistances très
élevées (100%) à la majorité des â-lactamines
(AMX, AMP, TIC, C3G, C4G et l'aztréonam). Alors que l'IMP, ERT, MER, AMK
et FOS restent les antibiotiques les plus actifs. Le profil plasmidique des
souches EBLSE confirme qu'elles peuvent héberger de 1 à 4 types
de plasmides. La caractérisation des gènes des BLSE
effectuée sur l'ADN (plasmidique ou chromosomique) de 33 souchespar
l'amplification multiple des gènes (CTX-M, TEM et SHV)
a permis l'obtention d'une
prévalence de CTX-M (87.87%) suivie de SHV
(42.42%) et de TEM (27.27%). Toutefois, cinq souches K. pneumoniae
(n=4) et E. aerogenes (n=1) semblent porter les 3 gènes de
résistance en même temps, indiquant la gravité de ce
phénomène.
Key Words: Enterobacteria, BLSE, plasmids,
PCR-multiplex.
Résumé
Abstract
The evaluation of the resistance to the enterobacteria
resulting from external patients or hospitalized in the different services of
Setif CHU, from January 2013 to April 2017.
Carried out on the data of the saveguard software «Whonet
5, 6», utilized at laboratory. 2421 isolated strains of enterobacteria are
identified. E.coli the most frequent strain (n = 1160), followed by
K.pneumonia (n=479) and P.mirabilis (n=216).
Theses strains usually come from the hospital milieu (57.71%)
and seem to attain adults rather than children where the feminine sex
represents a risk factor.
The resistance profile of theses enterobacteria manifests a
higher rate of resistance to beta-lactamines.
The maximal values are noted for amoxicilline where (98.40%)
of the strains are resistant and the weakest resistance is noted for the
carbapenemes (imipenemes and ertapenemes) and the cefoxitine. The 56
enterobacteria which produces BLSE isolated along the year 2017 represent 35%
of the whole: 23 E. coli, 22 enterobacteria previously isolated during
this period. They are divided as follows: K. pneumoniae, 4 E.
cloacae, 3 M. morganii, 2 S. marcescens, 1 E.
aerogenes and 1 C. freundii. They are the
outcome of nosocomial infections (75%) rather than communitory (25%).The mostly
concerned services are the infection and the nephrology services and the most
requested samples are the urines and the puss.
Theses EBLSE strains possess a higher resistance (100%) to the
majority of B-lactamines (AMX, AMP, TIC, C3G, C4G and aztreonam). While IMP,
ERT, MER, AMK and FOS remain the most active antibiotics. The plasmidic profile
of the EBLSE strains confirms that they can host from 1 to 4 types of plasmids.
The EBLSE genes characterization carried out on DNA (plasmidic or chromosomic)
of 33 strains via the multiple amplification of the genes (CTX-M, TEM and SHY)
allowed the obtainment of a prevalence of CTX-M (87.87%) followed by SHY
(42.42%) and by TEM (27.27%). However, five strains K. pneumonia (n=4)
and E. aerogenes (n=1) seem to carry 3 genes of resistance at the same
time, a factor that clearly indicates the seriousness of this phenomenon.
INTRODUCTION
1
Introduction
Introduction
Depuis la découvert des antibiotiques en 1928 par
Alexander Fleming et dés leur utilisation durant la seconde guerre
mondiale, les antibiotiques de la famille des â-lactamines ont
été confrontés à l'émergence de la
résistance.
Cependant, la résistance bactérienne aux
antibiotiques est en perpétuelle évolution. Cette
résistance est la résultante d'interactions complexes entre la
bactérie d'une part et son environnement d'autre part. Elle est
liée essentiellement à un usage excessif des antibiotiques aussi
bien en médecine humaine, qu'en médecine
vétérinaire ou dans l'alimentation animale (Bradford. 2001b). Les
bactéries, pour faire face à la pression de sélection
exercée par les antibiotiques utilisent des parades leur permettant de
s'adapter aux conditions hostiles de leur environnement (Benredjeb et
al. 2000).
Selon le rapport de l'OMS en 2014, sur la surveillance
mondiale de la résistance aux antimicrobiens, la résistance aux
antibiotiques est une réalité existant partout dans le monde et
constitue désormais une grave menace pour la santé publique
(Anonyme. 2014). En effet, on assiste à l'émergence et à
la dissémination des bactéries multirésistantes par
hyperproduction de céphalosporinase (AmpC) ou production de
bêtalactamases à spectre élargi (BLSE) ou encore de
carbapénémases.
Jusque dans les années 2000, la diffusion des
entérobactéries productrices de BLSE concernait essentiellement
le milieu hospitalier, de nombreuses épidémies
hospitalières en réanimation ou en hospitalisation de longs
séjours ayant été décrites. Mais aujourd'hui, la
diffusion à grande échelle dans le domaine communautaire de ce
type de résistance laisse augurer un problème majeur de
santé publique. Les infections causées par les souches
productrices de BLSE sont associées à une morbidité et une
mortalité élevées, à une prolongation de la
durée de l'hospitalisation et à une augmentation des coûts
d'hospitalisation.
Découvertes au début des années 1980, les
BLSE sont une grande famille d'enzymes bactériens capables d'hydrolyser
les pénicillines, les quatre générations de
céphalosporines et l'aztréonam. Elles n'hydrolysent pas les
carbapénèmes ni les céphamides. Elles sont
retrouvées essentiellement dans la famille des
entérobactéries, principalement Escherichia coli et
Klebsiella spp. (Bradford, 2001a).
La plupart des BLSE détectées auparavant
étaient les types classiques TEM et SHV qui diffusaient majoritairement
chez des souches de Klebsiella pneumoniae et d'Enterobacter
spp
2
Introduction
étroitement associés à des infections
nosocomiales. Actuellement, un nouveau type d'enzyme, le CTX-M dissémine
au sein des souches communautaires. Les gènes blaTEM, blaSHV,
blaCTX-M, blaOXA ont été décrits dans plusieurs
études épidémiologiques. La résistance
bactérienne par production de BLSE est devenue un problème de
santé publique mondiale car compromettant gravement les thérapies
antimicrobiennes.
De ce fait, le but de ce travail est l'évaluation de la
résistance aux f3-lactamines, des souches
d'entérobactéries d'isolement courant dans le CHU de
Sétif, afin de déterminer la fréquence de
résistance aux céphalosporines de 3éme
génération et des résistances associées au sein des
espèces.
Par la suite, une caractérisation par la technique
moléculaire (PCR) des f3-lactamases dessouches
d'entérobactéries productrices pour mettre en évidence le
degré de leur diversité génétique.
CHAPITRE 1
SYNTHESE
BIBLIOGRAPHIQUE
3
Chpaitr 1 : Synthese bibliographique
1- Les entérobactéries 1-1-
Définition
Les entérobactéries sont des bactéries
commensales ou pathologiques du tube digestif de l'homme et des animaux,
d'où leur nom (entéro= intestin grêle). Elles peuvent
être transitoirement présentes, en dehors du tube digestif, sur
différentes parties du revêtement cutanéo-muqueux (Nauciel
et Vildé. 2005) et également dans l'environnement. Certaines sont
des pathogènes obligatoires provoquent dès leur entrée
dans le tube digestif des malades. D'autres, présentent un
caractère opportuniste, elles ne sont pathogènes que sur fond
d'immunodépression. D'autres groupes prolifèrent dans
l'environnement et peuvent être une source de contamination (Grosjean et
al. 2011).
1-2- Classification
La position phylogénétique de la famille des
Enterobacteriaceae se trouve dans l'ordre des Enterobacteriales de la classe
des Gammaproteobacteria. Cette famille comprend 176 espèces
nommées ou plus, répertoriées en 44 genres (Garrity et
al. 2005), dont les plus récents sont Alterococcus,
Arsenophorus, Brenneria, Pectobacterium, Raoultella, Samsonia et Sodalis.
Les relations phylogénétiques des genres sont fondées
sur l'analyse des séquences de l'ARNr 16S des souches types de
l'espèce (Garrity et al. 2005).Les genres de cette famille sont
regroupés en cinq tribus, d'après leurs propriétés
fermentatives : Escherichiae, Klebsielleae, Proteae, Yersiniae et
Erwiniae.
1-3- Caractères
bactériologiques
Les Enterobacteriaceae sont des bacilles à Gram
négatif mobiles ou immobiles, avec des extrémités
arrondies de 0.5- 1.5 ìm d'épaisseur et 2-4 ìm de
longueur, parfois capsulés. Leurs exigences nutritionnelles sont, en
général, réduites se développent bien dans un
bouillon ou sur gélose ordinaire incubés 18 heures à
37°C. Sur milieu gélosé, ils peuvent donner
différentes formes de colonies. Les formes S (smooth) sont l'aspect
habituel des colonies lisses, bombés, brillantes et humides. Les formes
R (rough) s'observent surtout avec les souches ayant subi plusieurs repiquage
où les colonies sont rugueuse, sèches, à contours
irréguliers et de teinte mate (Avril et al. 2000).
4
Chpaitr 1 : Synthese bibliographique
1-4- Caractères biochimiques
L'identification des Enterobacteriaceae repose sur
l'étude des caractères biochimiques. Elles donnent une
réaction d'oxydase négative, acidifient le glucose par
métabolisme fermentatif, avec ou sans production de gaz et
réduisent les nitrates en nitrites. Des galeries biochimiques permettent
de déterminer avec précision le genre et l'espèce, se
basant sur :
· Production d'hydrogène sulfuré
· Recherche de l'uréase.
· Production d'indole.
· Recherche des décarboxylases.
· Recherche des désaminases oxydatives.
· Utilisation du citrate.
· Utilisation du mannitol.
· Recherche de l'acétoïne ou réaction
Voges-Proskauer (VP).
· Test à l'ONPG (Orthonitrophényl
b-D-Galactopyranoside).
1-5- Caractères antigéniques
L'identification des antigènes et sérogroupes a
permis de différencier les souches pathogènes des souches
commensales. En effet, certains sérotypes ne sont jamais, ou rarement,
associés à des maladies tandis que d'autres le sont très
fréquemment.
L'antigène somatique O, définissant le
sérogroupe, est contenu dans les lipopolysaccharides présents sur
la paroi bactérienne. L'antigène flagellaire H est de nature
protéique entrant dans la structure du flagelle permettant la
mobilité de la bactérie. L'antigène de surface K est
présent de façon inconstante il bloque l'agglutinabilité
de l'antigène O qui peut être restituée après
chauffage de la souche à 100°C car il est détruit par
ébullition. Les antigènes de surface aussi appelés
antigènes de capsule ou d'enveloppe sont appelés « Vi »
chez Salmonella (Avril et al. 2000).
1-6- Pouvoir Pathogène
Les entérobactéries occupent une place
très importante en pathologie humaine infectieuse. Cette importance
s'explique aussi bien par la variété des espèces
bactériennes qui les composent qu'à leur incidence au niveau de
la santé des populations. Les Enterobacteriaceae
5
Chpaitr 1 : Synthese bibliographique
forment une vaste famille de bactéries qui sont
à l'origine de maladies de gravité très variable, en
raison de mécanismes pathogéniques distincts. La fréquence
et la gravité des infections, dont elles sont responsables
(septicémies, infections nosocomiales, méningites...), traduisent
des difficultés de prise en charge liées entre autres à
des difficultés d'identification et à leur résistance aux
antibiotiques. On les rencontre dans les prélèvements d'origine
divers, mais particulièrement dans les urines et les
prélèvements sanguins qui constituent une part très
importante des activités du laboratoire de bactériologie. Les
entérobactéries constituent plus de 80% des germes isolés
en laboratoire : Escherichia, Salmonella, Shigella, Klebsiella,
Enterobacter, Serratia, Proteus, Morganella et Yersinia sont les
bactéries les plus souvent retrouvés.
Escherichia coli est un germe
très courant. Son habitat est le colon humain où il est le plus
abondant. Chez l'homme, la colonisation par E. coli est
précoce, et peut être responsable d'un nombre varié de
pathologie. Toutefois, trois types de syndromes majeurs résultent de
l'infection par de souches E. coli pathogènes :
- Infections urinaires (impliqué dans
80 % des infections urinaires), l'incidence de ces infections est plus
marquée chez les personnes de sexe féminin en milieu
extrahospitalier en raison notamment de la colonisation de la région
péri-urétrale. En milieu hospitalier, l'incidence est
égale entre les deux sexes en rapport essentiellement avec l'utilisation
fréquente des sondes urinaires.
- Infections digestives (diarrhées,
infections hépatobiliaires et autres), E. coli cause
principalement des infections du tractus digestif, en raison de la
contamination de l'eau ou des aliments par la flore fécale des malades
ou des porteurs. E. coli est subdivisé en 6 pathovars majeurs
selon le type de maladie engendrée et les facteurs de virulence
associés : les ETEC (E. coli Entérotoxinogènes),
les EHEC (E. coli Entérohémorragiques), les EPEC (E.
coli Entéropathogènes), les DAEC (E. coli à
Adhérence Diffuse), les EAEC (E. coli
Entéroagrégatives) et les EIEC (E. coli
Entéroinvasives) (Avril et al.2000).
- Méningites néonatales et
septicémies, E. coli à l'origine de maladies
extra- intestinales, a acquis la capacité de déjouer les
défenses immunitaires de l'hôte, et à se propager dans
l'organisme et il peut coloniser les voies génitales et
générer des méningites néonatales.
Klebsiella pneumoniae habite le
tractus digestif et le système respiratoire supérieur. Ce germe
est principalement isolé en milieu hospitalier, le portage étant
fortement accru chez les
6
Chpaitr 1 : Synthese bibliographique
patients hospitalisés. Toutefois, il est
également présent en dehors des hôpitaux, notamment chez
des patients diabétiques, fortement débilités, ou
souffrant de maladies respiratoires chroniques. Klebsiella pneumoniae
peut causer des pneumonies lobaires, des bronchites et broncho-pneumonies,
la contamination pulmonaire se faisant surtout par voie aérienne, mais
la voie hématogène n'étant pas exclue. Klebsiella
pneumoniae est également retrouvé dans des infections
urinaires suite au passage de la flore fécale aux voies urinaires.
Finalement, des bactériémies compliquent parfois les infections
localisées.
Klebsiella oxytoca est une
bactérie isolée dans la majorité des cas dans les selles,
mais peut aussi être isolée dans les urines, le sang et les
sécrétions naso-pharyngées et trachéales. A
l'instar de Klebsiella pneumoniae, Klebsiella oxytoca peut
infecter les voies urinaires et respiratoires des patients
hospitalisés.
Salmonella constitue un vaste groupe
de bactéries, composé de plus de 2000 variétés, et
subdivisé en deux sous-groupes. Le premier comprend les Salmonella
dites majeures qui causent les fièvres typhoïdes et
paratyphoïdes dues à la transmission féco-orale, l'eau ou la
nourriture étant contaminées par les selles d'un malade ou d'un
porteur sain.
Le second est composé des Salmonella dites
mineures qui sont à l'origine d'intoxications alimentaires (aliments
provenant d'animaux infectés ou contaminés ; viande, produits
laitiers...), de gastro-entérites et de bactériémies.
Enterobacter cloacae est un germe
colonisant souvent les patients hospitalisés et plus
particulièrement ceux traités par antibiotiques. Il a
été associé à des épidémies
nosocomiales et est considéré comme pathogène opportuniste
(Hart. 2006). Il peut causer de nombreux types d'infections, y compris
abcès cérébraux, pneumonie, méningite,
septicémie et infection de plaies, infection des voies urinaires et des
infections de la cavité abdominale ou des intestins (Farmer et
al. 2007). De plus, il a été observé dans des
infections liées à des appareils intra-vasculaires et des
infections au point de chirurgie (surtout des infections postopératoires
ou liées à des dispositifs comme des prothèses
biliaires).
Serratia marcescens colonise les
systèmes respiratoires, digestifs et urinaires des patients. Bien que
les germes isolés en milieu hospitalier soient le plus souvent
responsables d'une colonisation asymptomatique, quelques infections
nosocomiales sont relatées dans la
7
Chpaitr 1 : Synthese bibliographique
littérature: ce sont essentiellement des
bactériémies, des infections des voies respiratoires
inférieures, des infections urinaires et cutanées. Ces infections
sont dues à des germes qui proviennent des patients eux-mêmes ou
de leur environnement (Mirabaud. 2003).
Citrobacter freundii est un agent
pathogène nosocomial opportuniste rare, qui entraîne normalement
des infections des voies urinaires, des bactériémies, des sepsis
abdominaux et des abcès cérébraux ainsi que des pneumonies
et d'autres infections néonatales (Pepperell et al. 2002).
2- Les â-lactamines
Les f3-lactamines demeurent les antibiotiques les plus
utilisés dans la pratique clinique courante. Cette large utilisation est
principalement liée à leur faible toxicité, à leur
pouvoir bactéricide et à la diversité des molécules
(Robin et al. 2012).
L'histoire des B-lactamines débute depuis
l'introduction de la pénicilline G en thérapeutique dans les
années 1940.Il faudra attendre le début des années 1960
pour voir apparaître les premières synthèses de
B-lactamines permettant leur développement à l'échelle
industrielle. Un grand nombre de molécules incluant les
pénicillines, les céphalosporines, les monobactames et les
carbapénémes a été développé.
Cependant, la grande utilisation des B-lactamines depuis plus de 60 ans s'est
accompagnée d'une augmentation importante de la résistance
bactérienne à ces antibiotiques. D'autre part, le
développement de nouveaux antibiotiques connaît un franc
ralentissement depuis plus de 10 ans (Robin et al. 2012).
Cette famille est caractérisée par la
présence constante de cycle f3-lactame associé à des
cycles et des chaînes latérales variables expliquant les
propriétés pharmacocinétiques et le spectre
d'activité des différents produits (Cavallo et al.
2004).
2-1- Classification des â-lactamines
Quatre groupes de cette classe d'antibiotique peuvent
être décrits et ce en fonction de leurs caractéristiques
structurales : les pénames (pénicillines), les
céphèmes (céphalosporines), les pénèmes et
les monobactames (Fig.1).

Chpaitr 1 : Synthese bibliographique
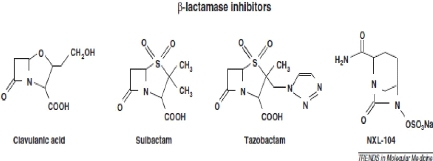
8
Figure 1. Structures chimiques des principales
f3-lactamines et inhibiteurs de f3-lactamases
(Nordmann et al.
2012)
2-1-1- Les pénicillines (noyau
péname)
La formule générale des pénicillines associe
un noyau B-lactame à un cycle thiazolidine et une chaîne
latérale en C-6 (Fig.1). Selon la nature de la chaîne
latérale reliée à cette molécule, on distingue
plusieurs sous-classes de pénicillines(Tab.1) (Gazengel. 2007).
2-1-2- Les céphalosporines (noyau
céphème)
Les céphalosporines se distinguent chimiquement des
pénicillines par le remplacement du cycle thiazolidine par un cycle
dihydrothiazine (noyau « céphème ») avec un atome de
soufre
9
Chpaitr 1 : Synthese bibliographique
en position 1 (Fig.1). Ces composés sont obtenus par
synthèse ou semi-synthèse, à partir de la
céphalosporine C, qui est un produit de fermentation d'un champignon
(Cephalosporium acremonium), grand producteur de l'acide
amino-7-céphalosporanique (noyau de base) (Gazengel. 2007). On distingue
cinq générations de céphalosporines (Tab.1). Elles sont
classées en fonction de leur date d'apparition, qui correspond à
chaque fois à l'acquisition de nouvelles propriétés :
? Les céphalosporines de 1ère
génération (C1G)
Ont un niveau d'activité assez limité
vis-à-vis des bacilles à Gram (-), en raison de leur
sensibilité aux f3-lactamases (Nauciel et Vildé. 2005).
? Les céphalosporines de 2éme
génération (G)
Caractérisées par une meilleure
résistance aux B-lactamases et un spectre d'action plus large. Elles
possèdent un pouvoir antibactérien un peu plus
élevé (CMI plus basses) et un spectre plus large que le
1er groupe vers les entérobactéries (Gazengel.
2007).
? Les céphalosporines de 3éme
génération (C3G)
L'ensemble de leurs propriétés
antibactériennes et pharmacocinétiques autorise des posologies
réduites pour une efficacité accrue. Les C3G sont inactives,
à des degrés divers par les f3-lactamases à spectre
élargi (Nauciel et Vildé. 2005).
? Les céphalosporines de 4éme
génération (C4G)
Représentées par Céfépime et
Céfpirome, elles restent actives chez les entérobactéries
ayant acquis une résistance aux C3G par hyperproduction d'une
céphalosporinase, et inactives en cas de f3-lactamase à spectre
étendu. Elles sont une substitution possible aux céphalosporines
de troisième génération pour le traitement de germes
résistants (Hardman et Limbird. 1998). ? Les
céphalosporines de 5éme génération
(C5G)
Représentées par la Ceftaroline, Ceftolozane et
Ceftobiprole. Ces antibiotiques sont à usage hospitalier. La Ceftaroline
a un large spectre d'activité contre de nombreuses bactéries
à Gram négatif (Kenneth et al. 2017).
10
Chpaitr 1 : Synthese bibliographique

Figure 2. Structure chimique de la
ceftaroline (Jacquline et Tattevin. 2016). 2-1-3- Les
pénèmes (Carbapénèmes)
Les Carbapénèmes sont des f3-lactamines
présentant un très large spectre d'activité et une grande
stabilité vis à vis de la plupart des ß-lactamases.
L'imipenème et le méropeneme ont été les deux
premiers antibiotiques disponibles en clinique. Une troisième
molécule s'est ajoutée, l'ertapénème. Parmi les
nouvelles Carbapénèmes, le doripénème garde une
meilleure activité sur les bacilles à Gram négatif. Les
Carbapénèmes sont historiquement considérées comme
le traitement de choix des infections sévères à
bactéries Gram négatives (Grall et Muller-Serieys. 2013).
2-1-4- Les monobactames
L'aztréonam fut le premier monobactame fabriqué
synthétiquement, il est réservé au traitement
parentéral des infections à bactéries Gram (-). Son
spectre d'activité est voisin de celui des C3G (bonne stabilité
vis-à-vis des f3-lactamases) (Gazengel. 2007).
2-1-5- Les inhibiteurs des â-lactamases
Ces substances n'ont pas une grande activité
bactéricide mais ils sont capables de protéger les f3-lactamines
en inhibant les pénicillinases. Ce groupe est composé de :
? L'acide clavulanique, utilisé en
association fixe avec l'amoxicilline (Augmentin®) ou la ticarcilline
(Claventin®).
? Le sulbactam (Bétamaze®),
éventuellement en association avec l'ampicilline (sulbactam 500 mg +
ampicilline 1 g (Unacim®).
? Le tazobactam associé à la
pipéracilline (Tazocilline®) (Gazengel. 2007).
? L'avibactam, utilisé en association
avec ceftazidime, ceftaroline et aztreonam. L'Avibactam (NXL104) est un
non-f3-lactame inhibiteur de BLSE avec activité contre la plupart des
classe A et classe C f3-lactamases ainsi que certaines enzymes de classe D
Chpaitr 1 : Synthese bibliographique
(Buyck et al. 2016). Actuellement, l'avibactam est en
développement dans de nombreux essais cliniques.
Tableau 1. Classification des
â-lactamines.
Noyau Sous-famille Molécules
Pénames Pénicillines G et V
pénicilline G
pénicilline V
Pénicillines A
ampicilline
amoxicilline
pivmecillinam
Pénicillines M
oxacilline , cloxacilline
Carboxypénicillines
Uréidopénicillines
Inhibiteurs de
bêta-
lactamases
ticarcilline, temocilline
mezlocilline,
acide clavulanique (+ amoxicilline +
ticarcilline)
sulbactam (seul ou + ampicilline)
tazobactam (+
pipéracilline)
avibactam (+ ceftazidime)
Pénémes Carbapénèmes
imipéneme
méropéneme ertapéneme
doripénéme
Monobactames Monobactame aztréonam
|
Céphèmes C1G (1ère
génération)
|
céfalotine, céfalexine, céfapirine,
céfazoline,
|
céfadroxil, céfaclor, céfatrizine,
céfradine
|
G (2ème génération)
C3G (3ème génération)
|
céfamandole, céfuroxime, céfoxitine
céfotétan
céfotaxime, ceftizoxime,
céfopérazone,
ceftriaxone, ceftazidime, cefsulodine
|
11
12
Chpaitr 1 : Synthese bibliographique
céfixime, cefpodoxime proxétyl, céfotiam
hexétil
|
C4G (4ème génération) C5G (5ème
génération)
|
céfépime, cefpirome
ceftaroline, ceftobiprole,
ceftolozane
|
2-2- Mode d'action des â-lactamines
Les f3-lactamines ciblent les protéines de la membrane
cytoplasmique qu'on appelle les PLP (protéines de liaison aux
pénicillines) ou PBP (Penicillin Binding Proteins). Ces PLP sont en
effet les enzymes impliqués dans la synthèse du peptidoglycane
(les transpeptidases, les carboxypeptidases et les transglycosylases). Les
f3-lactamines présentent une analogie structurale avec un constituant du
peptidoglycane en formation, le dipeptide D-ala-D-ala qui est le substrat
naturel de ces enzymes. Elles agissent en « substrat suicide » en
bloquant le fonctionnement de ces enzymes inhibant ainsi la formation du
peptidoglycane. Par conséquent, cette inhibition entraîne un
arrêt de la croissance bactérienne (bactériostase). Le
blocage de la multiplication est suivi d'une lyse bactérienne. Le
peptidoglycane est dégradé sous l'action d'autolysines, ce qui
entraîne finalement la lyse (bactéricide).
Le nombre et la nature des PLP varient selon les
espèces bactériennes, donc l'affinité des divers PLP n'est
pas la même pour toutes les molécules des f3-lactamines. L'effet
d'une f3-lactamine est en fonction de son affinité pour les
différentes PLP de la bactérie considérée (Gaudy et
Buxeraud. 2005).
3- Résistance des entérobactéries
aux â-lactamines
La résistance bactérienne aux antibiotiques est
un véritable problème de santé publique. Depuis
l'utilisation des antibiotiques, la résistance n'a jamais cessé
d'augmenter et les bactéries ont toujours réussi à
contourner les antibiotiques destinés à les tuer. Jusqu'à
maintenant, cette évolution était compensée par la mise
sur le marché de nouveaux antibiotiques, mais celle-ci s'est
considérablement amenuisée laissant le risque de voir
apparaître une aire dite « post-antibiotique »
(Armand-Lefévre. 2017).
13
Chpaitr 1 : Synthese bibliographique
3-1- Types de résistance
La bactérie est considérée
résistante lorsqu'elle supporte une concentration d'antibiotique
nettement plus élevée que celle qui inhibe la multiplication de
la majorité des autres bactéries de la même espèce
(Boulahbal. 2010) ou de la tuer (Poole. 2004 ; Schwarz et Chaslus-Dancla.
2001). Les conditions d'activité d'un antibiotique sont de
posséder une cible spécifique, de demeurer sous forme active,
d'accéder à la cible et d'interagir efficacement avec elle en la
désactivant.
Les entérobactéries sont soit sensibles aux
â-lactamines, soit naturellement résistantes ou soit ont une
résistance acquise (Vora et Auckenthaler. 2009).
3-1-1- La résistance naturelle
La résistance naturelle est programmée sur le
génome et constante à l'intérieur du taxon ; elle
constitue un critère d'identification stable d'une espèce. La
résistance naturelle ou intrinsèque à un antibiotique est
essentiellement due à la présence de gènes chromosomiques;
elle est donc commune à toutes les bactéries d'une même
espèce et transmise à la descendance. Dans tous les cas, la
résistance naturelle fait partie des caractères normaux de
l'espèce ; elle détermine le niveau de sensibilité «
basal » des bactéries et définit le phénotype sauvage
d'une espèce. Elle est constitutive et touche toute une famille
d'antibiotiques (Mayer et al. 2000). Ainsi, les
entérobactéries ont une résistance naturelle à la
pénicilline G, V et les pénicillines du groupe M (Philippon et
Arlet. 2012).
3-1-2- La résistance acquise
Ce terme est utilisé pour désigner le
résultat d'un processus permettant à des bactéries d'une
espèce originellement sensible de devenir résistante à un
ou plusieurs antibiotiques. L'acquisition de ces résistances est
déterminée par des modifications génétiques
consécutives à des mutations ponctuelles (plutôt rare) ou
à l'acquisition de nouveaux gènes de résistance
exogènes (plutôt fréquent) par le biais de plasmides, de
bactériophages, de transposons ou d'intégrons (Davies. 1997 ;
Chopra et al. 2003).
La capacité de multiplication très rapide des
bactéries favorise la sélection d'évènements
génétiques favorables et la possibilité d'échange
d'information même entre espèces lointaines leur conférant
un très grand pouvoir d'adaptation aux contraintes du milieu.
L'évolution des
14
Chpaitr 1 : Synthese bibliographique
mécanismes de résistance aux antibiotiques, et
notamment aux â-lactamines illustre parfaitement ce
phénomène.
3-2- Supports génétiques de la
résistance
Parmi les modalités d'acquisition de la
résistance aux antibiotiques, le transfert horizontal de gènes
est un élément-clé. Ce type de transfert a probablement
lieu dans tous les écosystèmes terrestres colonisés par
les bactéries. Ainsi ces transferts ont été mis en
évidence dans de nombreux écosystèmes, tels les sols, les
rivières, les environnements marins, mais aussi les tubes digestifs
d'insectes ou de mammifères. Différents éléments
génétiques sont impliqués dans ce transfert de
gènes.
3-2-1- Le chromosome
La résistance chromosomique acquise résulte d'une
mutation. La fréquence de ces mutations est faible et variable
(10-6 à 10-9) mais la mutation est stable et
transmissible à la descendance. La résistance apparaît dans
ce cas au hasard et n'est donc pas influencée par l'antibiotique quine
fait que la révéler.
3-2-2- Les éléments
génétiques mobiles
La résistance aux antibiotiques acquise par les
bactéries est principalement due à la présence de trois
types d'éléments extra-chromosomiques portant des gènes de
résistance : les plasmides, les transposons et des cassettes de
résistances insérées sur un intégrons (Bennett.
2008 ; Martinez. 2010 ; Walsh. 2006). Ce type de résistance est
transmissible d'une souche ou d'une espèce à une autre selon
trois modes de transmission, à savoir la conjugaison (transfert direct
entre deux bactéries ayant établi temporairement un contact
physique), la transformation (transfert d'ADN nu) et la transduction (transport
d'ADN bactérien par des bactériophages).
15
Chpaitr 1 : Synthese bibliographique
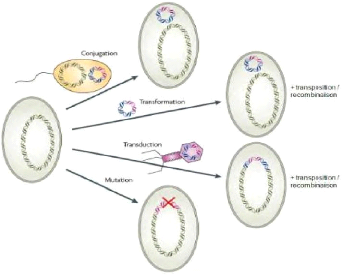
Figure 3. Modes de transmission du
matériel génétique : la conjugaison, la transformation et
la transduction chez les bactéries (Andersson et Hughes. 2010).
a) Plasmides
Les plasmides bactériens revêtent une importance
toute particulière dans l'étude des phénomènes de
résistance car ils constituent à la fois un vecteur de premier
plan pour la dissémination des résistances et un immense
réservoir génétique. Comme les chromosomes, les plasmides
codent pour des protéines et des molécules d'ARN et se
répliquent durant la croissance bactérienne. Les plasmides
partagent certaines fonctions de partition et quelques recombinasses
spécifiques de site avec le chromosome de l'hôte (Snyder et
Champness. 2007). Contrairement aux chromosomes, les plasmides ne portent
toutefois pas de gènes essentiels à la croissance
bactérienne. Ils codent plutôt pour des protéines dont la
fonction est de conférer un avantage sélectif comme la
résistance aux antibiotiques ou encore des facteurs de virulence.
Les plasmides peuvent conférer une résistance
aux principales classes d'antibiotiques (Carattoli. 2009). Une même
bactérie peut porter un ou plusieurs plasmides selon leur
compatibilité mutuelle et chacun d'eux peut contenir un ou plusieurs
gènes de résistance (Harbottle et al. 2006).Les
plasmides peuvent également se transmettre à une souche sensible
principalement par conjugaison mais aussi par transformation ou transduction
à l'aide de bactériophages. Les plasmides, en fonction de leur
efficacité de conjugaison, vont permettre le
16
Chpaitr 1 : Synthese bibliographique
transfert horizontal de ces composantes à d'autres
bactéries qui peuvent être de différentes espèces,
genres et familles (Thomas et Nielsen. 2005).Ils peuvent acquérir
d'autres gènes de résistance par l'intermédiaire
d'éléments transposables et d'intégrons par exemple. La
survenue épidémique de bactéries à Gram
négatif multirésistantes est largement attribuée à
ce type de transmission.
b) Séquences d'insertion et
transposons
Ces éléments transposables jouent un rôle
très important dans l'évolution des bactéries puisqu'ils
sont à l'origine de multiples réarrangements de leur
génome. Les transposons (2-20 kpb) sont composés de
séquences d'ADN qui fonctionnent comme des sites de recombinaison et de
gènes codants pour des protéines participant à la
réaction de recombinaison. Les sites de recombinaison sont situés
aux deux extrémités du transposon et organisés en
séquences inversées répétées. Les
éléments transposables ont la capacité de se
déplacer et de s'insérer à différents endroits dans
un chromosome ou un plasmide par un processus de transposition anarchique. Il
existe différentes types d'éléments transposables ; les
séquences d'insertion (SI) étant de courtes séquences
génétiques de 800 à 2000 pb, elles codent uniquement pour
la transposition et les transposons composites codent pour les
déterminants de la transposition ainsi que pour d'autres fonctions,
notamment celle de conférer la résistance aux antibiotiques (El
Salabi et al. 2013).
c) Les intégrons
Les intégrons, plus récemment décrits
vers la fin des années 80, jouent un rôle majeur dans
l'acquisition et l'expression de gènes de résistance aux
antibiotiques, notamment chez les bactéries à Gram
négatif. Le rôle d'une partie de ces intégrons, parfois
nommés intégrons de résistance (IR), est majeur dans la
dissémination de la résistance aux antibiotiques et notamment
chez des souches d'intérêt clinique. L'ampleur de la diffusion de
la résistance est attribuable en majeure partie au déplacement
des gènes de résistance par le biais d'intégrons
(Domingues et al. 2012).Ceux-ci sont des éléments
génétiques mobiles avec une structure spécifique
composée de deux segments conservés encadrant une région
centrale appelée «cassette » (Domingues et al. 2012 ;
Recchla et Hall. 1997). La transposition des intégrons n'est pas
anarchique et a lieu au niveau de sites spécifiques, celle-ci peut
recevoir, a différents moment des gènes variés dont ceux
de la résistance aux antibiotiques qui vont s'insérer toujours au
même endroit « site d'insertion » ce qui provoque l'allongement
de l'intégrons
17
Chpaitr 1 : Synthese bibliographique
dans un sens. Les intégrons peuvent être
associés à des (SI), des transposons ou des plasmides pouvant
leur conférer une mobilité (Domingues et al. 2012).
3-3- Mécanismes de résistance aux
â-lactamines
Les différents mécanismes de résistance
aux f3-lactamines peuvent s'exprimer seuls ou de façon concomitante
agissant alors de façon synergique. Il peut s'agir d'une diminution
d'affinité de la cible, d'une diminution de la
perméabilité de la membrane, de l'expression d'une pompe d'efflux
actif ou de la production d'enzyme de type f3-lactamase (Vora et Auckenthaler.
2009).
3-3-1- Diminution de la
perméabilité
La pénétration des f3-lactamines, à
travers la membrane externe s'effectue à travers les porines qui sont
des canaux protéiques remplis d'eau. Ainsi, la sensibilité aux
f3-lactamines dépend du nombre de porines fonctionnelles.
L'altération des porines par mutation est à l'origine de
résistances acquises aux f3-lactamines, soit par une modification
structurale d'une porine essentielle, ce qui a été décrit
chez E. coli, soit par une diminution quantitative des porines, qui
est la situation la plus fréquente (Kumar et Schweizer. 2005).
3-3-2- Modification de la cible
La résistance aux f3-lactamines, conférée
par les PLPs, chez les bactéries à Gram négatif joue un
rôle mineur dans la résistance (Spratt et al. 1989).
Cette résistance peut avoir lieu par des mutations dans les gènes
chromosomiques codant pour les PLPs ou par l'acquisition de gènes
étrangers codant pour de nouveaux PLPs ayant une affinité
différente aux f3-lactamines (Georgopapadakou. 1993). Une diminution de
la production de la PLP1A a été associée à la
résistance pour l'imipénème et au mécillinam chez
P. mirabilis suite à une perte d'affinité de la PLP2 et
à une diminution de la quantité de PLP1 (Neuwirth et al.
1995).Ces mutations chez les entérobactéries restent
rares (Robin et al. 2012).
3-3-3- Hyperproduction du système
d'efflux
Les systèmes d'efflux éliminent activement les
f3-lactamines dans le milieu extérieur en utilisant la force
proton-motrice. Ainsi, ils diminuent la concentration d'antibiotique au contact
de sa cible et donc son efficacité. Le système d'efflux actif est
efficace grâce aux
Chpaitr 1 : Synthese bibliographique
protéines transmembranaires ancrées dans la
membrane cytoplasmique mais également dans la membrane externe des
bactéries à Gram négatif (Walsh. 2003).
L'implication des systèmes d'efflux dans la
résistance aux f3-lactamines a été clairement
identifiée dans plusieurs études en particulier chez K.
pneumoniae. Cependant ce type de mécanisme touchant
préférentiellement la céfoxitine et les G semble difficile
à distinguer du point de vue phénotypique des résistances
par modification des porines (Bialek-Davenet et al. 2011 ; Robin et
al. 2012).
3-3-4- Production d'enzymes
La production des f3-lactamases est le principal
mécanisme de résistance des entérobactéries aux
f3-lactamines (Abbas et al. 2012).Cependant, toutes les
entérobactéries, quel que soit leur groupe, sont capables
d'intégrer des gènes de résistance codant pour une
f3-lactamase (Zoghleb et Dupont. 2005).
4- Les â-lactamases
C'est le mécanisme qui nous intéresse le plus
dans notre étude. Les f3-lactamases sont des enzymes bactériennes
hydrolysant la fonction amine du cycle f3-lactame et rendant l'antibiotique
inactif avant qu'il n'atteigne sa cible : les PLP. Les f3-lactamases sont des
enzymes constitutionnelles ou acquises, produites par les bactéries.
Leur activité enzymatique provoque l'ouverture du cycle f3-lactame et
crée un intermédiaire acyl-enzyme instable qui est ensuite
dégradé en acide inactif (Vodovara et al. 2013).Les
gènes codant pour ces enzymes sont principalement situés sur des
éléments génétiques mobiles (plasmides,
transposons, intégrons) (Boyer et al. 2011).
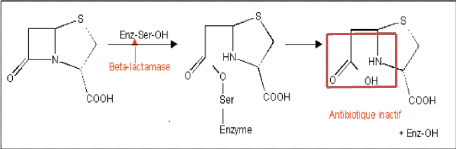
18
Figure 4. Mode d'action des f3-lactamases
(Barrial et Scotet. 2006).
19
Chpaitr 1 : Synthese bibliographique
4-1- Classification des â-lactamases
Les deux classifications couramment utilisées sont
celle d'Ambler et de Bush, Jacoby et Medeiros. La classification structurale
d'Ambler est basée sur la séquence peptidique du site enzymatique
et distingue quatre classes, alors que la classification fonctionnelle de Bush
et ses collaborateurs repose sur l'activité hydrolytique et la
sensibilité des f3-lactamases aux inhibiteurs. Elle rend compte de leur
diversité fonctionnelle au sein des quatre classes structurales
d'Ambler, notamment dans sa classe A (Vodovara et al.
2013).Aujourd'hui, les classifications d'Ambler et de Bush-Jacoby-Medeiros sont
considérées comme étant les plus pertinentes (Bush et
al. 1995).
4-1-1- Classification d'Ambler
? Les â-lactamases de classe A
Les f3-lactamases de classe A, d'origine chromosomique ou
plasmidique, se caractérisent par leur capacité à
hydrolyser l'amide cyclique lié à la molécule de
f3-lactame. Cette activité hydrolytique, assurée par une
sérine conservée (Ser-70)- induit la formation d'acide
pénicilloïque pour la pénicilline et de
céphalosporoate, analogue aux pénicillinoates, pour les
céphalosporines. Cette sérine est retrouvée dans le site
actif de la f3-lactamase et assure un fort taux de résistance aux
pénicillines, céphalosporines et carbénicillines.
Cependant, les f3-lactamases de classe A sont sensibles aux inhibiteurs de
f3-lactamases (Livermore. 1995).
? Les â-lactamases de la classe B
Les f3-lactamases de classe B sont des
métallo-f3-lactamases et utilisent un ion de zinc (Zn2+) comme
cofacteur permettant ainsi la décomposition de l'anneau f3-lactame
(Ambler. 1980 ; Bandoh et al. 1991). Ces enzymes ont
émergés depuis une dizaine d'années, d'abord chez
Pseudomonas aeruginosa et Acinetobacter baumannii, puis chez
les entérobactéries et d'autres bacilles à Gram
négatif. L'importance clinique des métallo-bêta-lactamases
est liée au fait qu'elles hydrolysent les carbapenèmes,
composés qui échappent à l'activité des
f3-lactamases à sérine active. La plupart des
métallo-f3-lactamases hydrolysent une variété de
pénicillines et de céphalosporines, et sont insensibles aux
inhibiteurs suicides classiques (Bebrone. 2007).
20
Chpaitr 1 : Synthese bibliographique
? Les â-lactamases de la classe C
Les f3-lactamases de classe C, décrites en 1981 par
Jaurin et Grundstrom, ont longtemps été ubiquitaires aux
chromosomes de différentes espèces bactériennes
entériques (Jaurin et Grundstrom. 1981).Dans la classe C, on retrouve
les céphalosporinases AmpC qui sont codées par des gènes
qui étaient primitivement situés sur le chromosome de nombreuses
bactéries à Gram négatif telles que Citrobacter
freundii, Serratia marcescens et Enterobacter spp. Chez
ces bactéries, l'expression d'AmpC est inductible, les gènes
codant pour ces enzymes sont aussi présents chez E. coli,
où ils ne sont pas inductibles. Ces enzymes sont résistantes
à l'acide clavulanique (Philippon et al. 2002).
? Les â-lactamases de la classe D
Les f3-lactamases de classe D se distinguent par leur
capacité à hydrolyser les pénicillines isoxazolyl
(oxacilline) et la méthicilline, et sont faiblement inhibées par
l'acide clavulanique. Elles sont retrouvées fréquemment chez
Pseudomonas aeruginosa et Acinetobacter spp. (OXA-11, 14, 16,
17 et 19) et peu chez les enterobactéries (Paterson et Bonomo. 2005).De
plus, certains membres de cette famille, dont OXA-10 (PSE-2) et OXA-11 (Hall et
al. 1993), possèdent un large spectre d'activité
assurant une forte résistance aux céphalosporines de
troisième génération dont la céfotaxime, le
céfoperazone et la ceftazidime (Hall et al. 1993 ; Livermore.
1995).
4-1-2- Classification de
Bush-Jacoby-Medeiros
Elle est établie selon les propriétés
fonctionnelles de l'enzyme définies par son substrat
préférentiel et son profil d'hydrolyse (Bush et al.
1995). Ces auteurs divisent ces enzymes en quatre groupes (1 à 4) avec
plusieurs sous-groupes. Les bêtalactamases de la classe A sont ainsi
subdivisées en 8 groupes fonctionnels 2a, 2b, 2be, 2br,2ber, 2c, 2e et
2f (Bush et Jacoby. 2010).
Cette classification phénotypique de f3-lactamases pose
un certain problème puisque différentes mutations ponctuelles
peuvent influencer leur susceptibilité à ces substrats et
inhibiteurs (Jacoby et Medeiros. 1991). Un autre aspect restrictif de cette
classification relève du fait que l'attribution d'un acronyme à
un gène de résistance n'a plus la même signification. Par
exemple, les f3- lactamases PSE, ou "Pseudomonas Specific Enzymes",
ont aussi été identifiées chez les Enterobacteriaceae, ce
qui remet en question la pertinence de ce préfixe (Matthew et
al. 1979).
21
Chpaitr 1 : Synthese bibliographique
Tableau 2. Classification global des BLSE (Bush
et al. 2010).

22
Chpaitr 1 : Synthese bibliographique
5- Les â-lactamases à spectre élargi
BLSE
Les BLSE ont été décrites pour la
première fois en 1983, en Allemagne (Knothe et al. 1983), il
n'y a pas de consensus concernant la définition de f3-lactamases
à spectre étendu (BLSE). Classiquement, les BLSE sont
définies comme des enzymes appartenant à la classe A (à
l'exception des BLSE de type OXA classe D de la classification d'Ambler),
capables d'hydrolyser les pénicillines, céphalosporines de
première, deuxième, troisième et quatrième
génération (céfépime ou cefpirome) et
l'aztréonam. Elles sont inhibées in vitro par les
inhibiteurs des f3-lactamases (acide clavulanique, tazobactam et sulbactam)
(Livermore. 1995). Par contre, les BLSE sont sensibles aux céphamycines
(céfotétan et cefoxitine) ainsi qu'aux
carbapénèmes. Une co-résistance avec les aminosides, les
tétracyclines et les fluoroquinolones est fréquente. Les
bactéries possédant des BLSE sont dites multirésistantes
(Paterson et Bonomo. 2005).
Il s'agit d'un mécanisme de résistance de type
plasmidique, et donc transmissible à d'autres bactéries. La
présence de ce type de mécanisme de résistance au sein de
souches pathogènes fait peser un risque majeur d'inadéquation
thérapeutique et donc d'échec thérapeutique (Schwaber et
Carmeli. 2007), et est également un facteur de diffusion.
Au sein des entérobactéries, Klebsiella
pneumoniae et Escherichia coli sont les deux espèces les
plus fréquemment porteuses de ces mécanismes de
résistance. Toutefois, ces enzymes ont été
retrouvées au sein de nombreuses autres espèces
bactériennes, entérobactéries et bacilles non fermentant
(tels que Pseudomonas aeruginosa et Acinetobacter baumannii)
(Jacoby et Munoz-Price. 2005).
5-1- Différents types de BLSE
Elles sont classées selon leurs types
moléculaires, les plus fréquents étant les types TEM, SHV,
CTX-M (Jacoby et Munoz-Price. 2005) et les nouvelles étant les
carbapénèmases.
5-1-1- BLSE de type TEM (Temoneira - nom du
patient)
La première f3-lactamase plasmidique de type TEM
(TEM-1) a été isolée en 1965, en Grèce, à
partir d'une souche d'E. coli isolée chez une patiente
nommée Temoneira, d'où la nomination (Datta et
Kontomichalou. 1965). La majorité des BLSE de ce type dérivent
par quatre à sept mutations ponctuelles de l'enzyme originale (TEM-1 ou
TEM-2). Les substitutions les plus courantes sont le glutamate en lysine en
position 104, l'arginine en
Chpaitr 1 : Synthese bibliographique
sérine en position 164, la glycine en sérine en
position 238 et le glutamate en lysine en position 240 (Bradford. 2001). Une
seule de ces substitutions peut entraîner une importante modification de
l'affinité de l'enzyme. Par exemple, lorsque l'acide aminé
devient une sérine, l'interaction entre le -NH3 de la sérine et
le groupement oxy-amino des C3G se traduit par une meilleure stabilité
de la â-lactamine dans le site actif de l'enzyme. Ces mutations rendent
l'enzyme capable d'hydrolyser les C3G, mais aussi plus vulnérable
à l'action des inhibiteurs (acide clavulanique).
Cependant, d'autres mutations peuvent conférer la
résistance aux inhibiteurs. Ces variantes sont appelées TRI (TEM
résistantes aux inhibiteurs). Les enzymes dérivées par
mutations permettant d'hydrolyser à la fois les C3G et les inhibiteurs
sont de plus en plus fréquentes (Rodriguez-Villalobos et Struelens.
2006).

Figure 5.Les BLSE dérivées de TEM
(Ambler et al. 1991).
23
24
Chpaitr 1 : Synthese bibliographique
5-1-2- BLSE de type SHV : (Sulfhydryl
variable)
Les enzymes BLSE de type SHV dérivent par mutations
ponctuelles de l'enzyme originale SHV-1 qui correspond a un gène
blaSHV de pénicillinase chromosomique de K. pneumoniae
(Brisse et Verhoef. 2001 ; Haeggman et al. 2004), et cette
désignation SHV est liée au sulfhydryl variable. La
majorité des BLSE de type SHV est caractérisée par la
substitution d'acides aminés Gly238Ser ou Gly238Ser et Glu240Lys. Le
résidu sérine à la position 238 est indispensable pour
l'hydrolyse efficace du céfotaxime et le résidu lysine est
crucial pour l'hydrolyse efficace de la ceftazidime (Elhani. 2012).
La majorité des BLSE de type SHV ont été
décrite chez les souches de K.pneumoniae, toutefois ces enzymes
ont été trouvées chez C. freundii, C. diversus, E.
coli, E. cloacae (Bradford. 2001).La présence de la séquence
d'insertion IS26 sur le gène SHV faciliterait l'acquisition du
phénotype BLSE (Hammond et al. 2005).
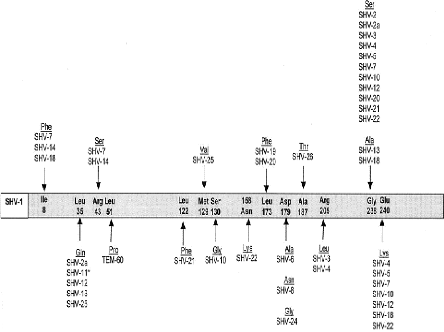
Figure 6.Les BLSE dérivées de SHV
(Bradford. 1999).
25
Chpaitr 1 : Synthese bibliographique
5-1-3- BLSE de type CTX-M
(Cefotaximase-Munich)
Les BLSE de type CTX-M ont été décrites
initialement en 1986 (FEC-1) au Japon, en Allemagne et en France en 1989
(CTXM-1) et ont depuis lors disséminé largement dans le monde
(Thomson et Moland. 2000). Ces enzymes représentent à l'heure
actuelle les BLSE les plus fréquentes au niveau mondial après
leur diffusion rapide depuis les années 90 (Bonnet. 2004 ; Livermore et
al. 2007).
Le groupe CTX-M (pour céfotaximase) conférait
à l'origine, chez les entérobactéries, un plus haut niveau
de résistance au céfotaxime (ou ceftriaxone),
céfépime et aztréonam qu'à la ceftazidime (Arlet et
Philippon. 2003 ; Bonnet. 2004). Certaines d'entre elles ont
évolué plus récemment par mutation (ponctuelle ou non)
générant un haut niveau de résistance à la
ceftazidime telles les enzymes CTX-M-15, CTX-M-16, CTX-M-19, CTX-M-23 et
CTX-M-32 (Bonnet. 2004). Les CTX-M sont plus fortement inhibées par le
tazobactam que par l'acide clavulanique.
Ces nouvelles BLSE ne sont pas étroitement liées
aux â-lactamases de type TEM ou SHV puisqu'elles ne présentent que
40 % d'homologie avec ces BLSE classiques (Elhani. 2012). Les analyses
génétiques ont montré que les gènes
progéniteurs appartiennent au genre Kluyvera,
entérobactéries d'isolement très rare en
bactériologie médicale (Bonnet. 2004 ; Humeniuk et al.
2002). La â-lactamase naturelle de Kluyvera cryocrescens
(KLUC-1) présente 95 à 100 % d'identité avec les
enzymes plasmidique du phylum CTX-M-1 (Decousser et al. 2001).
5-1-4- Autres types de BLSE
D'autres BLSE ont une distribution moins large,
caractérisées par un haut niveau de résistance à la
ceftazidime et parfois à l'aztréonam plutôt qu'au
céfotaxime (Arlet et Philippon. 2003 ; Bradford. 2001), qui sont
individualisées en BES-1 (brazilian extended spectrum), GES-1
(Guyana extended spectrum), PER-1 (Pseudomonas extended
resistance) (Weldhagen et al. 2003), SFO-1 (Serratia
fonticola), TLA-1 (Tlahuicas, tribu mexicaine), et VEB-1 (Vietnam
extended spectrum). Des enzymes proches de GES-1 ont été
découvertes en Grèce, malheureusement dénommées
à tort IBC (integron borne cephalosporinase) (IBC-1, IBC-2)
(Philippon et Arlet. 2006). Enfin, l'OXA-1 qui a une grande activité
catalytique pour la cloxacilline, l'oxacilline et la méticilline.
26
Chpaitr 1 : Synthese bibliographique
5-1-5- Les Carbapénèmases
Les Carbapénèmases décrites chez les
entérobactéries appartiennent aux quatre classes connues de
f3-lactamases (classe A, B, C, D de la classification d'Ambler) (Queenan et
Bush, 2007 ; Poirel et al. 2007). Les plus importantes actuellement en
clinique sont les KPC, MBL et OXA-48. L'activité des
Carbapénèmases de classe A est inhibé in vitro
totalement ou partiellement par les inhibiteurs des f3-lactamases (acide
clavulanique et tazobactam), alors que les autres carbapénèmases
sont insensibles à ces inhibiteurs classiques de f3-lactamases (Nordmann
et Carrer. 2010).
a) Classification des carbapénèmases ? Les
Carbapénèmases de classe A
Les carbapénèmases de classe A ont
été tout d'abord rapportées dans plusieurs souches
d'entérobactéries isolées de l'environnement
(Serratia, Enterobacter), produisant des
bêta-lactamases dont l'activité était inhibée par
l'acide clavulanique. Elles hydrolysaient, à divers degrés,
toutes les bêtalactamines. Leurs gènes sont chromosomiques ou
plasmidique. Il s'agissait des bêtalactamases NmcA, Sme-1, Sme-2/Sme-3,
IMI-1/IMI-2, ou SFC-1. D'autres carbapénémases, de type GES, ont
été identifiées chez K. pneumoniae et E.
coli. Ces enzymes hydrolysent les carbapénèmes faiblement
(Queenan et Bush. 2007 ; Poirel et al. 2007). Les
carbapénémases de classe A, les plus fréquentes et les
plus menaçantes, sont les carbapénémases de type KPC
(Nordmann et al. 2009). La première souche exprimant KPC fut
identifiée chez une souche de K. pneumoniae en 1996, en
Caroline du Nord aux États-Unis (Yigit et al. 2001). Elle
hydrolyse toutes les bêtalactamines. Son activité est
partiellement inhibée par l'acide clavulanique ou le tazobactam. Le plus
souvent les souches qui produisent KPC expriment également d'autres
bêta-lactamases dont de nombreux types de BLSE (TEM, SHV, CTX-M) et
possèdent un certain degré de résistance par
imperméabilité. Les souches KPC apparaissent donc le plus souvent
multirésistantes aux bêta-lactamines, l'ertapénème
étant la carbapénème dont le niveau de résistance
est le plus élevé. Les études génétiques
montrent que ces gènes KPC sont localisés sur une
variété importante de plasmides mais qu'ils sont associés
à des transposons de même nature de type Tn3. La
mobilité de ces plasmides et transposons contribuerait fortement
à la diffusion interespèce de ces gènes KPC (Nordmann et
al. 2009). L'association des gènes KPC à d'autres
gènes de résistance aux
27
Chpaitr 1 : Synthese bibliographique
antibiotiques sur de mêmes structures
génétiques explique en grande partie la multirésistance de
ces souches.
Tableau 3.Principales
carbapénémases des entérobactéries (Nordmann et
al.2012).
|
classificatio n Ambler
|
Nomde l'enzyme
|
Plasmide/ Chromosome
|
Spectre d'hydrolyse
|
inhibiteurs
|
|
|
|
Pen
|
C1G
|
G
|
C3G
|
ATM
|
CARBA
|
|
|
A
|
SEM-1 to -3
|
Chromosome
|
++
|
++
|
-
|
+
|
+
|
+
|
acid clav, TAZ, SULB, NXL-104
|
|
NMC-A
|
Chromosome
|
++
|
++
|
-
|
+
|
-
|
++
|
|
IMI-2
|
Plasmide
|
++
|
++
|
-
|
+
|
-
|
++
|
|
GES-4,- 5,-6
|
Plasmide
|
++
|
++
|
+
|
+
|
-
|
+
|
|
KPC-2 to -12
|
Plasmide
|
++
|
++
|
-
|
++
|
+
|
++
|
acid clav
TAZ, SULB, acidBoron ic
|
|
B
|
IMP-1 to
-33
|
Plasmide
|
++
|
++
|
++
|
++
|
-
|
++
|
EDTA
|
|
VIM-1 to -33
|
Plasmide
|
++
|
++
|
++
|
++
|
-
|
++
|
|
NDM-1 to -6
|
Plasmide
|
++
|
++
|
++
|
++
|
-
|
+
|
|
KHM-1
|
Plasmide
|
++
|
++
|
++
|
++
|
-
|
++
|
|
D
|
OXA-48
|
Plasmide
|
++
|
++
|
+/-
|
+/-
|
-
|
+
|
NaCl
|
|
|
|
|
|
|
|
|
|
OXA-181
|
Plasmide
|
++
|
++
|
+/-
|
+/-
|
-
|
+
|
28
Chpaitr 1 : Synthese bibliographique
? Les Carbapénèmases de classe
B
Les premières carbapénémases de classe B
(IMP) (ou métallo bêtalactamases, MBL) avaient été
identifiées chez des espèces d'entérobactéries
typiquement hospitalières (Serratia, Citrobacter et
Enterobacter) au Japon (Queenan et Bush. 2007 ; Walsh. 2008). Puis
d'autres MBL ont été isolées dans le monde entier ; il
s'agit de nombreuses variétés de bêtalactamases de type IMP
et VIM, GIM-1, KHM-1 et NDM-1 (Walsh. 2008 ; Yong et al. 2009). Ce
sont des métallo-enzymes qui contiennent des ions zinc dans leur site
actif. Ces enzymes hydrolysent fortement toutes les bêtalactamines
à l'exception de l'aztreonam. Leur activité n'est inhibée
ni par l'acide clavulanique ni par le tazobactam. Les niveaux de
résistance aux carbapénèmes sont assez variables (Queenan
et Bush. 2007 ; Walsh. 2008). Dans de nombreux cas, les souches productrices de
MBL produisent aussi des BLSE (Walsh. 2008). Les gènes de ces MBL sont,
le plus souvent, plasmidique et associés au sein d'intégrons et
de transposons, structures assurant la mobilité de ces gènes de
résistance et la multirésistance aux antibiotiques des souches
(Queenan et Bush. 2007 ; Walsh. 2008).
? Les Carbapénèmases de classe C et
D
Les carbapénèmases de classe D, OXA-48,
décrites tout d'abord chez K. pneumoniae (Poirel et
al. 2004) hydrolysent fortement les carbapénèmes et
n'hydrolysent pas les céphalosporines de 3e
génération. Son activité n'est pas inhibée par
l'acide clavulanique (Poirel et al. 2004) OXA-48 est souvent
associée à d'autres bêtalactamases, en particulier des
BLSE, ce qui contribue à la multirésistance des souches (Carrer
et al. 2010 ; Gülmez et al. 2008). En l'absence d'autres
bêtalactamases, les souches qui ne produisent que l'OXA-48 peuvent ne
présenter qu'une légère diminution de la
sensibilité aux carbapénèmes (Aktas et al. 2008).
Le réservoir naturel de ce gène OXA-48 a été
identifié ; il s'agit de Shewanella sp., ce qui suggère
le transfert de ce gène de résistance en milieu aqueux (Poirel et
al. 2004) Le gène d'OXA-48 est localisé au sein d'un
transposon comportant deux séquences d'insertion identiques assurant
mobilité et expression (Poirel et al. 2004).
29
Chpaitr 1 : Synthese bibliographique
6- Epidémiologie mondiale des
entérobactéries BLSE 6-1- Les BLSE classiques
Depuis plus de 30 ans, la résistance des
entérobactéries aux C3G ne cesse de se renforcer notamment par
l'acquisition de BLSE. De nombreuses études relatent la progression
continue à l'échelle mondiale de ce type de résistance
(Belmonte et al. 2010).
L'avènement des BLSE a commencé en Europe de
l'Ouest mais a gagné rapidement les Etats-Unis, l'Asie et même
l'Afrique. Aujourd'hui leur répartition est mondiale et leur
prévalence très variable selon la localisation, l'espèce
bactérienne et l'origine des isolats (Bradfor. 2001a).
L'épidémiologie des BLSE a été
étudiée par de grands programmes de surveillance antimicrobienne
notamment le programme SENTRY dans la région Asie-pacifique entre 1998
et 2002 et le programme MYSTIC (Meropenem Yearly Susceptibility Test
Information Collection) en Europe et aux Etats-Unis entre 1997 et 2004.
6-1-1- En Europe
La première bêta-lactamase a été
isolée en Allemagne. Il s'agissait du SHV-2 décrit chez K.
pneumoniae en 1983 (Knothe et al. 1983) puis en 1985 chez K.
ozaenae (Kliebe et al. 1985). En France, en 1984, une variante de
TEM-2, nommée TEM-3 est détectée également chez
K. pneumoniae dans différents hôpitaux. En 1989, presque
simultanément en Allemagne en France et en Italie, une nouvelle famille
de BLSE a été reconnue et nommée CTX-M. En 1991, à
Ankara (Turquie), et plus tard en France, les enzymes de type OXA, ont
été découvertes (Canton et al. 2008).
Les données du programme de surveillance MYSTIC en
Europe entre 1997 et 2004 ont montré une augmentation de la
prévalence et la dispersion des souches productrices de BLSE. Chez les
souches de E. coli productrices de BLSE, la prévalence va de
2,1 % en 1997 à 10,8 % en 2004; chez K. pneumoniae elle va de
9,0 % à 13,6 % (Goossens et Grabein. 2005).
6-1-2- En Afrique
Deux études ont permis de faire le point sur la
production des BLSE en Afrique. La première relève la
diversité de la prévalence des souches productrices de BLSE sur
le continent et conclut que la proportion de ces souches est inférieure
à 15 % (Tansarli et al. 2014). Dans la partie nord de
l'Afrique, on retrouve les gènes CTX-M-3, CTX-M-15 et TEM-1 chez
K.
30
Chpaitr 1 : Synthese bibliographique
pneumoniae et TEM, SHV et CTX-M-1 chez E.
cloacae avec une prévalence de 32,1% parmi les
entérobactéries en Algérie (Messai et al. 2008;
Ahmed et al. 2012; Nedjai et al. 2013).Au Maroc, la
prévalence est de 1.3 % avec les types TEM-1, SHV-5 et CTX-M-15 chez
E. coli (Bourjilat et al. 2011). En Ethiopie, la
prévalence de Klebsiella spp productrice de BLSE est de 33,3 %
(Seid et Asrat. 2005) En Afrique du sud, une étude identifie TEM-1,-26,
SHV-2,-5 chez Proteus mirabilis et E. coli (Pitout et
al. 1998). En Afrique centrale, il a été rapporté
au Cameroun les BLSE de type SHV-12, CTX-M-15, OXA-1, OXA-30, TEM-1 avec un
portage fécale de 54,06 % (Lonchel et al. 2013a; Lonchel et
al. 2013b).En Centrafrique, la prévalence est de 4 % chez les
entérobactéries avec les types SHV-2A, SHV-12, CTX-M-3, CTX-M-15
et TEM-1 (Frank et al. 2006). En Afrique de l'Ouest, E. coli
productrice de CTX-M-15 est retrouvé chez les enfants au
Sénégal (Ruppé et al. 2009) et où la
prévalence est estimée à 3,8 % (Sire et al.
2007). Au Nigeria, CTX-M-15, SHV, TEM ont été signalés
chez K. pneumoniae (Soge et al. 2006) et OXA-10, VEB-1, CMY
chez Providencia spp (Aibinu et al. 2011).
La deuxième étude conclut que les
entérobactéries hospitalières et communautaires
productrices de BLSE varient entre les pays et selon les échantillons
considérés mais reste communes en Afrique. Ce sont les enzymes de
classe A et D (Storberg. 2014). La vraie prédominance des BLSE n'est pas
connue, probablement en raison des difficultés de leur détection.
Mais il est clair que leur prévalence et leur distribution mondiale
augmente de jours en jours.
6-1-3- En Asie
En Asie, des études sur la résistance aux
antibiotiques ont montré des niveaux élevés des
phénotypes BLSE en Chine, en Corée, au Japon et en Inde. Des
travaux à la fin des années 1990 suggèrent que SHV-5 et
SHV-12 étaient les plus communs. En Chine, et d'autres pays d'Asie, les
types dominants sont CTX-M-14 et CTX-M-3 et CTX-M-2 au Japon. En Inde la
prévalence des phénotypes BLSE chez E. coli et K.
pneumoniae est de68 % (Hawkey. 2008). Dans le cadre du programme SENTRY,
l'incidence de la production de BLSE chez E. coli varie de 13 %
à 35 % et un taux > 20 % chez K. pneumoniae, en Chine, au
Japon, en Taïwan, au Singapore (Hirakata et al. 2005).
31
Chpaitr 1 : Synthese bibliographique
6-1-4- En Amérique
Aux Etats-Unis, les grandes études de surveillance ont
été menées par le CDC et par le programme MYSTIC.
L'occurrence de la production de BLSE chez les entérobactéries
s'étend de 0 à 25%, selon les établissements, avec une
moyenne autour de 3% (CDC National Nosocomial Infections Surveillance
http://www.cdc.gov/ncidod/hip/SURVEILL/NNIS.HTM
et Bradford, 2001a). Une analyse des données du programme MYSTIC entre
1999-2004 aux Etats-Unis a révélé 5,1 % et 7,2 % de
phénotype BLSE chez les souches de E. coli et Klebsiella
spp, respectivement en 1999 et une baisse en 2004, notamment 1,4 % pour
E. coli et 4,4 % pour Klebsiella spp (Goossens et Grabein.
2005). Peu de CTX-M ont été signalés aux Etats-Unis,
contrairement au reste du monde (Bush. 2008). Au Canada, une grande
variété de BLSE a été identifiée, avec des
membres de la classe TEM, SHV et CTX-M-14. Au Mexique l'enzyme TLA-1
hébergé par une souche d'E. coli a été
rapportée en 1993 (Naas et al. 2008).
En Amérique du Sud le taux de BLSE est parmi les plus
élevés du monde. Les types SHV et TEM sont fréquents,
CTX-M est endémique et largement dominant. Selon de récentes
enquêtes multi-continentales, les Klebsiella isolés de
l'Amérique latine ont la prévalence la plus élevée
de BLSE (45,4 %). Chez E. coli, elle est de 8,5 % et chez P.
mirabilis, 22,4 % (Données programme SENTRY) (Winokur et
al. 2001).
En 15 ans, la diffusion mondiale des BLSE de type CTX-M chez
les entérobactéries a explosé de façon
extrêmement rapide, d'où le terme de «pandémie
CTX-M» (Rossolini et al. 2008). Les études
épidémiologiques récentes rapportent que la situation est
endémique dans la plupart des pays d'Europe, d'Asie et d'Amérique
du Sud avec de forts taux de prévalence de CTX-M parmi les souches
productrices de BLSE : E. coli (de 30 à 90%) et de K.
pneumoniae (de 10 à 60 %) (Livermore et al. 2007 ;
Rossolini et al. 2008). A noter que quelques CTX-M sont
retrouvées spécifiquement dans certains pays (comme CTX-M-9 et
CTX-M-14 en Espagne, CTX-M-1 en Italie ou CTX-M-2 en Amérique du Sud, au
Japon et en Palestine) tandis que CTX-M-15 est mondialement distribuée
et est responsable chez les humains des cas d'infections nosocomiales et
communautaires (Rossolini et al. 2008).
32
Chpaitr 1 : Synthese bibliographique
6-2- Les BLSE de type
carbapénèmases
Les carbapénémases de type KPC (Klebsiella
pneumoniae carbapénémase) ont tout d'abord été
décrites sur la côte Est des États Unis en 1996 (Yigit et
al. 2001) avant d'être identifiées dans la plupart des
états du pays, avec notamment une très forte prévalence
dans l'état de New York. Ces souches ont également
été décrites en Grèce et en Palestine où
elles semblent à l'heure actuelle endémiques (Nordmann et
al. 2009). Plusieurs variants ont été décrits
à ce jour (KPC-1 à KPC-11), le gène blaKPC-2 est
actuellement le plus répandu sur la planète. Si les niveaux de
résistance aux carbapénèmes des souches productrices
d'enzymes de type KPC sont très variables, l'ertapénème
est la molécule ayant la plus faible activité. Les souches
productrices de KPC sont très fréquemment des souches
multirésistantes et les options thérapeutiques sont très
souvent réduites. De ce fait, la mortalité associée aux
infections par les bactéries productrices d'enzyme de type KPC est
élevée, très souvent supérieure à 50%
(Nordmann et al. 2011).
Les enzymes de type VIM (Verona Integron encoded
Metallo-bêta-lactamase) et IMP (Imipénémase) qui
représentent la majorité des carbapénémases de
classe B ont été rejointes en 2008 par une nouvelle enzyme
appelée NDM-1 (NewDelhi metallo-bêta-lactamase 1).La
première métallo â-lactamase de type IMP a
été décrite au Japon en 1991 (Ito et al. 1995)
Depuis, ce type d'enzyme a diffusé dans le monde entier et les enzymes
de type VIM et IMP sont désormais endémiques dans certains pays
tels que la Grèce, l'Italie, l'Espagne, Taiwan et le Japon (Nordmann et
al. 2011).
La plupart des souches productrices de
métallo-â-lactamases sont des K. pneumoniae
multirésistantes acquises dans les établissements de soins.
La mortalité associée à ce type de souches varie de 18
à 67% (Nordmann et al. 2011).
La première souche de K. pneumoniae
productrice d'OXA-48 a été isolée en Turquie en 2003
(Poirel et al. 2004). Depuis, les bactéries productrices
d'oxacillinases, notamment OXA-48, ont très largement
émergé dans tous les pays du pourtour méditerranéen
et en Afrique (Nordmann et al. 2011). Plus récemment, des
souches produisant une oxacillinase similaire, OXA-181, ont été
isolées en Inde ou chez des patients d'origine indienne (Castanheira et
al. 2011). OXA-48 est principalement retrouvée chez K.
pneumoniae et E. coli, même si d'autres espèces
d'entérobactéries peuvent produire ce type d'enzyme. Les
carbapénémases de classe D hydrolysent plus faiblement les
carbapénèmes et les céphalosporines de
3ème génération, ce
33
Chpaitr 1 : Synthese bibliographique
qui sous-estime probablement leur fréquence de
diffusion chez les entérobactéries du monde entier.
Toutefois, le niveau de résistance aux
carbapénèmes est plus élevé lorsqu'elles sont
associées à une BLSE et à un défaut de
perméabilité membranaire (Carrer et al. 2010 ; Cuzon et
al. 2011). La mortalité associée à ce type d'infection
demeure inconnue à ce jour.
7- Facteurs de risque d'acquérir une
BLSE
Différents facteurs de risque ont été
fréquemment associés avec l'acquisition d'une souche productrice
de BLSE. Le premier facteur concerne l'utilisation accrue des antibiotiques de
type céphalosporines de 3ème génération quelques
années avant l'apparition des premières BLSE, et par
conséquent, la mise en évidence d'un lien de causalité
entre cette utilisation et l'émergence des BLSE (Sirot. 1989). En
d'autres termes, les antibiotiques exercent une pression de sélection
non-négligeable (Jacobson et al. 1995), et cette pression de
sélection est d'autant plus marquée que le nombre de patients
traités est important et que la durée de l'antibiothérapie
est longue (Asensio et al.2000). De plus, on peut constater que la
restriction de l'utilisation des antibiotiques a permis la diminution du nombre
de BLSE (Chow et al. 1991 ; Follath et al. 1987).
Le deuxième facteur de risque concerne la
dissémination des souches résistantes et englobe d'une part le
problème des « réservoirs » et d'autre part la
transmission des germes. De plus, différents facteurs en relation avec
l'acquisition de bactéries productrices de BLSE concerne des patients
gravement malades, suite à une hospitalisation prolongée et
après exposition à des dispositifs invasifs (cathéters
veineux, sonde urinaire ou tube endotrachéal). Un séjour de
longue durée implique une plus longue exposition au risque
d'acquérir une bactérie multi-résistante comme E. coli
et Klebsiella (Kassis-Chikhani et al. 2004 ; Wiener et
al. 1999), ce qui signifie une augmentation du risque pour le patient
d'être colonisé (Goldsteinet al. 1995). D'autres facteurs
de risque sont la malnutrition, l'hémodialyse, la nutrition
parentérale totale, l'admission en réanimation ou
l'hospitalisation préalable (Lautenbach et al. 2001).
CHAPITRE 2
MATERIEL ET
METHODES
34
Chpaitr 02 : Materiels et méthodes
Matériel et méthodes
Cette étude portant sur l'évaluation de la
résistance aux antibiotiques des entérobactéries est
divisée en deux étapes :
- La première étape se propose de faire une
étude rétrospective des données, de l'isolement des
entérobactéries provenant de malades externes ou
hospitalisés dans les différents services du CHU SAADNA Mohamed
Abdennour de Sétif, durant la période de Janvier 2013 à
Avril 2017. Le logiciel de sauvegarde «Whonet 5.6 » utilisé au
laboratoire a été notre source d'information.
- Dans un deuxième temps, les
entérobactéries BLSE positifs isolées durant
l'année 2017, sont caractérisées du point de vue
moléculaire par l'extraction de plasmides et la détermination des
gènes de résistance codant pour ces â-lactamases en
utilisant une PCR-Multiplex.
1- Prélèvement et
identification
Les prélèvements issus des malades des
différents services hospitaliers ou ceux du milieu extra hospitalier
sont recueillis et guidés selon la nature du prélèvement
à la paillasse approprié comme suit :
- Paillasse Divers(1) : Pus, PB, Crachat.
- Paillasse Divers(2) : PV, KT, Sonde.
- Paillasse ECBU : Urine.
- Paillasse Hémoculture : Sang.
- Paillasse Coproculture : Selles, PR.
- Paillasse Ponction : LCR, LP, Liquide
Sinovial...
Après isolement et purification des
entérobactéries sur milieu sélectif BCP ou Hektoen. Leur
caractérisation est déterminée par une identification
biochimiques en utilisant différentes méthodes, la galerie
classique, la galerie Api 20E (la plus pratique) ou l'automate
SIEMENS MicroScan WalkAway.
1-1- La mini galerie (galerie classique)
La mini galerie repose sur plusieurs tests biochimiques
? L'urée-indole : permettant la recherche
de l'uréase et de l'indole
35
Chpaitr 02 : Materiels et méthodes
· La gélose TSI (Triple Sugar Iron)
:l'utilisation des sucres (glucose, lactose et saccharose) est
détectée par le virage de l'indicateur de pH, et la production
H2S est révélée la présence d'un
précipité noir.
· La gélose Mannitol-Mobilité :
l'utilisation du mannitol (changement de couleur du rouge au jaune) et
de la mobilité du germe.
· La gélose citrate de Simmons :
utilisé pour étudier la capacité de la
bactérie à croître en présence du citrate de sodium
comme seule source de carbone.
· Le milieu Clarck & Lubs : la mise
en évidence des voies fermentaires, voie du butan-2,3-diol (VP+) ou voie
des acides mixtes (RM+).
· Les acides aminés (ADH-ODC-LDC) :
permettant de voir le métabolisme protéique pour
différencier chaque espèce d'une autre.
Les milieux ensemencés sont incubés à
37°C/24h et la lecture se fait selon le test. 1-2- La galerie Api
20E
L'identification par la galerie rapide API 20E
(Analytical profil index) est un système pour l'identification des
entérobactéries utilisant 20 tests biochimiques
standardisés et miniaturisés, ainsi qu'une base de
données.
La galerie est ensemencée, à l'aide d'une
pipette Pasteur, par une suspension bactérienne dont l'opacité
est équivalente à 0,5 McFarland. Les tubes et les cupules de la
galerie sont remplis selon les recommandations du fournisseur. L'incubation se
fait à 37 C° #177; 1C° pendant 18-24h.
La lecture des galeries se fait selon les indications du
fournisseur. Après codification des réactions en un profil
numérique, on se réfère à un catalogue analytique
où l'identification est donnée avec un pourcentage et une
appréciation.
1-3- Le systeme MicroScan WalkAways SIEMENS
Le système WalkAways est un automate-incubateur
relié à un ordinateur comportant le logiciel d'identification des
bactéries, il ressemble à la galerie Api 20E mais de
manière plus sophistiquée. Ce système utilise des plaques
de 96 puits avec des réactifs lyophilisés et chaque
microorganisme à sa plaque spécifique comme suit :
· Combo 54 : bacille Gram négatif non fermentant
(Lactose -).
· Combo 53 : bacille Gram négatif fermentant
(Lactose +).
36
Chpaitr 02 : Materiels et méthodes
? Combo 28 : cocci Gram positif
(Staphylocoques).
? Combo PC1A : cocci Gram positif
(Streptocoques).
La plaque ensemencée sera incubée dans
l'automate pendent 24h/37°C, puis elle sera lu grâce au logiciel et
le résultat est imprimé.
2- Antibiogramme
Une fois la souche isolée et purifiée, sa
sensibilité aux antibiotiques est testée par l'antibiogramme
standard réalisé par la méthode de diffusion des
disques(CA-SFM/EUCAST, 2014) qui permet la détermination du
phénotype sensible (S) de l'espèce et les différentes
résistances acquises (I ou R) avec des termes de diminution
significative de sensibilité (synergie, antagonisme).Une suspension
bactérienne préparée à partir d'une culture pure de
18-24 h est ajustée au standard 0,5 McFarland. A partir de cette
suspension bactérienne diluée au 1/10 dans l'eau physiologique
(0,9 % NaCl) des géloses de Mueller-Hinton sont ensemencées par
écouvillonnage. Après séchage des boîtes, des
disques de papier buvard imprégnés d'antibiotiques sont
déposés à la surface du milieu manuellement ou à
l'aide d'un distributeur automatique. L'incubation se fait 37 °C pendant
18 à 24h. La mesure du diamètre des zones d'inhibition autour de
chaque disque et comparée aux standards du CA-SFM et permet de
déterminer les phénotypes sensibles (S), intermédiaires
(I) et résistants (R).
Par l'automate, l'antibiogramme est
effectué grâce aux antibiotiques lyophilisés dans la
plaque. L'automate peut tester la sensibilité d'une souche envers un
antibiotique. Dans ce cas, l'inoculum préparé est valable
à la fois pour l'identification et l'antibiogramme. La plaque contient
les réactifs pour identifier et pour tester la sensibilité en
même temps, d'où l'avantage de l'automate, identification et
antibiogramme en 24h.
37
Chpaitr 02 : Materiels et méthodes
Tableau 4. Antibiotiques testés.
Antibiotiques
|
Abréviations
|
Charge (jig)
|
Antibiotiques
|
Abréviations
|
CH (jig/UI)
|
Ampicille
|
AMP
|
10
|
|
Ertapeneme
|
ETP
|
10
|
|
Amoxicilline
|
AMX
|
25
|
|
Meropeneme
|
MEP
|
10
|
|
Ticarcilline
|
TIC
|
|
|
Imipeneme
|
IPM
|
10
|
|
Amoxicilline/ Acide
Clavulanique
|
AMC
|
25 /
|
10
|
Acide
Nalidixique
|
AN
|
30
|
|
Piperacilline/ Tazobactam
|
PTZ
|
75 /
|
10
|
Gentamycine
|
GM
|
10
|
|
Aztreonam
|
AZM
|
30
|
|
Amikacine
|
AMK
|
30
|
|
Cefazoline
|
CZO
|
30
|
|
Tobramycine
|
TOB
|
10
|
|
Cefuroxime
|
CXM
|
30
|
|
Tigecilline
|
TGY
|
30
|
|
Cephalothine
|
CEP
|
30
|
|
Minocycline
|
MIC
|
30
|
|
Cefoxitine
|
FOX
|
30
|
|
Ciprofloxacin
|
CIP
|
5
|
|
Cefepime
|
FEP
|
30
|
|
Levofloxacin
|
LVX
|
5
|
|
Cefotaxime
|
CTX
|
30
|
|
Fosfomycine
|
FOS
|
50
|
|
Ceftazidime
|
CAZ
|
30
|
|
Sulfaméthoxa zole-
triméthoprim
|
SXT
|
1.25
|
/ 23.75
|
|
3- Détection phénotypique des
EBLSE
Dans le cas de l'automate, la détection des EBLSE est
automatique par la technique des CMI. Par contre, sur l'antibiogramme standard
la réduction de la sensibilité aux céphalosporines de
troisième génération nécessite la mise en
évidence de l'existence d'une BLSE par la recherche d'une synergie entre
l'acide clavulanique et les céphalosporines de troisième
génération selon les techniques suivantes :
38
Chpaitr 02 : Materiels et méthodes
3-1- Test de synergie
La détection de f3-lactamases à spectre
élargi peut être mise en évidence par la méthode des
disques consistant à rechercher une image de synergie entre un disque
d'antibiotique contenant un inhibiteur de f3-lactamase et les disques de
céphalosporines de troisième génération
(cefotaxime, ceftazidime et cefopérazone) et l'aztréonam. Cette
synergie se traduitpar l'élargissement de la zone d'inhibition en regard
du disque de C3G sous forme d'un "bouchon de champagne" (Jarlier et
al. 1988).
Les disques d'amoxicilline + acide clavulanique (AMC 20/10)
et les disques de C3G (CTX 30ìg, FEP 30ìg et CAZ 30ìg) et
l'aztréonam (ATM 30ìg) sont déposés à la
surface du milieu MH à une distance de 20 à 30 mm entre les
disques. Après incubation à 37°C/18hle test est
considéré positif quand le diamètre d'inhibition du disque
de céphalosporine de troisième génération
appliqué après pré-diffusion du disque de l'AMC est
supérieur ou égale de 4 à 5 mm par rapport au
diamètre d'inhibition du disque C3G.
CAZ
AMC
ATM
CTX
FEP
Figure 7. Disposition des disques d'antibiotique
pour le test de synergie.
3-2- Test du double disque
Le test espagnol comme il est appelé, consiste
à rechercher une augmentation de la zone d'inhibition d'un disque de
C3G, précédé par l'application d'un disque contenant
l'AMC, comparé à un autre disque portant la même
céphalosporine et placé côte à côte sur la
gélose
39
Chpaitr 02 : Materiels et méthodes
de MH (Rahal et al. 2005).On procède de la
même manière que la technique d'antibiogramme dans la
préparation de l'inoculum et l'ensemencement, puis on place deux
antibiotiques : un disque d'AMC et un disque de CTX (C3G) à une distance
de 25 mm selon la figure. On laisse diffuser à température
ambiante pendant une heure et on remplace le disque d'AMC par un disque de CTX
(C3G).On incube pendant 18 h à 37°C. Le test du double disque est
considéré positif quand le diamètre d'inhibition du disque
de céphalosporine de troisième génération
appliqué après pré-diffusion du disque de l'AMC est
supérieur ou égale de 4 à 5 mm par rapport au
diamètre d'inhibition du disque C3G.

CTX
CTX
1 heure
Après
AMC CTX
Figure 8. Schéma de détection de
BLSE par le test du double disque
(Rahal et al. 2005).
3-3- Test à la cloxacilline
Le test à la cloxacilline est effectué pour
identifier une BLSE associée à une céphalosporinase
déréprimée. La cloxacilline ajoutée au milieu MH
(0,25 mg/ml) inhibe très fortement toutes les céphalosporinases
hyper-produites (Drieux et al. 2008 ; Philippon et Arlet. 2006). Si un
tel mécanisme de résistance existe, les boites de Pétri
contenant le milieu MH additionné de cloxacilline présentent une
restauration de l'activité de â-lactamases et apparition de
l'image de synergie en bouchon de champagne recherchée.
40
Chpaitr 02 : Materiels et méthodes
4- Détection génotypique des
EBLSE
4- 1- Extraction d'ADN
L'extraction est réalisée par deux
méthodes selon la nature plasmidique ou chromosomique de l'ADN.
a) Extraction d'ADN plasmidique par la lyse
alcaline
La méthode de la lyse alcaline est une technique
rapide permettant l'extraction des plasmides (ADN extra-chromosomiques) de
différentes tailles, de petite et de grande taille (>100kb).Elle met
à profit les propriétés structurales de l'ADN plasmidique,
qui est libérée après la lyse des bactéries sous
des conditions dénaturant l'ADN chromosomique (Kado et Liu. 1981).
L'extraction est réalisée selon le protocole
suivant :
- Des cultures bactériennes sur milieu LB sont
réalisées la veille.1ml de la culture est centrifugé dans
une tube Eppendorf (13000 rpm/1 min). Le culot ainsi obtenu est resuspendu dans
300 ìl du tampon P1 et agité.300 ìl du tampon P2 sont
ajoutés (tampon de lyse) et les tubes sont agités par inversion
environ 6 fois.300 ìl du tampon P3 est additionné et le tube est
agité par inversion (formation d'un précipité).Le
précipité est centrifugé à 13000 rpm/10 mn et le
surnageant est transféré dans un nouveau tube Eppendorf auquel
sont ajoutés 630ìl d'isopropanol refroidie à -20°C
pour précipiter l'ADN. Après centrifugationà13000 rpm/15
mn le surnageant est éliminé et le culot est lavé de ses
sels avec 700ìl d'éthanol à 70%. L'alcool
éliminé, le culot est, ensuite, séché à
l'air libre ou à l'étuve puis il est resuspendu dans 50ìl
de TE (Tris/HCl-EDTA) et conservé à -20°C.
Tableau 5. Composition des différents
tampons d'extraction.
Tampon P1
|
- 50 mM Tris/HCl (pH 8) - 10 mM EDTA (pH 8) - 1 ìl
d'ARNase
|
Tampon P2
|
- 0.2 M NaOH - 1 % SDS
|
Tampon P3
|
- 88.3 g K-acétate
- 15 ml acide formique - 300 ml H2O
|
|
41
42
Chpaitr 02 : Materiels et méthodes
b) Extraction de l'ADN chromosomique par
Boiling
L'ADN des entérobactéries est extrait par la
méthode du boiling. 1.5ml de la culture sur milieu LB est
centrifugé à 12000 rpm/1mn. Le culot bactérien est
resuspendu dans 180 ìl de TE et agité. Ce dernier est
porté à ébullition à 100 °C/10mn puis refroidi
à -20°C/10mn. Après centrifugation à 12000rpm/10 mn,
le surnageant est récupéré dans un nouveau tube Eppendorf
et conservé à 4°C pendant une nuit pour éventuelle
dégradation de l'ARN puis à - 20°C pour une utilisation
ultérieure.
4-2- Amplification par PCR (Polymerase Chain
Reaction)
La recherche des gènes de résistance
blaCTX-M, blaSHV et blaTEM a été
effectué par une PCR-Multiplex.
4-2-1- Principe
La technique de réaction de polymérisation en
chaîne ou PCR permet d'amplifier en un nombre élevé de
copies une séquence particulière d'ADN. La polymérisation
se réalise dans un mélange réactionnel contenant de
faibles quantités d'ADN possédant la séquence à
amplifier, les amorces nucléotidiques complémentaires des
séquences qui encadrent la cible à amplifier, l'ADN
polymérase et un mélange des quatre dNTP (dATP, dTTP, dCTP et
dGTP). Elle débute par une étape de dénaturation thermique
de l'ADN à amplifier. Des séquences oligonucléotidiques
complémentaires ou amorces sont alors hybridées aux
extrémités 3' des deux brins du fragment d'ADN matrice.
L'allongement des amorces dans le sens 5'--> 3' est, ensuite, assuré
par l'ADN polymérase Taq. Les nouvelles molécules d'ADN ainsi
formées sont dénaturées et un nouveau cycle peut
commencer, la réaction se répétant ainsi jusqu'à
plusieurs dizaines de fois (Bonnet et al. 2000b ; Hennequin et
al. 2012).
Tableau 6. Amorces utilisées (Jouini et
al. 2007).
Génes
|
Amorces
|
Séquences
|
Tm°C
|
blaCTX-M
|
For
|
5'-CTC CGC TTT GCG ATG TGA AG -3'
|
59.4
|
|
Rev
|
5'-ACC GCG ATA TCG TTG GT -3'
|
52.8
|
blaSHV
|
For
|
5'-CAC TCA AGG ATG TAT TGT G -3'
|
52.4
|
|
Rev
|
5'-TTA GCG TTG CCA GTG CTC G -3'
|
58.8
|
blaTEM
|
For
|
5'-ATT CTT GAA GAC GAA AGG GC-3'
|
55.3
|
|
Rev
|
5'-ACG CTC AGT GGA ACG AAA AC-3'
|
57.3
|
|
Chpaitr 02 : Materiels et méthodes
La réaction PCR d'un volume final de 25 ìl, est
composée de
- dNTP 0,5ul,
- MgCl2+ tampon 4ul,
- Taq-polymérase (solis biodine) 0,3ul,
- ADN de l'échantillon 5ul,
- Amorces spécifiques du gène à
amplifier : 0,2ul (de chaque),
- H2O (eau ultra pure stérile) qsp 25ul.
L'amplification de l'ADN se déroule selon les
étapes décrites ci -dessous
Tableau 7.Etapes de la PCR-Multiplex.
blaCTX-M/SHV-TEM
|
|
|
Dénaturation initial
|
94°C
|
/ 5 min
|
Dénaturation
|
94°C
|
/ 1 min
|
Hybridation
|
52°C
|
/ 1 min
|
Elongation
|
72°C
|
/ 1 min
|
Elongation final
|
72°C
|
/ 5 min
|
Nombre de cycles
|
30
|
|
|
Pour chaque PCR, il faut toujours un témoin positif et un
témoin négatif. L'amplification se déroule dans un
thermocycleur TC-4000.

Figure 9. Photo du thermocycleur TC-4000.
Chpaitr 02 : Materiels et méthodes
4-3- Electrophorèse sur gel d'agarose
Les produits d'amplification sont analysés sur gel
d'agarose à 1.5% (p/v) contenant 10ul d'une solution de bromure
d'éthidium (0,5 mg/l).Par contre, l'analyse des plasmides se fait sur un
gel à 0.8%. La migration est réalisé pendant 30mn à
100V et le gel est visualisé sous lumière UV et
photographié.

Figure 10. Photo d'une migration d'ADN par
électrophorèse.

43
Figure 11. Photo du transilluminateur
Fisher-Bioblock UV.
CHAPITRE 3
RESULTATS ET
DISCUSSION
44
Chapitre 03 : Résultats et discussion
Résultats
1 -Entérobactéries
Durant cette étude, 2421 souches
d'entérobactéries ont été isolées pendant la
période de 4 ans et 4 mois (du 01/01/2013 au 30/04/2017), de patients
hospitalisés ou externes du Centre Hospitalo-universitaire de
Sétif.
1-1- Répartition des
entérobactéries par espèces
Les souches d'entérobactéries
identifiées sont réparties comme suit ; E. coli
est la souche la plus fréquente (n= 1160), suivie par K.
pneumoniae (n=479) et P.mirabilis (n=216) ces trois souches
représentent plus de 67% des entérobatéries
isolées. Les autres espèces minoritaires sont E. cloacae
(n=91), Enterobacter sp (n=59), Klebsiella sp (n=42), M.
morganii (n=40), S. macescens (n=38), K. oxytoca (n=35),
Citrobacter sp (n=30), C. freundii (n=28) et P. vulgaris
(n=27). D'autresespèces rarement isoléessont
identifiées comme Enterobacter gergoviae, Pantoea, Providencia,
Salmonella, Shigella, Hafnia, Cedecae devisae, Ewingella... (n=176)
(fig.12).

45,00%
40,00%
50,00%
35,00%
30,00%
25,00%
20,00%
15,00%
10,00%
5,00%
0,00%
47,91%
19,78%
8,92% 3,76%
2,44%
1,73%
1,65%
1,57%
1,45%
1,24%
1,16%
7,28%
1,12%
Figure 12. Répartition des
entérobactéries selon l'espèce.
45
Chpaitr 03 : Résultats et discussion
1-2- Répartition des
entérobactéries selon l'origine de l'infection
Plus de la moitié des souches
d'entérobactéries isolées proviennent du milieu
hospitalier soit 57.71 %. Par contre (42.29%) des prélèvements
représentent des infections communautaires (fig.13).

57.71%
42.29%
OUT IN
Figure 13. Pourcentages des
entérobactéries selon l'origine de l'infection.
1-3- Pourcentage des entérobactéries selon
le sexe
La population du sexe féminin est la plus touchée
par les infections à entérobactéries (54.29%) par rapport
au sexe masculin (44.97%) (fig.14).
46
Chpaitr 03 : Résultats et discussion
60,00%
50,00%
40,00%
30,00%
20,00%
10,00%
0,00%
Feminin
54,29%
Masculin
44,97%
Inconnue
0,74%
Figure 14. Pourcentages des souches
entériques selon le sexe.
1-4- Répartition des
entérobactéries selon les tranches d'âge
La majorité des entérobactéries sont
isolées, plus chez les adultes que chez les enfants ; mais lors de la
répartition de l'âge par tranche, la tranche d'âge 1-10 ans
est la plus élevé (14.38 %), suivi de la tranche 61 et plus
(13.06 %), la tranche 21-30 ans est en troisième position (11.42%), de
31-40 ans (8.33%), de 51-60 ans (7.30%), de 11-20 ans (6.93%), de 41-50 ans
(6.18%), alors que les nourrissons de <1 ans en dernier(1.20%). Cependant,
(31.20%) d'entérobactéries ont été isolées
d'un sexe inconnu (fig. 15).
35,00%
30,00%
25,00%
20,00%
15,00% 11,42%
8,33% 6,18% 7,30%
10,00%
5,00%
0,00%
Inconnu < 1 ans 01-10
ans
31,20%
1,20%
14,38%
6,93%
11-20
ans
21-30
ans
31-40
ans
41-50
51-60 61 ans
ans ans et plus
13,06%
Figure 15. Répartition des
entérobactéries selon les tranches d'âge.
47
Chpaitr 03 : Résultats et discussion
1-5- Profil de résistance des
entérobactéries aux antibiotiques
L'étude de la sensibilité des
entérobactéries aux antibiotiques atteste d'un taux très
élevé de résistance aux bêtalactamines (fig.16). Les
résistances maximales sont notées pour
l'amoxicilline où 98.40% des souches sont
résistantes, suivie par la céfazoline
(88.90%) et
l'aztréonam (52.79%). La résistance aux C3G est également
élevée, elle est de 40.72% et 30.10% respectivement pour la
cefotaxime et la ceftazidime. En revanche, une sensibilité relative
existe pour l'augmentin (12.58%), la céfépime (8.39%) et le
ciprofloxacine (8.24%).Les plus faibles résistances sont notées
pour les carbapénèmes (l'imipeneme et l'ertapeneme) et la
cefoxitine.
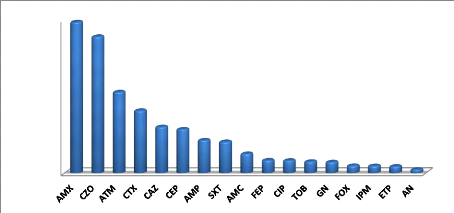
100,00%
40,00%
90,00%
80,00%
70,00%
60,00%
50,00%
30,00%
20,00%
10,00%
0,00%
98,40%
88,90%
52,79%
40,72%
30,10%
28,57%
21,43%
20,38%
12,58%
8,39%
8,24%
7,53%
4,82%
7,22%
4,71%
4,36%
2,35%
Figure 16. Pourcentages de résistance des
entérobactéries aux antibiotiques
1-6- Profil de résistance des
entérobactéries aux antibiotiques par année
Les données décrites ci-dessous relatent d'une
manière plus détaillée l'évolution de la
résistance des entérobactéries aux f3-lactamines et aux
autres antibiotiques par année, durant la période du 01/01/2013
au 30/04/2017 :
a) Résistance aux â-lactamines
La résistance des entérobactéries aux
f3-lactamines montre un taux élevé de résistance à
AMX de presque 100% pendant les 5 années étudiés et
à la CZO (C1G) d'un taux d'environ 90%. Une augmentation importante de
résistance à AMC, AMP et les C3G d'une moyenne de 510% par an est
notée. L'apparence considérable de la FOX et l'augmentation de la
résistance
48
Chpaitr 03 : Résultats et discussion
des antibiotiques du dernier recours IPM et ETP dans le
traitement constitue un véritable problème de santé
publique.
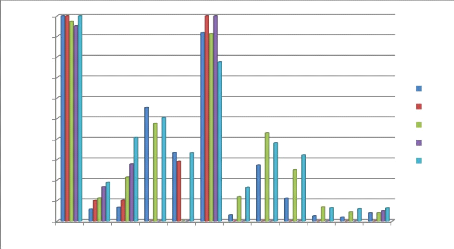
100,00%
40,00%
90,00%
80,00%
70,00%
60,00%
50,00%
30,00%
20,00%
10,00%
0,00%
AMX AMC AMP ATM CPN CZO FEP CTX CAZ FOX ETP
IPM
2013 2014 2015 2016 2017
Figure 17. Pourcentages de résistance des
entérobactéries aux f3-lactamines par année. b)
Résistance aux autres antibiotiques
Le graphe montre que l'évolution des
résistances aux quinolones et aux sulfamides est similaire à
celle des f3-lactamines, elle est en augmentation croissante à des taux
qu'il faut prendre en considération. En revanche, la résistance
aux aminosides est presque stable durant les cinq années avec une nette
diminution pour la gentamycine. Par contre, l'acide nalidixique reste
l'antibiotique le plus actif.
49
Chpaitr 03 : Résultats et discussion
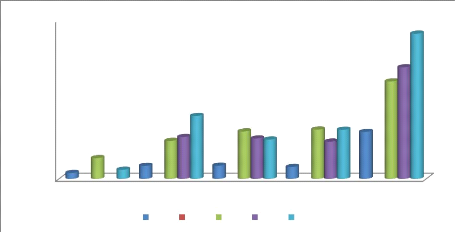
35,00%
30,00%
25,00%
20,00%
15,00%
10,00%
5,00%
0,00%
1,29%
4,57%
AN CIP GEN TOB SXT
2,00% 2,85%
2013 2014 2015 2016 2017
8,36%
9,22% 8,89%
13,86% 10,88%
10,45% 8,67% 10,82%
2,90% 2,61%
8,21%
10,35%
21,47%
24,60%
32,02%
Figure 18. Pourcentages de résistance des
entérobactéries aux autres antibotiques par année.
2- Les EBLSE
Le test de synergie est positif pour 53 souches
d'entérobactéries étudiées. Les 3 autres souches
ont montré un test de double synergie négatif. Pour ces souches,
il a été nécessaire de pratiquer le test de synergie sur
MH contenant de la cloxacilline à cause de la forte expression de leur
céphalosporinase.
2-1- Fréquence d'isolement des EBLSE
La prévalence de l'année 2017 a
débuté avec un pourcentage élevé des EBLSE
(35%) par rapport à la totalité des
entérobactéries isolées durant cette période.
EBLSE +
35%
EBLSE -
65%
Figure 19.Fréquence des EBLSE.
50
Chpaitr 03 : Résultats et discussion
2-2- Répartition des EBLSE selon
l'espèce
Les 56 entérobactéries productrices de
â-lactamases à spectre élargie isolées durant cette
période de 4 mois sont réparties comme suit : 23 E.
coli, 22 K. pneumoniae, 4 E. cloacae, 3 M.
morganii, 2 S. marcescens, 1 E. aerogenes et 1 C.
freundii.

4% 2% 2%
5%
7%
39%
41%
E. coli
K. pneumoniae E. cloacae
M. morganii S. marcescens E. aerogenes C. freundii
Figure 20. Répartition des EBLSE par
espèce.
2-3- Répartition des EBLSE selon l'origine de
l'infection
La majorité (75%) des entérobactéries
sécrétrices de BLSE sont isolées des infections d'origine
hospitalières. Les espèces bactériennes impliquées
sont E. cloacae et E. aerogenes isolées exclusivement
du milieu hospitalier et représentent des germes typiquement d'origine
nosocomiales. L'espèce C. freundii est issue d'une infection
communautaire. L'espèce S. marcescens semble ubiquitaire elle
est issue d'infections nosocomiale et communautaire. Par contre, les
espèces E. coli et K. pneumoniae et qui
représentent un nombre plus important de souches, leurs
fréquences d'isolement des infections nosocomiales sont plus
élevées et de l'ordre de (82.61%) et de (72.73%)
respectivement.
Chpaitr 03 : Résultats et discussion

100,00%
40,00%
90,00%
80,00%
70,00%
60,00%
50,00%
30,00%
20,00%
10,00%
0,00%
INF COMMU INF NOSO
Figure 21. Répartition des EBLSE selon
l'origine de l'infection.
2-4- Répartition des EBLSE selon le
service
Certains services hospitaliers sont apparus plus
concernés par le problème de résistance liée
à la production de BLSE, notamment les services des maladies
infectieuses et de la néphrologie d'où proviennent respectivement
(31%) et (12%) des souches BLSE, suivi par le service de pédiatrie avec
un taux de (10%). Au niveau des autres services l'isolement de souches BLSE est
faible et réparti comme suit : Hématologie (7%), Neurochirurgie
(7%), Médecine interne (7%), Cardiologie (5%), Bloc d'Urgence
Chirurgical (5%), Orthopédie (5%), Néo-natal (3%), Chirurgie
Femme (2%), Bloc d'Urgence Médical (2%), Réa-Médical (2%),
et Pneumo-phtisiologie (2%).

ORT
5%
CHF BUM
2% 2%
N-NAT
3%
CARDIO
5%
BUC
5%
7%
MI
N-CH
7%
HTO
7%
R-MED
2%
PED
10%
31%
CX
NEPH
12%
PPHT
2%
51
Figure 22. Répartition des EBLSE selon
le service.
Chpaitr 03 : Résultats et discussion
2-5- Répartition des EBLSE selon la nature du
prélèvement
La répartition des sites anatomiques
présentée dans la figure 23, montre que le site le plus
concerné par les EBLSE sont les urines (32%), suivi du pus (30%) dans
lesquels les germes ont été isolés. Les taux
retrouvés dans les autres prélèvements sont comme suit :
hémocultures (16%), LCR (11%), liquide d'ascite (3%) et (2%) pour chacun
des autres prélèvements Drain, Prélèvement Buccal,
sonde urinaire et trachéal.
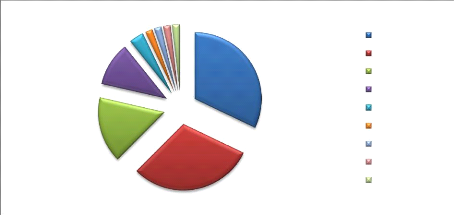
16%
11%
3%
2% 2% 2%
2%
30%
32%
URINE
PUS HEMOCULTURE LCR L. ASCITE
DRAIN
P. BUCCAL SONDE URINAIRE TRACHEAL
Figure 23. Répartition des EBLSE selon
la nature du prélèvement.
2-6- Répartition des EBLSE selon le
sexe
Le pourcentage est inversé par rapport à la
totalité des entérobactéries isolées, le sexe
masculin représente un taux plus élevé (51.79%) de la
fréquence d'isolement des EBLSE.
Féminin;
48,21%
Masculin;
51,79%
52
Figure 24. Répartition des EBLSE selon
le sexe.
53
Chpaitr 03 : Résultats et discussion
2-7- Profil de résistance des EBLSE

Figure 25.Test de synergie.
L'antibiogramme effectué pour les souches d'EBLSE a
permis d'étudier leurs profils de résistance vis-à-vis des
antibiotiques testés.
2-7-1- Résistance aux
â-lactamines
Durant la période d'étude, le taux de
résistance aux céphalosporines de
3émegénération a connu une augmentation
très remarquable. Les résultats de la résistance des 56
isolats d'entérobactéries productrices de BLSE montrent que
toutes les souches sont totalement
résistantes (100%) à l'ampicilline,
l'amoxicilline et la ticarcilline, ainsi, qu'aux
céphalosporines de
1ére génération (C1G) la céfazoline et
la céfalotine, aux G ; la céfuroxime, aux C3G (CAZ, CTX),
à la Céfepime (C4G) et à l'aztréonam. Concernant la
résistance de ces souches aux inhibiteurs des â-lactamines on note
une résistance élevée vis-à-vis de l'association
amoxicilline/acide clavulanique. Par contre, l'association
pipéracilline/tazobactam demeure plus active.
Il est à signaler aussi dans ce travail, la
présence de 16 souches différentes résistantes à la
céfoxitine (FOX), suggérant la possible production d'une
céphalosporinase plasmidique de type AmpC.
L'imipéneme et l'ertapeneme restent les antibiotiques
de choix avec la sensibilité de 54 souches pour l'imipéneme
contre 2 résistantes, 53 souches sensibles à l'ertapeneme contre
3
54
Chpaitr 03 : Résultats et discussion
souches. La sensibilité au méropeneme portant
sur 4 souches se sont montrées toutes sensibles. Cependant, la
légère résistance aux IPM et ERT est à prendre en
considération.
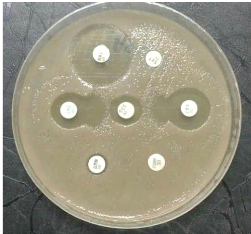
Figure 26. Test de synergie vu dans un
antibiogramme d'une souche Klebsiella pneumoniae
BLSE +.
2-7-2- Résistance aux aminosides
Des résistances associées aux aminosides sont
retrouvées chez plus de 60% des souches EBLSE, ce qui présente un
fort risque d'échec thérapeutique. Par contre, l'amikacine reste
l'antibiotique le plus efficace sur les souches EBLSE avec un taux de
sensibilité de 84%. 2-7-3- Résistance aux
quinolones
La résistance de souches EBLSE aux C3G est
associée à une résistance aux quinolones et aux
fluoroquinolones. Cette résistance est observée chez (70%) des
souches pour la ciprofloxacineet (35%) pour la levofloxacine.
2-7-4- Résistance aux autres
antibiotiques
L'étude de la sensibilité aux autres familles
d'antibiotiques a révélé aussi des résistances plus
au moins faibles. Quelques souches sont résistantes à la
tigécycline, la minocycline, l'association sulfaméthoxazole +
triméthoprime et la fosfomycine. Toutefois, la tigécycline et la
fosfomycine se sont montré les antibiotiques les plus actifs sur toutes
les souches testées.
55
Chpaitr 03 : Résultats et discussion

100%
40%
90%
80%
70%
60%
50%
30%
20%
10%
0%
AMP/AMX AMX/ACV PIP/TAZ TIC AZM CAZ CTX FEP CPN CZO CXM
FOX ETP IPM MER AMK GEN TOB TGY MIC SXT CIP LVX FOS
S I
R
Figure 27. Profil de résistance des
EBLSE aux antibiotiques.
3- Support génétique mobile de la
résistance ? Les plasmides
Les souches d'entérobactéries productrices de
BLSE ont subit une extraction de leur contenu plasmidique par la méthode
de (Kado et Liu. 1981).
L'analyse du contenu plasmidique des souches, après
migration sur gel d'agarose (0.8%) a révélé que la
majorité de souches hébergent un à quatre types de
plasmides.
56
Chpaitr 03 : Résultats et discussion
Tableau 8. Différents types de plasmides
extraits des EBLSE.
Nom de la souche
|
Code de la souche
|
Nombre de types de plasmide
|
Nombre d'isolats
|
Escherichia coli
|
Eco
|
1
|
N=19
|
|
|
|
(1.11.12.14.15.16.18.19.20.21
|
|
|
|
23.24.32.34.39.41.43.53.54)
|
Escherichia coli
|
Eco
|
2
|
N=1
|
|
|
|
(48)
|
Escherichia coli
|
Eco
|
3
|
N=2
|
|
|
|
(4.44)
|
Escherichia coli
|
Eco
|
4
|
N=1
|
|
|
|
(25)
|
Klebsiella pneumoniae
|
Kpn
|
1
|
N=12
|
|
|
|
(3.6.22.26.28.29.30
|
|
|
|
31.36.38.40.50)
|
Klebsiella pneumoniae
|
Kpn
|
2
|
N=6
|
|
|
|
(9.37.42.45.47.52)
|
Klebsiella pneumoniae
|
Kpn
|
3
|
N=3
|
|
|
|
(2.7.8)
|
Klebsiella pneumoniae
|
Kpn
|
4
|
N=1
|
|
|
|
(51)
|
Enterobacter cloacae
|
Ecl
|
1
|
N=3=)
|
|
|
|
(13.27.33)
|
Enterobacter cloacae
|
Ecl
|
3
|
N =1
|
|
|
|
(56)
|
Morganella morganii
|
Mmo
|
1
|
N=3
|
|
|
|
(17.46.55)
|
Serratia marcescens
|
Sma
|
1
|
N=2
|
|
|
|
(5.35)
|
Enterobacter
|
Eae
|
1
|
N=1
|
aerogenes
|
|
|
(10)
|
Citrobacter freundii
|
Cfr
|
1
|
N=1
|
|
|
|
(49)
|
|
( ) Numéros des isolats
Chpaitr 03 : Résultats et discussion

Figure 28. Profil plasmidique des 56 souches
d'EBLSE.
57
58
Chpaitr 03 : Résultats et discussion
4- Caractérisation des BLSE par la
PCR-multiplex
La caractérisation des gènes de BLSE
effectuée sur l'ADN (plasmidique ou chromosomique) de l'ensemble des
souches par l'amplification multiple des gènes CTX-M, TEM et SHV a
permis l'obtention de différents gènes de résistance
amplifiés sur 33 souches.
4-1- BLSE de type CTX-M
Ce type de BLSE se reconnaît facilement sur
l'antibiogramme en gélose car le CTX est habituellement la
molécule la plus touchée avec une très bonne inhibition
autour du disque contenant un inhibiteur de f3-lactamases.
La caractérisation moléculaire de f3-lactamases
par PCR a révélé que 29/33des souches
d'entérobactéries productrices de f3-lactamases à spectre
étendue provenant des patients à titre externe et de patients
hospitalisés aux différents services à l'hôpital,
sont toutes porteuses d'un gène blaCTX-M (Fig.29), soit une
prévalence de (87.88 %). Montré et Faire les espèces
59
Chpaitr 03 : Résultats et discussion

Figure 29. Amplifications par PCR-Multiplex des
gènes blaCTX-M, blaSHV et blaTEM.
60
Chpaitr 03 : Résultats et discussion
4-2- BLSE de type SHV
La BLSE de type SHV a été observée chez
15souches d'EBLSE soit une prévalence de (42.42 %) existant chez 12
souches K. pneumoniae, 2 d'E. coli et une souche d'E.
aerogenes.
4-3- BLSE de type TEM
Les BLSE de type TEM sont peu fréquentes, elles ont
été retrouvées chez 9 souches appartenant à K.
pneumoniae (N=6), E. coli (n=2) et une seule souche E.
aerogenes, soit une prévalence de (27.27 %).
souches %
|
E. coli (14)
|
K.
pneumoni
ae (14)
|
E. cloacae
(2)
|
M.
morganii
(2)
|
E.
aerogenes
(1)
|
blaCTX-M
|
68,75%
|
44,83%
|
100,00%
|
100,00%
|
33,33%
|
blaSHV
|
12,50%
|
37,93%
|
|
|
33,33%
|
blaTEM
|
18,75%
|
17,24%
|
|
|
33,33%
|
|
100,00%
40,00%
80,00%
60,00%
20,00%
0,00%
Figure 30. Prévalence des gènes de
résistance chez les souches amplifiées.
4-4- Résistances multiples
- BLSE de type CTX-M/SHV
La BLSE de type CTX-M/SHV est observée chez 13 souches
soit une prévalence de
(39.39%), 11 K. pneumoniae, une E. aerogenes
et une E. coli.
- BLSE de type CTX-M/TEM
Ce type de BLSE a été observé chez 6
souches soit (18.18%), 4 K. pneumoniae, 1 E.
aerogenes et 1 E. coli.
- BLSE de type CTX-M/SHV/TEM
La BLSE de type CTX-M/SHV/TEM est observée chez 5 souches
soit une prévalence de
(15.15 %), 4 K. pneumoniae et une seule souche
E. aerogenes.
61
Chpaitr 03 : Résultats et discussion
Tableau 9.Caractéristiques
générales et moléculaires des souches
d'entérobactéries productrices de â-lactamases à
spectre étendu isolées au CHU de Sétif.
Souches
|
Code
|
Services
|
prélèvement
|
Sexe
|
Gène BLSE
|
Kpn
|
1(72c)
|
Orth
|
Pus
|
F
|
blaCTX-M
|
Kpn
|
2(62c)
|
Hto
|
Sang
|
F
|
blaCTX-M
|
Kpn
|
3(72s)
|
Orth
|
Pus
|
F
|
blaSHV
|
Kpn
|
4(62s)
|
Hto
|
Sang
|
F
|
blaSHV
|
Kpn
|
5(72t)
|
Ortho
|
Pus
|
F
|
blaTEM
|
Kpn
|
6(62t)
|
Hto
|
Sang
|
F
|
blaTEM
|
Kpn
|
7
|
Ext
|
Urine
|
M
|
blaTEM
|
Kpn
|
10
|
BUC
|
LCR
|
M
|
blaCTX-M
blaSHV
|
Kpn
|
11
|
N-Ch
|
Trachéal
|
M
|
blaCTX-M
blaSHV
blaTEM
|
Kpn
|
18
|
Ext
|
Pus
|
M
|
blaCTX-M
blaSHV
|
Kpn
|
19
|
Cx
|
Urine
|
M
|
blaCTX-M
blaSHV
|
Kpn
|
20
|
Ext
|
Urine
|
M
|
blaCTX-M
blaSHV
|
Kpn
|
22
|
Orth
|
Pus
|
F
|
blaCTX-M
blaSHV
|
Kpn
|
23
|
Ext
|
Pus
|
F
|
blaCTX-M
blaSHV
|
Kpn
|
24
|
N-nat
|
LCR
|
F
|
blaCTX-M
|
Kpn
|
31
|
Néph
|
Sonde Uri
|
M
|
blaCTX-M
blaSHV
blaTEM
|
Kpn
|
32
|
Péd
|
LCR
|
M
|
blaCTX-M
blaSHV
|
Kpn
|
36
|
Péd
|
Urine
|
M
|
blaCTX-M
|
Kpn
|
38
|
Ortho
|
Pus
|
F
|
blaCTX-M
blaSHV
blaTEM
|
Kpn
|
39
|
Hto
|
Sang
|
F
|
blaCTX-M
blaSHV
blaTEM
|
|
62
Chpaitr 03 : Résultats et discussion
Eco
|
9
|
CHF
|
Pus
|
F
|
blaTEM
|
Eco
|
13
|
Cx
|
Pus
|
M
|
blaCTX-M
|
Eco
|
14
|
BUM
|
Pus
|
F
|
blaCTX-M
|
Eco
|
15
|
Ext
|
Urine
|
F
|
blaCTX-M
|
Eco
|
16
|
Péd
|
Urine
|
F
|
blaSHV
|
Eco
|
17
|
Cx
|
Urine
|
M
|
blaCTX-M blaSHV
|
Eco
|
21
|
Ext
|
Urine
|
F
|
blaCTX-M blaTEM
|
Eco
|
25
|
Cx
|
Pus
|
M
|
blaCTX-M
|
Eco
|
26
|
Péd
|
LCR
|
M
|
blaCTX-M
|
Eco
|
29
|
Cx
|
Urine
|
M
|
blaCTX-M
|
Eco
|
30
|
BUC
|
Pus
|
M
|
blaCTX-M
|
Eco
|
33
|
Cx
|
Urine
|
F
|
blaTEM
|
Eco
|
35
|
Cx
|
Urine
|
F
|
blaCTX-M
|
Eco
|
37
|
Ext
|
Urine
|
F
|
blaCTX-M
|
Eae
|
12
|
Cardio
|
Pus
|
M
|
blaCTX-M blaSHV blaTEM
|
Ecl
|
8
|
Ext
|
Urine
|
F
|
blaCTX-M
|
Ecl
|
27
|
Néph
|
Urine
|
F
|
blaCTX-M
|
Mmo
|
28
|
MI
|
Pus
|
M
|
blaCTX-M
|
Mmo
|
34
|
MI
|
Pus
|
M
|
blaCTX-M
|
|
63
Chpaitr 03 : Résultats et discussion
Discussion
Depuis plusieurs années, le nombre de bactéries
résistantes aux antibiotiques est en constante augmentation,
principalement en milieu hospitalier. La menace la plus importante à
l'heure actuelle est observée chez les entérobactéries
(Armand-Lefévre. 2017) où on assiste à l'émergence
d'un grand nombre d'entérobactéries productrices de
bêta-lactamases à spectre élargi (BLSE).
Cette étude s'est déroulée du 01/01/2013
au 30/4/2017, période au cours de laquelle le recueil des données
de différents types de prélèvements de patients
hospitalisés des différents services au CHU de Sétif et de
patients à titre externe a permis d'isoler 2421 souches
d'entérobactéries. La souche E. coli est l'espèce
la plus fréquemment isolée avec 49.91%, suivi par K.
pneumoniae (19.78%) et P. mirabilis (8.92%). Des études
similaires confirme que E. coli est le pathogène le plus
répandu (68,2 %), suivi de Klebsiella spp. (Thibaut et
al. 2017). Le même constat a été fait au Maroc en
2005 où E. coli est également classé le germe le
plus prédominant avec 44.7 % suivie par Klebsiella sp.
(Sekhsokh et al. 2008). Toutefois, en France, E. coli
représente la première cause d'infection pathologique mais avec
un pourcentage de 62% (Diamantis et al. 2017) ou de 93% (Ménard
et al.2017).
La prévalence des entérobactéries
isolées des infections nosocomiales est plus élevée par
rapport à celles isolées des infections communautaires. Un taux
plus élevé (74%) est isolée des infections
hospitalière lors d'une enquête faite au CHU de Batna (Benammar et
al. 2017). Cependant, ce pourcentage est inversé quand il
s'agit d'infections urinaire où la fréquence d'isolement des
entérobactéries provient plus des malades à titre externe
(Lahlou et al. 2008 ; Hailaji et al. 2016). Il est
également noté que les patients de sexe féminin sont un
facteur de risque (Nadmi et al. 2010 ; Thibaut et al. 2017 ;
Rakotovao-Ravahatra et al. 2017). Les infections
particulièrement urinaires sont plus fréquentes chez les femmes
avec un pourcentage élevé comparativement aux hommes. Par
ailleurs, la majorité de la population semble infectée. Selon les
données de la répartition des entérobactéries
suivant l'âge des patients. Les différentes tranches d'âge
représentent des taux de colonisation plus au moins proches. Des
études ailleurs soulèvent des constats mitigés. Certaines,
supposent que les jeunes sont les plus infectés par rapport au vieux de
plus de 65 ans (Ben Ayed et al. 2017). Par contre d'autres auteurs
suggèrent que les personnes âgées sont les plus
infectés, révélant ainsi que la démographie en
termes d'âge, les traditions et les cultures de chaque région sont
pratiquement les causes de ces différences.
64
Chpaitr 03 : Résultats et discussion
Les antibiotiques les plus utilisées dans les
infections à entérobactéries sont les â-lactamines.
L'étude de la sensibilité des entérobactéries aux
antibiotiques atteste d'un taux très élevé de
résistance aux bêtalactamines. Les résistances maximales
sont notées pour l'amoxiclline où (98.40%) des souches sont
résistantes, suivie par la céfazoline (88.90%) et
l'aztréonam (52.79%). La résistance aux C3G est également
élevée, elle est de (40.72% et (30.10%) respectivement pour la
cefotaxime et la ceftazidime. Notre étude démontre à
l'instar de nombreux autres travaux que la résistance des
entérobactéries est un phénomène grandissant. La
mauvaise utilisation des antibiotiques, l'automédication, la
recrudescence de la vente illicite des antibiotiques, la mauvaise conservation
des antibiotiques, les erreurs de prescription pourraient expliquer en partie
l'évolution de la fréquence des résistances.
Une sensibilité remarquable de l'ampicilline (80%) est
observée, la restauration de l'activité de cet antibiotique est
réapparue suite à son non utilisation au niveau communautaire
(sauf en dermatologie) qu'hospitalier. Concernant les aminosides, notre
étude révèle une sensibilité élevée
(>90%) à la gentamicine et la tobramycine. Ces taux de
sensibilité sont bien encadrés par les travaux antérieurs
(Tiouit. 2001). Notre étude révèle un faible taux de
sensibilité au cotrimoxazole (20.38%). Cette constatation a
été faite par certains auteurs au Maghreb où ils ont
trouvé (29,94%) de souches sensibles. Par contre d'autres auteurs ont
trouvé des pourcentages plus élevés de souches sensibles
au cotrimoxazole (Weber. 1993). La résistance à l'AMC
présente un taux plus faible que celui trouvé dans l'étude
de Diamantis et al. 2017. Des taux faibles mais non négligeable
aux C4G (8.39%) constitue un signe d'alerte et l'apparition de
résistance combinée avec d'autres antibiotiques est un
échec thérapeutique. Alors que, la FOX, IPM, ETP et AN restent
les antibiotiques les plus efficaces, malgré la présence d'une
légère résistance respectivement de 4.82, 4.71, 4.36 et
2.35%. Ces résultats sont conformes à certains travaux (Benammar
et al. 2017).
Les bactéries productrices de BLSE constituent une
préoccupation majeure en milieu hospitalier en raison de leur diffusion
épidémique et de leur multirésistance aux antibiotiques.
En parallèle, le milieu communautaire joue un rôle primordial dans
leur propagation. En effet, les BLSE sont retrouvées chez une vaste
proportion de bacilles à Gram négatif, et les
entérobactéries représentent les germes les plus
incriminés (Gniadkowski. 2001).
Dans cette étude s'étalant sur quatre mois,
l'incidence des souches d'entérobactéries productrices de BLSE
est de 35%.Cette prévalence correspond à peu près à
celle retrouvée dans certaines études nationales, en particulier
celle faite à Annaba (31.40 %) (Nedjai et al.
65
Chpaitr 03 : Résultats et discussion
2013) et à Tlemcen (39.22%) (Baba Ahmed-Kazi Tani et
al. 2013); mais assez différente de celles rapportées
dans d'autres pays, tels au Japon (6,4 %) (Luvsansharav et al. 2011)
ou en Belgique (6.6%) (Rodriguez-Villa-Lobos et al. 2011). D'autres
études récentes rapportent des taux d'EBLSE variant de 6 à
64 % selon les zones géographiques (Sood et Gupta. 2012 ; Kizilca et
al. 2012) ces taux ont augmenté de 16 % en 2005 à 44 %
en 2010 (Bourigault et al. 2013).
Les espèces d'entérobactéries les plus
productrices de BLSE sont E. coli et K. pneumoniae avec des
proportions de (41.07%) et (39.29%) respectivement, suivie par E.
cloacae (7.14%) ceci corroborent les résultats d'autres
études (Neulier et al. 2014). Par contre, les autres
entérobactéries telles que M. morganii, S. marcescens, E.
aerogenes et C. freundii sont faiblement
représentées. Cependant, des taux plus élevés de la
prévalence de Klebsiella productrices de BLSE sont
détectées en Amérique du Sud (45,4% à 51,9 %)
(Villegas et al. 2008) et en Arabie Saoudite (55%) (Al-Agamy et
al. 2009). En Iran, la prévalence des K. pneumoniae
productrices de BLSE était très élevée, soit
un taux alarmant de 72.1% (Feizabadi et al. 2010).Les résultats
des taux d'E. cloacae (7.14%) productrices de BLSE rapportés
dans cette étude sont plus faibles que les proportions trouvées
dans les pays africains où des taux élevés ont
été rapporté, soit (18.5%) au Mali (Duval et al. 2009).
La plupart des EBLSE (75%) sont isolées des infections
hospitalières, donnant ainsi une haute prévalence que confirme
l'étude nationale faite au CHU de Batna (74%) (Benammar et al. 2017).
Pratiquement, la prévalence globale de la production
de BLSE a varié considérablement selon les zones
géographiques des pays et dans différents structures
hospitalières. Durant les quatre mois d'étude, les souches EBLSE
ont été retrouvées dans tous les types de services.
Cependant, certaines spécialités sont apparues plus
concernées, tels que les services des maladies infectieuses, de
néphrologie et de pédiatrie. Dans ces services, les
bactéries sont soumises à une pression élevée des
antibiotiques. Bien plus, plusieurs de ces patients sont
particulièrement vulnérables aux infections suite à une
hospitalisation prolongée et après exposition à des
dispositifs invasifs (sonde urinaire, cathéters veineux, ou tube
endotrachéal). En effet, les patients hospitalisés au sein des
unités de soins intensifs présentent plus de risques à
contracter une BLSE (Stürenburg et Dietrich. 2003).En
général, les services de réanimation sont
fréquemment considérés comme des foyers d'origine pour la
dissémination des bactéries productrices de BLSE en causant de
nombreuses épidémies (Rodriguez-
66
Chpaitr 03 : Résultats et discussion
Villalobos et Struelens. 2006), ce qui est pratiquement le
contraire des résultats de cette étude. Les services de
réanimation ont mis des stratégies pour limiter cette
dissémination.
En effet, un nombre significativement important des
prélèvements recensés sont les urines (32%)
prélevées surtout chez les patients hospitalisés. Ce
résultat confirme que la majorité des souches BLSE proviennent
des urines (Canton et al. 2008 ; Ebongue et al. 2015).
Dans notre étude, le sexe masculin (52%) est
retrouvé comme facteur de risque. Ces résultats sont en accord
avec ceux rapportés par certaines études (Colodner et
al. 2004 ; Gupta et Datta. 2007). Néanmoins, d'autres ont
conclu à un risque plus élevé pour les femmes
(Rodriguez-Bano et al. 2008b ; Nedjai et al. 2013).
Les résultats de l'antibiogramme ont montré que
les souches présentent une résistance totale de 100% à
l'ampicilline, l'amoxicilline (aminopénicillines) et la ticarcilline
(carboxypénicillines). Ainsi, les souches sont résistantes
à 100 % aux céphalosporines de 1ére
génération (C1G) la céfazoline et la céfalotine,
aux G la céfuroxime, aux céphalosporines de
3émegénération (CAZ, CTX), à la
Céfepime (C4G) et l'aztréonam (monobactame).Concernant les
résistances des â-lactamines associé à l'acide
clavulanique, une résistance élevée à AMC
(amoxicilline/ac clavulanique) de 65% est notée. Contrairement à
l'association PTB (Pipéracilline/Tazobactam) qui est plus actif avec
seulement 23% de résistance.
La résistance aux C3G a été
signalée dans plusieurs pays africains, Sénégal,
Centre-Afrique, Maroc et Algérie (Frank et al. 2006;
Ruppé et al. 2009; Bourjilat et al. 2011; Ahmed et
al. 2012) avec des taux comparables à ceux trouvés.
Cependant, ils sont plus élevés que ceux trouvés en
Europe, aux USA et en Chine (Goossens et Grabein. 2005; Hirakata et
al. 2005; Winokur et al. 2001) et en Iran (Feizabadi et
al. 2010).
Les autres familles d'antibiotiques ont donné une
très forte résistance à l'association sulfamides SXT (75%)
; aux quinolones (70% pour la CIP et 35% pour la LVX) de même qu'aux
aminosides (61% pour la GN et 63% pour la TOB).
Récemment, un taux beaucoup plus important a
été retrouvé dans une étude algérienne,
où le pourcentage de la résistance de souches EBLSE est de 100%
de résistance à la majorité des antibiotiquesC3G
testés, gentamicine et tobramycine (Touati et al. 2012).
Les molécules les plus actives ont été
l'imipénème (95 %), l'amikacine (85 %) et la fosfomycine (80 %).
La céfoxitine n'est active que chez 70 % des souches.
Il est actuellement prouvé que l'utilisation des
antibiotiques, notamment les C3G dans un but thérapeutique est le
facteur de risque le plus important dans le développement de
résistances
67
Chpaitr 03 : Résultats et discussion
bactériennes (Rubin et Samore. 2002). Cette pratique
est devenue un problème majeur de santé public. Ainsi, des
résistances croisées sont observées avec les aminosides,
fluoroquinolones, quinolones, et les triméthoprimes-sulfamethoxazoles.
Ceci est en rapport avec l'utilisation abusive d'antibiotiques à large
spectre (pénicillines, céphalosporines, fluoroquinolones,
aminosides).
L'analyse du profil plasmidique des 56 souches BLSE
étudiées a montré que nos bactéries
possèdent de 1 à 4 types différents de plasmide.
Cependant, cette diversité est responsable de la résistance et la
co-résistance aux antibiotiques. Dans d'autres études
(Mendonça et al. 2009; Tandé et al. 2009, Ahmed
et al. 2012) la co-résistance serait due à la
présence chez ces souches de grands plasmides qui portent en plus des
gènes bêtalactamases d'autres gènes de
résistances.
La séparation par électrophorèse des
produits PCR-Multiplex de 33 souches d'entérobactéries
isolées a permis de mettre en évidence la production de trois
types de BLSE, avec une nette prédominance de BLSE de type CTX-M, soit
88%, suivie de SHV 45% et TEM 18%.
Quoique les prévalences soient relativement
différentes, le CTX-M est retrouvé majoritaire dans
plusieurs études, notamment au Cameroun où les BLSE de type CTX-M
est de 97 %, en Afrique du Sud (95 %), en Guinée-Bissau (94,8 %), au
Niger (91 %), en Algérie (76 %) au Burkina-Faso (65,49 %), en France (45
%) et en Allemagne (85%) (Giraud-Morin et Fosse. 2008; Peirano et al.
2011; Woerther et al., 2011; Isendahl et al., 2012; Lonchel
et al., 2012; Nedjai et al., 2013; Zongo et al. 2015
; Pietsch et al. 2017).
Le CTX-M appartient aux Bêtalactamases de classe A,
décrit pour la première fois en Europe Occidentale et dès
lors retrouvé un peu partout dans le monde (Bradford, 2001a; Bonnet,
2004; Paterson et Bonomo, 2005).Dans cette étude, la BLSE CTX-M a
dominé chez Escherichia (69%); cette observation est
également faite par plusieurs auteurs (Metuor Dabire. 2014; Zongo et
al. 2015).
Dans cette étude, 33 isolats producteurs de BLSE sont
détectés. Les souches BLSE sont toutes multirésistantes.
Les mêmes résultats ont été rapportés dans
l'étude d'Ahoyo et al. 2007, les souches BLSE porteuses de
gènes CTX-M, sont porteuses d'autres gènes de résistance.
Des études antérieures ont montré que les plasmides qui
interviennent dans la résistance aux BLSE peuvent transporter plus d'un
gène bêta-lactamase comme CTX-M, SHV et TEM, ce qui peut
potentialiser l'action des bêtalactamases. Ces gènes sont
portés par le même plasmide conjuguatif et peuvent être
transférés dans d'autres souches (Ahoyo et al. 2007).
Les bêtalactamases de type TEM sont décrites depuis assez
longtemps. Aujourd'hui,
68
Chpaitr 03 : Résultats et discussion
plus de100 dérivés de bêta-lactamase de
type TEM sont décrits et beaucoup d'entre eux sont des BLSE.
Contrairement aux bêtalactamases de type TEM, le groupe des
bêtalactamases CTX-Ma connu une évolution rapide ces
dernières années.
CONCLUSION
69
Conclusion
Conclusion et Perspectives
La résistance aux antibiotiques, surtout par
production de BLSE est une réalité en Algérie et partout
dans le monde et leur large dissémination risque de compromettre le
traitement des infections courantes dans la communauté comme dans les
hôpitaux.
Actuellement, la diffusion de ces souches
multi-résistantes, d'entérobactéries productrices de BLSE
et de céphalosporinase plasmidique constitue une menace de santé
publique, réduisant de manière importante les alternatives
thérapeutiques pour le traitement des infections
sévères.
Ce travail, réalisé de façon
rétrospective sur une période de quatre ans et quatre mois de
2013 jusqu'au 30/04/2017, a pour objectif de définir le profil
épidémiologique et moléculaire de souches isolées
de patients à titre externe et de patients hospitalisés aux
différents services du CHU de Sétif.
Durant cette période, 2421 souches
d'entérobactéries ont été isolées dont
E. coli est la souche la plus fréquente (n= 1160),
suivie par K. pneumoniae (n=479) et P.mirabilis (n=216). Ces
souches proviennent majoritairement du milieu hospitalier (57.71 %) et semblent
coloniser les adultes plutôt que les enfants où le sexe
féminin représente un facteur de risque.
Le profil de résistance de ces
entérobactéries atteste d'un taux très élevé
de résistance aux bétalactamines. Les valeurs maximales sont
notées pour l'amoxiclline où 98.40% des
souches sont résistantes et les plus faibles
résistances sont notées pour les
carbapénèmes
(l'imipeneme et l'ertapeneme) et la cefoxitine.
Les 56 entérobactéries productrices de BLSE
isolées durant l'année 2017 représentent 35% de la
totalité des entérobactéries isolées pendant cette
période. Elles sont réparties comme suit : 23 E. coli,
22 K. pneumoniae, 4 E. cloacae, 3 M. morganii, 2
S. marcescens, 1 E. aerogenes et 1 C. freundii.
Elles sont plutôt issues d'infections nosocomiales (75%)
que communautaires (25%). Les services les plus concernés sont
l'infectieux et la néphrologie et les prélèvements les
plus incriminés sont les urines et le pus.
Ces souches EBLSE ont des résistances très
élevées (100%) à la majorité des â-lactamines
(AMX, AMP, TIC, C3G, C4G et l'aztréonam).Alors que l'IMP, ERT, MER, AMK
et FOS restent les antibiotiques les plus actifs.
Le profil plasmidique des souches EBLSE confirme qu'elles
peuvent héberger de 1 à 4 types de plasmides.
70
Conclusion
La caractérisation des gènes des BLSE
effectuée sur l'ADN (plasmidique ou chromosomique) de 33 souches par
l'amplification multiple des gènes (CTX-M, TEM et SHV) a permis
l'obtention d'une prévalence de CTX-M (87.87%) suivie de SHV (42.42%) et
de TEM (27.27%). Toutefois, cinq souches (4 K. pneumoniae et 1 E.
aerogenes) semblent porter les 3 gènes de résistance en
même temps, indiquant la gravité de ce phénomène.
En effet, il est nécessaire de prendre des mesures de
prévention pour éviter toute infection par ces germes redoutables
qui sont une cause importante de morbidité et de mortalité au
niveau mondiale. Pour ce faire il faut améliorer l'observance de
l'hygiène des mains, diminuer la charge de travail par l'augmentation du
nombre des soignants et réduire la proportion de patients recevant des
antibiotiques à l'admission et la durée de traitement.
La lutte contre l'émergence et la diffusion de ces
souches multirésistantes aux antibiotiques passe par une meilleure et
moindre utilisation des antibiotiques. En effet, une politique
d'antibiothérapie justifiée et/ou d'une restriction dans la
prescription des céphalosporines de troisième
génération et même de toutes les céphalosporines
conduisent à une diminution significative de la fréquence des
BLSE.
Enfin, les scientifiques à l'heure actuelle cherchent
des alternatives qui aide à contrôler en
quelque sorte la résistance aux antibiotiques ou la
réguler une bonne fois pour toute :
- L'amélioration de la structure des anciens
antibiotiques,
- L'association avec des inhibiteurs des bêtalactamases
d'une structure améliorée,
- L'inhibition du transfert des plasmides,
- L'utilisation des peptides antimicrobiens (PAMs),
- Le contrôle de l'activité du
riborégulateur,
- L'utilisation de la nanotechnologie,
- L'utilisation des ARN interférents et
- La phagothérapie.
71
Conclusion
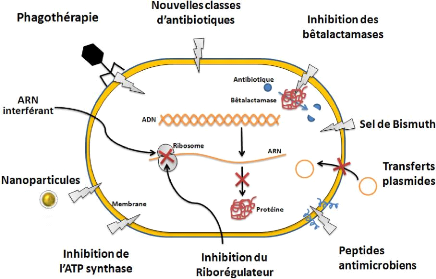
Figure 31. Stratégies et les cibles
bactériennes utilisées pour lutter contre la résistance
aux
antibiotiques (Lemaoui et al. 2017).
REFERENCES
BIBLIOGRAPHIQUE
72
Références bibliographiques
Références bibliographiques
· Abbas M, Cherkaoui A, Fankhauser C, Schrenzel
J et Harbarth S. Carbapénémases ; implications cliniques
et épidémiologique pour la Suisse. Méd Suisse
338: 882-884. 2012.
· Ahmed Z.B., Ayad A., Mesli E., Messai Y.,
Bakour R. et Drissi M. CTX-M-15 extended-spectrum beta-lactamases in
Enterobacteriaceae in the intensive care unit of Tlemcen Hospital,
Algeria. East Mediterr Health J, 18, 382-6.
2012.
· Ahoyo A.T., Baba-Moussa L., Anago A.E., Avogbe
P., Missihoun T.D., Loko F., Prevost G., Sanni A. and Dramane K.
[Incidence of infections dues to Escherichia coli strains
producing extended spectrum betalactamase, in the Zou/Collines Hospital Centre
(CHDZ/C) in Benin]. Med Mal Infect, 37, 746-52.
2007.
· Aibinu I.E., Pfeider Y., Ogunsola F., Odugbemi
T., Koenig W. et Gheberemedhin B.. Emergence of bêta-lactamases
OXA-10, VEB-1 and CMY in Providencia spp. from Nigeria. J
Antimicrob Chemother, 66, 1931-1932.
2011.
· Aktas Z, Kayacan CB and Schneider I.
Carbapenem- hydrolyzing oxacillinase OXA-48, persists in Istanbul, Turkey.
Chemother; 54:101-6. 2008.
· Al-Agamy M.H., Shibl A.M. and Tawfik A.F.
Prevalence and molecular characterization of extended-spectrum
beta-lactamase-producing Klebsiella pneumoniae in Riyadh, Saudi
Arabia. Ann Saudi Med. 29(4): 253-257. 2009.
· Ambler R.P., Coulson A.F.W., Frére
J.M., Ghuysen J.M., Joris B., Forsman M., Levesque R.C., Tiraby A. and Waley
S.G. A standard numbering scheme for the class A f3-lactamases.
Biochem. J. 276: 269-270. 1991.
· Ambler, R.P. The structure of
f3-lactamases. Phil Trans R Soc Lond Biol Sci. 289: 321331.
1980.
· Andersson D.I. and Hughes D.
Antibiotic resistance and its cost: is it possible to reverse
resistance? Nat Rev Microbial; 8:260-71. 2010.
· Anonyme. 2014. Antimicrobial
Resistance: Global report on surveillance 2014. FRANCE: (WHO) World Health
Organization.
· Arlet G. et Philippon, A. Les
nouvelles f3-lactamases à l'aube du troisième millénaire.
Rev Franç Lab. 352 : 41-55. 2003.
73
Références bibliographiques
· Armand-Lefèvre L. Antibiotic
resistance in the community: Myth or reality? The threat of ESBL producing
enterobacteria doi. j.antinf. 01.004. 2017.
· Asensio A., Oliver A., Gonzalez-Diego P.,
Baquero F., Perez-Diaz J.C., Ros P., Cobo J., Palacios M., Lasheras D. and
Canton R. Outbreak of a multiresistant Klebsiella pneumoniae
strain in an intensive care unit: antibiotic use as risk factor for
colonization and infection. Clin Infect Dis. 30: 55-60.
2000.
· Avril J.L., Denis F., Dabernat H. et Monteil
H. Bactériologie Clinique. 3éme
édition. Ellipses. Paris. 171-172. 2000.
· Baba Ahmed-Kazi Tani Z., Decré D.,
Genel N., Boucherit-Otmani Z., Arlet G. and Drissi, M. Molecular and
Epidemiological Characterization of Enterobacterial Multidrug65 Resistant
Strains in Tlemcen Hospital (Algeria) (2008-2010). Microbial Drug
Resistance. 19(3): 185-190. 2013.
· Bandoh K., Muto Y., Watanabe K., Katoh N. and
Ueno K. Biochemical properties and purification of metallo-B-lactamase
from Bacteroïdes fragiIis. Antimicrob. Agents Chemother.
35(2): 371-372. 1991.
· Barrial K. et Scotet J.
Classification raisonnée des â-lactamases chez les
bacilles Gram négatif. Perspective d'évolution. Tig.
Bact. 3-10. 2006.
· Bebrone C. Metallo-beta-lactamases
(classification, activity, genetic organization, structure, zinc coordination)
and their superfamily. Biochem Pharmacol. 74: 1686-701.
2007.
· Belmonte O., Drouet D., Alba J., Moiton M.-P.,
Kuli B., Lugagne-Delpon N., Mourlan C. et Jaffar-Bandjee M.C.
Evolution de la résistance des entérobactéries
aux antibiotiques sur l'île de la Réunion : émergence des
bêta-lactamases à spectre élargi. Pathologie Biologie
58, 18-24. 2010.
· Ben Ayed H., Gargouri M., Ben Jemaa T., Ben
Yahia M., Smaoui F., Marrakchi C., Koubaa M. et Ben Jemaa M. Facteurs
prédisposant d'infection urinaire à bactéries
multi-résistantes aux antibiotiques. Médecine et Maladies
Infectieuses, Vol 47, Issue 4, p S30. Juin 2017.
· Ben Redjeb, S., Ben Hassen, A., Hammami, A.,
and Kechrid, A. Epidémiologie des résistances
bactériennes en Tunisie. «Résistance aux
antibiotiques». Press. Méd. 1-5. 2000.
74
Références bibliographiques
· Benammar S., Benmehidi M., Bouziane F.,
Boukhalfa S. et Makhloufi M. Qu'en est-il de la résistance des
entérobactéries aux C3G en ville et à l'hôpital ?
Laboratoire de Microbiologie du CHU Batna, Algérie. J. Med Mal;
03: 071. 2017.
· Bennett P.M. Plasmid encoded
antibiotic resistance: acquisition and transfer of antibiotic resistance genes
in bacteria. Br J Pharmacol; 153 Suppl 1:S347-57.
2008.
· Béreaud J. Le technicien
d'analyse biologique guide et pratique. Edition médicales
internationales. Londres-Paris-new York. 871-984. 2001.
· Bialek-Davenet S., Marcon E., Leflon-Guibout
V., Lavigne J.P., Bert F., Moreau R. and Nicolas-Chanoine M.H. In
vitro selection of ram R and sox R mutants overexpressing.
2011.
· Bonnet R. Growing group of
extended-spectrum-beta-lactamases: the CTX-M enzymes. Antimicrob Agents
Chemother. 48: 1-14. 2004.
· Bonnet R., Sampaio J.L., Chanal C., Sirot D.,
De Champs C., Viallard J.L., Labia R. and Sirot J. A novel class A
extended-spectrum â-lactamase (BES-1) in Serratia marcescens
isolated in Brazil. Antimicrob. Agents Chemother. 44: 3061-3068.
2000a.
· Boulahbal F. Manuel de microbilogie.
2éme édition. Office des publications universitaires.
Algérie. 20-273. 2010.
· Bourdjilat F., Bouchrif B., Dersi N., Perrier
Gros-Claude J.D., Amarouche, H. et Timinouni M. Emergence of
extended-spectrum beta-lactamase-producing Escherichia coli in
community-acquired urinary infections in Casablanca, Morocco. J Infect Dev
Ctries 5, 850-855. 2011.
· Bourigault C., Corvec S., Bemer P., Juvin
M.E., Guillouzouic A., Crémet L., Reynaud A., Leprince C. et Lepelletier
D. Impact de l'augmentation de l'incidence des
entérobactéries productrices de bêta-lactamases à
spectre étendu (EBLSE) sur l'application des précautions
complémentaires dans un centre hospitalier universitaire. Pathologie
Biologie, Volume 61, Issue 5, Pages 217-222. October
2013.
· Boyer A., Clouzea B., Mzali f., Hilbert G. et
Gruson D. Les nouvelles résistances aux antibiotiques.
jarca. 2011.
· Bradford P. A. Extended-spectrum
beta-lactamases in the 21st century: characterization, epidemiology, and
detection of this important resistance threat. Clin Microbiol Rev,
14, 933-951. 2001a.
75
Références bibliographiques
· Bradford P.A. Automated thermal
cycling is superior to traditional methods for nucleotide sequencing of
blaSHV genes. Antimicrob. Agents Chemother. 43: 2960-2963.
1999.
· Bradford P.A. What's New in
beta-lactamases? Curr Infect Dis Rep, 3, 13-19.
2001b.
· Brisse S. and Verhoef J. Phylogenetic
diversity of Klebsiella pneumoniae and Klebsiella oxytoca
clinical isolates revealed by randomly amplified polymorphic DNA, gyrA
and parC genes sequencing and automated ribotyping. Int J
Syst Evol Microbiol. 51: 915-924. 2001.
· Bush K. and Jacoby G.A. Updated
functional classification of beta-lactamases. Antimicrob Agents Chemother;
54:969-76. 2010.
· Bush K. Extended-spectrum
beta-lactamases in North America, 1987-2006. Clin Microbiol Infect, 14
Suppl 1, 134-43. 2008.
· Bush K. Updated functional
classification of b-lactamases. Antimicrob Agents Chemother. p
54:969-76. 2010.
· Bush K., Jacoby G.A. and Medeiros A.A.
A functional classification scheme for Blactamases and its correlation
with molecular structure. Antimicrob Agents Chemother. 39(6):
1211-1233. 1995.
· Buyck J.M., Luyckx C., Ioli M.G.G., Krause
K.M., Nichols W.W., Tulkens P.M. and Bambeke F.V. Pharmacodynamics of
ceftazidime/avibactam against extracellular and intracellular forms of
Pseudomonas aeruginosa. JAC. 16-94. 2016.
· Canton R., Novais A., Valverde A., Machado E.,
Peixe L., Baquero F. et Coque T. M. Prevalence and spread of
extended-spectrum beta-lactamase-producing Enterobacteriaceae in
Europe. Clin Microbiol Infect, 14 Suppl 1, 144-53.
2008.
· Carattoli A. Resistance Plasmid
Families in Enterobacteriaceae Antimicrob. Agents Chemother; 53:
2227-2238. 2009.
· Carrer A., Poirel L., Yilmaz M.
Spread of OXA-48- encoding plasmid in Turkey and beyond. Journal
Antimicrob Agents Chemother, 54: 1369-73. 2010.
· CA-SFM 2013. Recommandations Paris
2013.
· CA-SFM/EUCAST 2014. Recommandations Paris
2014.
· Castanheira M, Deshpande LM and Mathai
D. Early dissemination of NDM-1- and OXA-181-producing
Enterobacteriaceae in Indian hospitals: report from the SENTRY
76
Références bibliographiques
Antimicrobial Surveillance Program, 2006-2007. Antimicrob
Agents Chemother; 55: 1274-8. 2011.
· Cavallo J.D., Faber R., Jehl F., Rapp C. et
Garrabé E. Bétalactamines. Mal. inf. 1: 129-20.
2004.
· Chopra I., O'Neill A. and Miller K.
The role of mutators in the emergence of antibiotic-resistant
bacteria. Drug Resist Updates. 6: 137-145. 2003.
· Chow J.W., Fine M.J., Shlaes D.M., Quinn J.P.,
Hooper D.C., Johnson M.P., Ramphal R., Wagener M.M., Miyashiro D.K. and Yu V.L.
Enterobacter bacteremia: clinical features and emergence of
antibiotic resistance during therapy. Ann Intern Med. 115(8): 585-590.
1991.
· Colodner R., Rock W., Chazan B., Keller N.,
Guy N., Sakran W. and Raz R. Risk Factors for development of
Extended-Spectrum Beta-lactamase-Producing Bacteria in Nonhospitalized
Patients. Eur J Microbiol Infect Dis. 23: 163-167.
2004.
· Cuzon G, Ouanich J, Gondret R, Naas T et Nordmann
P. Outbreak of OXA-48-positive carbapenem-resistant Klebsiella
pneumoniae isolates in France. Antimicrob Agents Chemother; 55:
2420-3. 2011.
· Datta N. and Kontomichalou, P.
Penicillinase synthesis controlled by infectious factors in
Enterobacteriaceae. Nature 208: 239-241. 1965.
· Davies J. Origins, acquisition and
dissemination of antibiotic resistance determinants.207:15-27.
1997.
· Dbaibo G.S. Old and new targets of
antibacterial therapy. Leb. Med. J. 48: 177-181.
2000.
· Decoster A.
Entérobactéries (internet). FLM. 1-16. 2005.
Available from:
http://anne.decoster.free.fr/btelechar/bpoly/enteroba05.pdf.
· Decousser J.W., Poirel L. and Nordmann P.
"Characterization of a chromosomally encoded extended-spectrum class A
beta-lactamase from Kluyvera cryocrescens". Antimicrob. Agents
Chemother. 45(12): 3595-3598. 2001.
· Delrras C. Microbiologie pratique
pour le laboratoire d'analyses ou de contrôle sanitaire. Edition
médicales internationales. Lavoisier. Paris. 247 p.
2007.
· Diamantis L, Beziz D., Bonnet-zamponi D.,
Armaingaud D., Ombandza E., Pitsch A., Aufaure S., Tangre L, F. Tubach F et
Gauzit R. Antibiorésistance des entérobactéries
isolées d'ECBU réalisés dans huit établissements
pour personnes âgées dépendantes (EHPAD) en France.
j.Med Mal ; 03.070.2017.
77
Références bibliographiques
· Domingues S, Da Silva G.J and Nielsen K.M.
Integrons: vehicles and pathways for horizontal dissemination in
bacteria. Mob Genent Elements; 2(5): 211-223.
2012.
· Drieux L., Brossier F., Sougakoff W. and
Jarlier V. Phenotypic detection of extended-spectrum beta-lactamase
production in Enterobacteriaceae: review and bench guide. Clin Microbiol
Infect. 14: 90-103. 2008.
· Duval V., Maiga I., Maiga A., Guillard T.,
Brasme L., Forte D., Madoux J., Vernet-Garnier V. and De Champs C.
High Prevalence of CTX-M-Type Bêta-Lactamases among Clinical
Isolates of Enterobacteriaceae in Bamako, Mali. Antimicrobial
Agents and Chemotherapy 53, 4957-4958.
2009.
· Ebongue C.O., Tsiazok M.D., Nda Mefo'o J.P.,
Ngaba G.P., Beyiha G. et Adiogo D. Evolution of antibiotic resistance
of Enterobacteriaceae isolated at the Douala General Hospital from 2005 to
2012. Pan Afr Med J; 20: 227. 2015.
· El Salabi A, Walsh T.R and Chouchani C.
Extended spectrum -lactamases, Carbapenemases and mobile genetic
elements responsible for antibiotics resistance in Gram-negative bacteria.
Crit Rev Microbiol; 39: 113-122. 2013.
· Elhani D. Les bêta-lactamases
à spectre étendu : le défi s'accentue. Ann Biol
Clin. 70 (2) ; 117-40. 2012.
· Farmer J.J, Boatwright K.D and Janda J.M.
Enterobacteriaceae: Introduction and identification. Manual of
Clinical microbiology. Washington, DC, USA: ASM press. 9th ed: 649-669.
2007.
· Feizabadi M.M., Mahamadi-Yeganeh S.,
Mirsalehian A., Mirafshar S.M., Mahboobi M., Nili F. and Yadegarinia D.
Genetic characterization of ESBL producing strains of Klebsiella
pneumoniae from Tehran hospitals. J Infect Dev Ctries. 4:
609-615. 2010.
· Follath F., Costa E., Thommen A., Frei R.,
Burdeska A. and Meyer J. Clinical consequences of development of
resistance to third generation cephalosporins. Eur J Clin Microbiol.
6(4): 446-450. 1987.
· Frank T., Arlet G., Gautier V.A.T. et Bercion
R. Extended-spectrum â-Lactamase- producing
Enterobacteriaceae, Central African Republic. Emerging Infectious
Diseases, 12, 863-865. 2006.
· Fritzh K., Bottger E.C., Zinkernagel M.,
Haller O., Eckert J. et Deplazes P. Manuel de poche de microbiologie.
293-764. 2008.
78
Références bibliographiques
· Gangoue-pieboji J., Bedenic B., Koulla-shiro
S., Randegger C., Adiogo D., Ngassam P., Ndumbe P., Hacheler H.
Extended-spectrum â-lactamase producing enterobacteriacea
in Yaounde , Cameroon. Journal Clin Micr. 43: 3273-3277.
2005.
· Garrity M.G., D. J. Brenner, N.R. Krieg et
J.T. Staley. BERGEY'S MANUAL_ OF Systematic Bacteriology,
Second Edition, Volume Two, The Proteobacteria
Part B, the Gammaproteobacteria.
2005.
· Gaudy C. et Buxeraud J. Antibiotiques
: Pharmacologie et thérapeutique. 16-17. 2005.
· Gazengel J.M. Le préparateur
en pharmacie. Technique et documentation 19-21. 2007.
· Georgopapadakou N.H.
Penicillin-binding proteins and bacterial resistance to
â-lactams. Antimicrob. Agents Chemother. 37: 2045-2053.
1993.
· Giraud-Morin C. and Fosse T. [Recent
evolution and characterization of extended-spectrum beta-lactamase producing
enterobacteria in the CHU of Nice (2005-2007)]. Pathol Biol
(Paris), 56, 417-23. 2008.
· Gniadkowski M. Evolution and
epidemiology of extended spectrum beta-lactamases and ESBL producing
micro-organisms. Clin. Microbial infect. 7: 557 -608.
2001.
· Goldstein F.W., Pean Y. and Gertner J.
Resistance to ceftriaxone and other betalactams in bacteria isolated
in the community. The Vigil'Roc Study Group. Antimicrob. Agents
Chemother. 39(11): 2516-2519. 1995.
· Goossens H. and Grabein B. Prevalence
and antimicrobial susceptibility data for extended-spectrum b-lactamase and
AmpC-producing Enterobacteriaceae from the MYSTIC Program in Europe
and the United States (1997-2004). Diagnostic Microbiology and Infectious
Disease 53, 257-264. 2005.
· Grall N. et Muller-Serieys C.
Carbapénèmes. Mal. inf. 1-16.
2013.
· Grosjean J.C. Darchambaud M. et Pasquier C.
Bacteriologie et virology pratique. 2éme édition. Boeck.
Paris. 131-133. 2011.
· Gülmez D, Woodford N and Palepou
MF. Carbapenem-resistant Escherichia coli and Klebsiella
pneumoniae isolates from Turkey with OXA-48-like carbapenemases and outer
membrane protein loss. Int J Antimicrob Agents; 31:523-6.
2008.
· Gupta V. and Datta, P.
Extended-spectrum beta-lactamases (ESBL) in community isolates from
North India: frequency and predisposing factors. Int J Infect Dis. 11:
88-89. 2007.
79
Références bibliographiques
· Haeggman S., Löfdahl S., Paauw A.,
Verhoef J. and Brisse S. Diversity and evolution of the class A
chromosomal beta-lactamase gene in Klebsiella pneumoniae.
Antimicrob. Agents Chemother. 48: 2400-2408. 2004.
· Hailaji A., Ould Salema M.L. et Ghaber S.M.
La sensibilité aux antibiotiques des bactéries
uropathogènes dans la ville de Nouakchott -- Mauritanie N.S.M.
Journal progrès en urologie ; 26, 346-352.
2016.
· Hall L.M., Livermore D.M., Gur D., Akova M.
and Akalin H.E. OXA-11, an extended-spectrum variant of OXA-10 (PSE-2)
â-lactamase from Pseudomonas aeruginosa. Antimicrob. Agents
Chemother. 37(8): 1637-1644. 1993.
· Hammond D.S., Schooneveldt J.M., Nimmo G.R.,
Huygens F. and Giffard P.M. blaSHV genes in Klebsiella
pneumoniae: different allele distributions are associated with different
paromoters within individual isolates. Antimicrob. Agents Chemother.
49: 256263. 2005.
· Harbottle H, Thakur S, Zhao D and White
G. Genetics of antimicrobial resistance. Anim Biotechnol;
17(2): 111-124. 2006.
· Hardman J.G. et Limbird L.E. Les
bases pharmacologiques de l'utilisation des medicaments. 9éme
édition. Nieuwegein, Pays-Bas, Mc Graw-Hill. 1069-1098.
1998.
· Hart C.A. Klebsiella,
Citrobacter, Enterobacter and Serratia spp. Principles and
practice of Clinical Bacteriology. England, UK: John Wiley and Sons Ltd. 2nd
ed: 377- 386. 2006.
· Hawkey P.M. Prevalence and clonality
of extended-spectrum beta-lactamases in Asia. Clin Microbiol Infect,
14 Suppl 1, 159-65. 2008.
· Hawkey P.M. Prevalence and clonality
of extended-spectrum â-lactamase in Asia clin Micr Infect; 14:
159-65. 2012.
· Hennequin C., Robin F., Cabrolier N., Bonnet
R. and Forestier C. Characterization of a DHA-1-producing
Klebsiella pneumoniae strain involved in an outbreak and role of the
AmpR regulator in virulence. Antimicrob. Agents Chemother. 56(1):
288-294. 2012.
· Hirakata Y., Matsuda J., Miyazaki Y., Kamihira
S., Kawakami S., Miyazawa Y., Ono, Y., Nakazaki N., Hirata Y., Inoue M.,
Turnidge J.D., Bell J.M., Jones R.N., Kohno S. et Participants T.S.A.P.
Regional variation in the prevalence of extended-spectrum
b-lactamase-producing clinical isolates in the Asia-Pacific region (SENTRY
1998-2002). Diagnostic Microbiology and Infectious Disease,
52, 323-329. 2005.
80
Références bibliographiques
· Humeniuk C., Arlet G., Gautier V., Grimont P.,
Labia R. and Philippon A. f3-lactamases of Kluyvera
ascorbata, probable progenitors of some plasmid-encoded CTX-M types.
Antimicrob. Agents Chemother. 46: 3045-9. 2002.
· Isendhal J., Turlej-Rogacka A., Manjuba C.,
Rodrigues A., Giske C.G. and Naucler P. Fecal Carriage of
ESBL-Producing E. coli and K. pneumoniae in Children in
Guinea-Bissau: A Hospital-Based Cross-Sectional Study. PLoS ONE,
7, e51981. 2012.
· Ito H, Arakawa Y and Ohsuka S.
Plasmid-mediated dissemination of the metallo-beta-lactamase gene
blaIMP among clinically isolated strains of Serratia
marcescens. Antimicrob Agents Chemother, 39: 824-9.
1995.
· Jacobson K.L., Cohen S.H., Inciardi J.F., King
J.H., Lippert W.E., Iglesias T. and Van Couwenberghe C.J. The
relationship between antecedent antibiotic use and resistance to
extended-spectrum cephalosporins in group I beta-lactamase-producing
organisms. Clin Infect Dis. 21(5): 1107-1113.
1995.
· Jacoby G.A. and Medeiros A.A. More
extended-spectrum f3-lactamases. Antimicrob. Agents Chemother. 35:
1697-1704. 1991.
· Jacoby G.A. and Munoz-Price L.S. The
new f3-lactamases. N Engl J Med. 352: 380391.
2005.
· Jacqueline C. and Tattevin P.
Ceftaroline, a cephalosporin with activity against methicillin
resistant Staphylococcus aureus: Experimental and clinical data,
therapeutic applications. J. Anti-infect. 14 (1): 3-10.
2012.
· Janvier A., Mérens A., Delaune D.,
Soler C. et Cavallo J.D. Portage digestif
d'entérobactéries de résistantes aux
céphalosporines de troisième génération dans une
population d'adultes jeunes asymptomatiques : évolution entre 1999 et
2009 F. Pathologie Biologie ; 59 : 97-101, 2011.
· Jarlier V., Nicolas M.H., Fournier G. and
Philippon A. Extended-broads-pectrum f3-lactamases conferring
transferable resistance to newer f3-lactam agents in Enterobacteriaceae:
hospital prevalence and susceptibility patterns. Rev Infect Dis. 10:
867-878. 1988.
· Jaurin B. and Grundstrom T. ampC
cephalosporinase of Escherichia coli K12 has a different evolutionary
origin from that of f3-lactamases of the penicillinase type. Proc Nat1 Acad
Sci. USA. 78(8): 4897-4901. 1981.
· Joly B. et Reynaud A.,
Entérobactéries : Systématique et méthodes de
diagnostic. Edition Te. 3-182. 2007.
81
Références bibliographiques
· Kado C.I. and Liu S.T. Rapid
procedure for detection and isolation of large and small plasmids. J.
Bacteriol. 145: 1365-1373. 1981.
· Kardas-Soma L., Lucet J., Pelat C.,
Perozziello A., Boëlle P., Andremont A. et Yazdanpanah Y. PADS
1-05 - Modélisation des coûts et bénéfices des
stratégies de contrôle de la transmission des
entérobactéries productrices de â-lactamases à
spectre étendu (EBLSE) dans un service de réanimation.
Médecine et Maladies Infectieuses, Volume 46, Issue 4,
Supplement 1, Page 89, June 2016.
· Kassis-Chikhani N., Vimont S., Asselat K.,
Trivalle C., Minassian B., Sengelin C., Gautier V., Mathieu D., Dussaix E. and
Arlet G. CTX-M â-lactamase-producing Escherichia coli
in longterm care facilities, France. Emerg Infect Dis ; 10:
1697-1698. 2004.
· Kenneth V.I., Rolston M.A., Jamal L., Nesher
Samuel A., Shelburne R., Issam A., Randall. In vitro activity
of ceftaroline and comparator agents against Grampositive and Gram-negative
clinical isolates from cancer patients. Intern. J. Antimicr. Agen.
2017.
· Kizilca O, Siraneci R, Yilmaz A, Hatipoglu N
et Ozturk E, Kiyak A. Risk factors for community-acquired urinary
tract infection caused by ESBL-producing bacteria in children. Pediatr
Int; 54(6):858--62. 2012.
· Kliebe C., Nies B. A., Meyer J. F.,
Tolxdoref-Neutzling R. M. et Wiedemann B. Evolution of plasmid-coded
resistance to broad-spectrum cephalosporins. Antimicrob Agents Chemother,
28, 302-7. 1985.
· Knothe H., P. Shah V. Krcmery M. Antal and
Mitsuhashi S. Transferable resistanceto cefotaxime, cefoxitin,
cefamandole and cefuroxime in clinical isolates of Klebsiella pneumoniae
and Serratia marcescens. J Infect. 11:315-317.
1983.
· Kong K.F., Schneper L. and Mathee K.
Beta-lactam antibiotics: From antibiosis to resistance and
bacteriology. APMIS. 118 (1): 1-36. 2010.
· Kumar A. and Schweizer H.P. Bacterial
resistance to antibiotics: Active efflux and reduced uptake. Adv Drug
Delivery Rev. 57: 1486-1513. 2005.
· Lahlou A., Chegri M. et L'kassmi H.
Épidémiologie et résistance aux antibiotiques des
entérobactéries isolées d'infections urinaires à
l'hôpital militaire Moulay-Ismail de Meknès I. j Ant ;
11, 90-96. 2009.
· Laupland K.B., Church D.L., Vidakocich J.,
Mucenski M. and Pitout J.D. Community-onser extended-spectrum
beta-lactamase ESBL producing Escherichia coli: Importance of
international travel. J. Infect. 57p. 2008.
82
Références bibliographiques
· Lautenbach E., Patel J.B., Bilker W.B.,
Edelstein P.H. and Fishman N.O. Extendedspectrum
f3-lactamase-producing Escherichia coli and Klebsiella
pneumoniae: risk factors for infection and impact of resistance on
outcomes. Clin Infect. 32: 1162- 1171. 2001.
· Lemaoui C.E, Layaida H, Badi A et Foudi N.
Stratégies de lutte contre la résistance aux
antibiotiques. Review article Journal des Anti-infectieux, Volume 19, Issue 1,
Pages 12-19. March 2017.
· Livermore D.M. «Beta-Lactamases
in laboratory and clinical resistance». Clin Microbiol Rev. 8(4):
557-584. 1995.
· Livermore D.M., Canton R., Gniadkowski M.,
Nordmann P., Rossolini G.M., Arlet G., Poirel L. and Woodford N.
CTX-M: changing the face of ESBLs in Europe. Journal Antimicrob
Chemother. 59(2): 165-174. 2007.
· Lonchel C., Melin P., Gangoue-Pieboji J.,
Okomo Assoumou M.C., Boreux R. et De Mol P. Extended-spectrum
beta-lactamase-producing Enterobacteriaceae in Cameroonian hospitals.
Eur J Clin Microbiol Infect Dis, 32, 79-87.
2013b.
· Lonchel C., Melin P., Gangoue-Pieboji J.,
Okomo Assoumou M.C., Boreux R. et De Mol. Prevalence and spread of
extended-spectrum f3-lactamase-producing Enterobacteriaceae in
Ngaoundere, Cameroon. Clinical Microbiology Infectious, 19,
416-420. 2013a.
· Lonchel C.M., Meex C.,
Gangoué-Piéboji J., Boreux R., Assoumoui M.C., Lelin P and de Mol
P. Proportion of extended-spectrum f3-lactamase producing
enterobacteriaceae in community setting in nagaoundere , Cameroon.
Infect. Diseas. 12: 53p. 2012.
· Luvsansharav U.O., Hirai I., Niki M., Nakata
A., Yoshinaga A., Moriyama T. and Yamamoto Y. Prevalence of fecal
carriage of extended-spectrum f3-lactamaseproducing Enterobacteriaceae among
healthy adult people in Japan. J Infect Chemother. 17(5): 722- 725.
2011.
· Martinez J.L. The role of natural
environments in the evolution of resistance traits in pathogenic bacteria.
Proc Biol Sci; 276:2521-30. 2009.
· Matthew M., Hedges R.W. and Smith J.T.
Types of f3-lactamase determined by plasmids in gram-negative
bacteria. J Bacteriol. 138(3): 657-662. 1979.
83
Références bibliographiques
· Mayer K., Opal S. and Medeiros A.
Mechanisms of antibiotic resistance. In: Mandell, Douglas and Bennett's
Principles and Practice of Infectious Diseases. 5th edition, Churchill
Livingstone. 2: 236-253. 2000.
· Mendonça N., Ferreira E., Louro D.,
Caniça M. and ARSIP Participants. Molecular epidemiology and
antimicrobial susceptibility of extended- and broad-spectrum
â-lactamaseproducing Klebsiella pneumoniae isolated in Portugal.
Int J Antimicrob Agents. 34: 29-37. 2009.
· Mérabaud M.I.
Entérobactéries à BLSE en pédiatrie en
1996. Thèse n°10303. 2003.
· Messai Y., Iabadene H., Benhassine T.,
Alouache S., Tazir M., Gautier V., Arlet G. et Bakour R. Prevalence
and characterization of extended-spectrum b-lactamases in Klebsiella
pneumoniae in Algiers hospitals (Algeria). Pathologie Biologie,
56, 319-325. 2008.
· Metuor Dabire A.
Caractérisations moléculaire et cinétique des
types de Ji-lactamases à spectre élargi (BLSE) de souches
bactériennes collectées au Centre Hospitalier Universitaire
Pédiatrique Charles De Gaulle (CHUP-CDG) de Ouagadougou. Biologie
Moléculaire / Enzymologie thèse de doctorat unique,
Université de Ouagadougou. 149p. 2014.
· Naas T. et Vaux S. Bactéries
hautement résistantes aux antibiotiques : épidémiologie
mondiale et française. 44-47. 2012.
· Naas T., Cuzon G., Villegas M.V. et
al. Genetic structures at the origin of acquisition of the ß-lactamase
blaKPC gene. Antimicr. Agen. Chemo. 52 : 63-1257.
2008.
· Nadmi H., Elotmani F., Talmi M., Zerouali K.,
Perrier Gros C. et Timinouni M. Profil de résistance aux
antibiotiques des entérobactéries uropathogènes
communautaires à El Jadida (Maroc). Journal Médecine et
maladies infectieuses ; 40 : 303-305, 2010.
· Nauciel C. et Vildé J.L.
Bactériologie médicale. 2éme édition.
Masson. Paris. 20-126. 2005.
· Nedjai S., Barguigua A., Djahmi N., Jamali L.,
Zerouali K., Dekhil M. and Timinouni M. Prevalence and
characterization of extended spectrum beta-lactamase-producing Enterobacter
cloacae strains in Algeria. J Infect Dev Ctries, 7,
804-811. 2013.
· Neulier C., Birgand G., Ruppé E.,
Armand-Lefèvre L., Lolom I., Yazdanpanah Y., Lucet J.C. et Andremont A.
Enterobacteriaceae bacteremia: Risk factors for
84
Références bibliographiques
ESBLPE. Médecine and Maladies Infectieuses,
Volume 44, Issue 1, 32-38, January 2014.
· Neuwirth C., Siebor E., Duez J.M.,
Péchinot A. and Kazmierczak A. Imipenem resistance in
clinical isolates of Proteus mirabilis associated with alterations in
penicillinbinding proteins. J Antimicrob Chemother. 36(2): 335-342.
1995.
· Nordmann P, Boulanger A.E. and Poirel L.
NDM-4 metallo-ss-lactamase with increased carbapenemase activity from
Escherichia coli. Antimicrob Agents Chemother. 12 Jan
2012.
· Nordmann P, Naas T. and Poirel L.
Global spread of Carbapenemase-producing Enterobacteriaceae. Emerg
Infect Dis; 17: 1791-8. 2011.
· Nordmann P. et Carrer A. Les
carbapénèmases des entérobactéries, Arch.
Péd. 16 (4) : S154-162. 2010.
· Nordmann P., Cuzon G. and Naas T. The
real threat of Klebsiella pneumoniae carbapenemase-producing bacteria.
Lancet. Infect. Dis. 9: 228-36. 2009.
· Nordmann P., Dortet L. and Poirel L.
Carbapenem resistance in Enterobacteriaceae: here is the storm!
Tren. Mol. Med. 18 (5): 263-272. 2012.
· Okalla Ebongue C., Dongmo Tsiazok M, Nda
Mefo'o J.P., Ngaba N.P., Beyiha G. et Adiogo D. Evolution of
antibiotic resistance of Enterobacteriaceae isolated at the Douala General
Hospital from 2005 to 2012. Pan Afr Med J; 20: 227.
2015.
· Papanicolaou G.A., Medeiros A.A., and Jacoby
G.A. Novel plasmid mediated betalactamase (MIR-1) conferring
resistance to oxyimino-and alpha-methoxy beta-lactams in clinical isolates of
Klebsiella pneumoniae. Antimicrob. Agents Chemother. 34:
2200-2209. 1990.
· Paterson D.L. and Bonomo R.A.
Extended-spectrum beta-lactamases: a clinical update. Clin
Microbiol Rev. 18: 657-686. 2005.
· Peery J., Staley J.T et Lory S.
Microbilogie .Edition Dunod. 423 p. 2004.
· Peirano G., Van Greuneb C.H.J. and Pitout
J.D.D. Characteristics of infections caused by extended-spectrum
â-lactamase-producing Escherichia coli from community hospitals
in South Africa. Diagnostic Microbiology and Infectious Disease, 69,
449-453. 2011.
85
Références bibliographiques
· Pepperell C., Kus J.V., Gardam M.A., Humar A.
and Burrows L.L. Low-Virulence Citrobacter Species Encode
Resistance to Multiple Antimicrobials. Antimicrob. Agents Chemother.
46(11): 3555-3560. 2002.
· Philippon A. and Arlet G.
f3-Lactamases de bacilles à Gram négatif : le mouvement
perpétuel ! Ann Biol Clin. 64(1): 37-51.
2006.
· Philippon A.
Entérobactéries des f3-lactamines. Biol. méd. 3
(4) : 1-18. 2008.
· Philippon A. et arlet G.
Entérobactéries et bétalactamine : Phénotypes de
résistance naturelle. Pathol. Biol. 60: 112-126.
2012.
· Philippon A., Arlet G. and Jacoby G.A.
Plasmid-determined AmpC-type f3-lactamases. Antimicrob. Agents
Chemother. 46: 1-11. 2002.
· Pietsch M., Eller C., Wendt C., Holfelder M.,
Falgenhauer L., Fruth A., Grössl T., Leistner R., Valenza G., Werner G.,
Pfeifer Y. RESET Study Group. Molecular characterisation of
extended-spectrum f3-lactamase (ESBL)-producing Escherichia coli
isolates from hospital and ambulatory patients in Germany. Veterinary
Microbiology, Volume 200, Pages 130-137. February
2017.
· Pitout J.D., Thomson K.S., Hanson N.D.,
Ehrhardt A.F., Moland E.S.et Sanders C. C. Beta-Lactamases responsible
for resistance to expanded-spectrum cephalosporins in Klebsiella
pneumoniae, Escherichia coli, and Proteus mirabilis isolates
recovered in South Africa. Antimicrob Agents Chemother, 42,
1350-4. 1998.
· Poirel L, Héritier C and Nordmann
P. Chromosome-encoded ambler class D beta-lactamase of Shewanella
oneidensis as a progenitor of carbapenem-hydrolyzing oxacillinase.
Antimicrob Agents Chemother; 48:348-51. 2004.
· Poirel L, Héritier C, Tolun V et
Nordmann P. Emergence of oxacillinase-mediated resistance to imipenem
in Klebsiella pneumoniae. Antimicrob Agents Chemother; 48;
1522. 2004.
· Poirel L., Pitout J.D. and Nordmann
P. Carbapenemases: Molecular diversity and clinical consequences.
Fut Micr; 2: 12-501. 2007.
· Poole K. "Resistance to beta-lactam
antibiotics." Cell Mol Life Sci. 61(17): 2200-2223.
2004.
86
Références bibliographiques
· Porres-Osante N., Dupont H., Torres C.,
Ammenouche N., De-Champs C. and Mammeri H. Avibactam activity against
extended-spectrum AmpC ß-lactamases. J. Antimicr. Chemo.
2014.
· Queenan A.M., Bush K. Carbapenemases:
The versatile beta-lactamases. Clin Micr Rev. 20: 440-58.
2007.
· Rahal K., Belouni R. and Benslimani A.
Standardisation de l'antibiogramme en médicine humaine à
l'échelle nationale. Rec de L'OMS. 4éme édition.
Algérie. 46-52. 2005.
· Rakotovao-Ravahatra Z.D., Randriatsarafara
F.M., Rasoanandrasana S., Raverohanta L. et Rakotovao A.L.
Phénotypes de résistance des souches d'Escherichia coli
responsables d'infection urinaire au laboratoire du Centre
Hospitalo-Universitaire de Befelatanana Antananarivo. Pan African Medical
Journal ; 26.166.11828. 2017.
· Recchla G.D and Hall R.M. Origins of
the mobile gene cassettes found in integrons. Trends Microbiol; 5(10):
389-394. 1997.
· Robin F., Gibolda l. et Bonneta R.
Résistance naturelles et acquise aux f3-lactamine chez les
entérobactéries : Comment les identifier en pratique quotidienne.
franco labos. 445 : 47-57. 2012.
· Rodriguez-Bano J., Lopez-Cerero L., Navarro
M.D., Diaz de Alba P. and Pascual A. Faecal carriage of
extended-spectrum beta-lactamase-producing Escherichia coli:
prevalence, risk factors and molecular epidemiology. J Antimicrob
Chemother. 62(5): 1142-1149. 2008b.
· Rodriguez-Villalobos M. et Struelens
M.J. Résistance bactérienne par f3-lactamase à
spectre étendu : Implication pour le réanimateur.
Réanimation. 15 : 205-2013. 2003.
· Rodriguez-Villalobos M. et Struelens,
M.J. Résistance bactérienne par f3-lactamases à
spectre étendu: implications pour le réanimateur. Rev
Réanimation 15. (3): 205-213. 2006.
· Rossolini G.M., D'Andrea M.M. and Mugnaioli C.
The spread of CTX- M- type extended- spectrum beta- lactamases. Clin
Microbiol Infect 14 Suppl 1:33- 41. 2008.
· Rubin M.A. and Samore M.H.
Antimicrobial Use and Resistance. Curr Infect Dis Rep. 4:
491-497. 2002.
87
Références bibliographiques
· Ruppé E., Woerther P.l., Diop A.M.,
Da-costa A. et Costa Arlet G. Carriage of CTX-M-15 producing
Escherichia coli isolates amoung children living in a remote in
Senegal. Anti. Agen. Chem. 53: 7-3135.
2009.
· Schwaber M.J and Carmeli Y. Mortality
and delay in effective therapy associated with extended-spectrum beta-lactamase
production in Enterobacteriaceae bacteraemia: a systematic review and
meta-analysis. J Antimicrob Chemother. 60: 913-920.
2007.
· Schwarz S. and Chaslus-Dancla E. "Use
of antimicrobials in veterinary medicine and mechanisms of resistance." Vet
Res. 32(3-4): 201-225. 2001.
· Seid J. and Asrat D. Occurrence of
extended spectrum â-lactamase enzymes in clinical isolates of
Klebsiella species from Harar region, eastern Ethiopia. Acta
Tropica 95, 143148. 2005.
· Sekhsokh Y., Chadli M. et El Hamzaoui S.A.
Fréquence et sensibilité aux antibiotiques des
bactéries isolées dans les urines. Méd et Mal inf
; 38 : 324-327. 2008.
· Singelton P. Bactériologie
pour la médcine, la biologie et la biotechnologie .6éme
édition. Dunod. Paris. 40-316. 2005.
· Sire J.M., Nabeth P., Perrier-Gros-Claude
J.D., Bahsoun I., Siby T., Macondo E.A., Gaye-Diallo A., Guyomard S., Seck A.,
Breurec S. and Garin B. Antimicrobial resistance in outpatient
Escherichia coli urinary isolates in Dakar, Senegal. J Infect Dev
Ctries, 1, 263-8. 2007.
· Sirot J. Prospective survey of
colonization and infection caused by expandedspectrum-beta-lactamase-producing
members of the family Enterobacteriaceae in an intensive care unit. J Clin
Microbiol. 27(12): 2887-2890. 1989.
· Snyder L. and Champness W. Molecular
genetics of bacteria. Washington, D.C. ASM Press.
2007.
· Soge O.O., Queenan A.M., Ojo K.K., Adeniyi
B.A. et Roberts M.C. CTX-M-15 extended-spectrum (beta)-lactamase from
Nigerian Klebsiella pneumoniae. J Antimicrob Chemother,
57, 24-30. 2006.
· Sood S. and Gupta R. Antibiotic
resistance pattern of community acquired uropathogens at a tertiary care
hospital in Jaipur, Rajasthan. Indian J Community Med; 37(1):39-44.
2012.
· Spratt B.G., Zhang Q.Y., Jones D.M.,
Hutchison A., Brannigan J.A. and Dowson C.G. Recruitment of a
penicillin-binding protein in gene from Neisseria flavescens
88
Références bibliographiques
during the emergence of penicillin resistance in
Neisseria meningitidis. Proc Natl Acad Sci. USA. 86(22):
8988-8992. 1989.
· Storberg V. ESBL-producing
Enterobacteriaceae in Africa - a non-systematic literature review of
research published 2008-2012. Infections Ecology and Epidemiology,
4, 1-16. 2014.
· Stürenburg E. and Dietrich M.
Extended spectrum beta- lactamases implication for the clinical
microbiology, therapy, and infection control. J Infect. 47: 273-295.
2003.
· Tandé D., Jallot N., Bougoudogo F.,
Montagnon T., Gouriou S. and Sizun J. Extended-Spectrum 3-Lactamase-
Producing Enterobacteriaceae in Malian Orphanage. Emerging
Infectious Diseases, 15, 472-474.
2009.
· Tandé D., Jallot N., Bougoudogo F.,
Montagnou T., Gouriou S. and Sizun J. Extended- spectrum
béta-lactamase producing enterobacteriacea isolates in Mexico.
Micr Drug Resis. 17: 497-505.
2005.
· Tansarli G.S., Poulikakos P., Kapaskelis A.
and Falagas M.E. Proportion of extended-spectrum beta-lactamase
(ESBL)-producing isolates among Enterobacteriaceae in Africa:
evaluation of the evidence--systematic review. J Antimicrob Chemother,
69, 1177-84. 2014.
· Thibaut S., Marquet A., Collet C, Grandjean
G., Boutoille D., Caillon J. Épidémiologie des
bactériuries des souches d'infections urinaires communautaires :
comparaison entre les patients vivant à domicile et en
établissements d'hébergements pour personnes âgées
dépendantes (EHPAD) M. Réseau Med Qual, CHU de Nantes EN
France. J Med Mal; 03.072. 2017.
· Thomas C.M and Nielsen K.M.
Mechanisms of, and barriers to, horizontal gene Transfer between
bacteria. Naature Review Microbiology, 3: 711-721.
2005.
· Thomson K.S. and Moland E.S. The new
3-lactamases of Gram negative bacteria at the dawn of the new millennium.
Microbes Infect. 2: 1225-1235. 2000.
· Tiouit D, Naim M. et Amhis W.
Médecine du Maghreb 2001 n°91 : Traitement antibiotique des
infections urinaires Page 35 à 38. 2001.
· Touati A., Medboua C., Touati D., Denine R.,
Brasme L. and de Champs C. CTXM-15- producing Enterobacteriaceae
isolates causing bloodstream infections at the Beni-Messous hospital in Algeirs
(Algeria). Int Res J Microbiol. 3: 181-185. 2012.
89
Références bibliographiques
· Villegas M.V., Kattan J.N., Quinteros M.G.,
Casellas J.M. Prevalence of extended-spectrum â-lactamase in
South America. Clin Micr Infect. 14: 8-154. 2008.
· Vodovar D, Marcad2 G, Raskine L, Malissin I et
Mégarbane B. Entérobactéries productrices de
bêta-lactamases a spectre élargi. Rev Méd Int,
Vol:34, issue 11 p:687693. Nov 2013.
· Vora S. et Auckenthaler R. Que
signifie «bêtalactamases à spectre élargi» en
pratique?. Rev Med Suisse. 5: 1991-1994.2009.
· Walsh C. Antibiotics: actions,
origins, resistance. American Society for Microbiology press.
Washington. 2003.
· Walsh T.R. Clinically signifi cant
carbapenemases: an update. Curr Opin Infect Dis; 21:367-71.
2008.
· Walsh T.R. Combinatorial genetic
evolution of multiresistance. Curr Opin Microbiol 9:476-82.
2006.
· WEBER Ph. Etat actuel de la
sensibilité à la ciprofloxacine des bactéries
isolées en pratique en ville: résultats d'une enquête
multicentrique. Méd Mal Infect ; 23 :342-7.
1993.
· Weldhagen G.F., Poirel L. and Nordmann P.
Ambler class A extended-spectrum â-lactamases in Pseudomonas
aeruginosa: novel developments and clinical impact. Antimicrob. Agents
Chemother. 47: 2385-2392. 2003.
· Wiener J., Quinn J.P., Bradford P.A., Goering
R.V., Nathan C., Bush K. and Weinstein R.A. Multiple
antibiotic-resistant Klebsiella and Escherichia coli in
nursing homes. JAMA. 281(6): 517-523. 1999.
· Winokur P.L., Canton R., Casellas J.M. et
Legakis N. Variations in the Prevalence of Strains Expressing an
Extended-Spectrum b-Lactamase Phenotype and Characterization of Isolates from
Europe, the Americas, and the Western Pacific Region. Clinical Infectious
Diseases 32, S94-S103. 2001.
· Woerther P.-L., Angebault C., Jacquier H.,
Hugede H.C., Janssens A.C., Sayadi S., El Mniai A., Armand-Lefévre L.,
Ruppé E., Barbier F., Raskine L., Page A.L., Rekeneire N. and Andermont
A. Massive Increase, Spread, and Exchange of Extended Spectrum
b-Lactamase-Encoding Genes Among Intestinal Enterobacteriaceae in
Hospitalized Children With Severe Acute Malnutrition in Niger. Clinical
Infectious Diseases 53, 677-685.
2011.
90
Références bibliographiques
? Woerther P.L., Angebault C., Lescat Mruppé
E., Skurnik D. et Mniai A.E. Emergence and dissemination of
extended-spectrum beta-lactamase producing enterobacteriacea in the
community: Lessons from the study of a remote and controlled population. J.
Infect. Dis. 202: 515-23. 2010.
? Yigit H., Queenan A.M., Anderson G.J. et al.
Novel carbapenem hydrolyzing beta-lactamase KPC-1 from a
carbapenem-resistant strain of Klebsiella pneumoniae. Anti. Agen.
Chemo. 45: 61-1151. 2001.
? Yong D, Toleman MA and Giske CG.
Characterization of a new metallo-beta-lactamase gene, (bla-NDM-1),
and a novel erythromycin esterase gene carried on a unique genetic structure in
Klebsiella pneumoniae sequence type 14 from India. Antimicrob
Agents Chemother; 53:5046-54. 2009.
? Zogheib, E., and Dupont, H.
Entérobactéries multirésistantes. Elsevier SAS.
Conférences d'actualisation ; 153-165. 2005.
? Zongo K.J., Metuor Dabire A., Compaore L.G., Sanou I.,
Sangare L., Simpore J. and Zeba B. First detection of bla TEM, SHV and
CTX-M among Gram negative bacilli exhibiting extended spectrum â-
lactamase phenotype isolated at University Hospital Center, Yalgado Ouedraogo,
Ouagadougou, Burkina Faso. African Journal of Biotechnology, 14
1174-1180. 2015.
ANNEXE
Figure 32. Feuille de résultats
d'antibiogramme.
Annexe
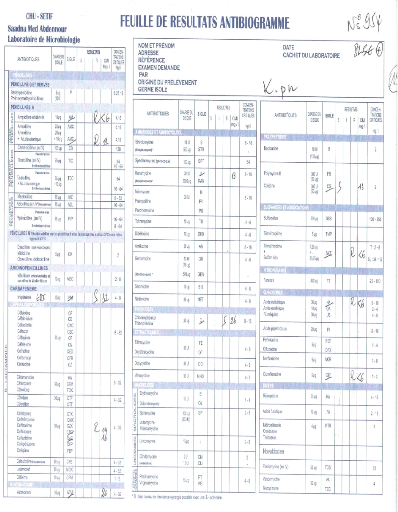

Figure 33. Résultat final d'un
antibiogramme fait par l'automate.
Annexe
Annexe

Figure 34. La plaque utilisée par
l'automate pour l'identification et l'antibiogramme.
Annexe

? Figure 35. L'automate MicroScan WalkAways.
| 


