

MEMOIRE BIBLIOGRAPHIQUE
ASPECT GÉNÉTIQUES ET MOLÉCULAIRES
DE LA RÉPONSE GÉNÉRALE AUX STRESS CHEZ Escherichia
coli : LE RÉGULON RPOS
Master 1 BCGMP
DUFOUR Virginie
Année 2006/2007
TABLE DES MATIÈRES
INTRODUCTION
I) IDENTIFICATION DES GÈNES RÉGULÉS PAR
RPOS
Matériels et méthodes
Résultats et discussion
II) CARACTÉRISATION DES PROMOTEURS DU RÉGULON RPOS
Matériels et méthodes
Résultats et discussion
Un promoteur consensus ?
Mécanismes moléculaires de la
sélectivité ó70/óS.
CONCLUSION
BIBLIOGRAPHIE
ANNEXES
Introduction :
Chez les bactéries, le coeur de l'ARN
polymérase est unique mais une sous-unité, le facteur sigma,
responsable de la reconnaissance des promoteurs et de la formation du complexe
ouvert, est présente en différents exemplaires. Ces facteurs
jouent un rôle majeur dans la régulation de l'expression des
gènes. Chez Escherichia coli, au moins sept facteurs
ó ont été identifiés. Ces sous-unités
permettent chacune la transcription directe d'un groupe de gènes
spécifiques, possédant un promoteur qu'elles sont capables de
reconnaître. Le facteur ó « d'entretien
cellulaire » de E. coli est nommé ó70,
mais ce facteur peut être partiellement remplacé en conditions de
stress par le facteur ó alternatif óS (ou RpoS), principalement
exprimé en phase stationnaire et en situation de stress (carences,
hyperosmolarité, choc acide, températures extrêmes). Il
s'agit d'un cas intéressant car certains promoteurs reconnus par
ó70 sont aussi reconnus par óS, alors que les autres
sous-unités ó ne reconnaissent que les promoteurs qui leur sont
spécifiques. Il est aussi connu que l'ARN polymérase holoenzyme
contenant óS (abrégé EóS) contrôle
directement ou indirectement l'expression de très nombreux
gènes : et est donc considéré comme le
régulateur maître de la réponse aux stress chez E. Coli, et
comme un objet d'étude majeur. (5. LACOUR, KOLB, LANDINI, 2003 ;
2.WEBER, et al, 2005 )
Cependant, de nombreuses questions se posent : combien
de gènes sont sous le contrôle de óS? Quelles sont les
fonctions physiologiques des produits de ces gènes ? Peut on
caractériser un profil commun à ces gènes ? (un
promoteur óS consensus?) L'expression de ces gènes est elle
simplement proportionnelle à la concentration cellulaire de RpoS? Qu'est
ce qui permet à óS de reconnaitre les promoteurs de ó70 et
comment se fait le choix? (2. WEBER, et al, 2005 ; 8. HENGGE-ARONIS, 2002) Les
réponses aux signaux externes nécessitent l'intégration de
nombreux signaux complexes : RpoS est la clé de la réponse
à de nombreux stress, mais comment les signaux sont
intégré, et qu'est ce qui régule l'expression de
RpoS ? En effet sa quantité (de forme active) augmente grandement
durant la phase stationnaire. Il semble être soumis à de
nombreuses régulations, intervenant à tous les niveaux :
transcription, traduction, protéolyse et activité.
Particulièrement, il semble régulé de manière
transcriptionnelle par CRP-AMPc et de manière post-transcriptionnelle
par de petits ARN régulateurs, par exemple DsrA. D'autres facteurs
influenceraient RpoS, comme la protéine histone-like H-NS, les ARN6s,
qui en mimant la structure des promoteurs reconnus par óS le
séquestrent, ou le régulateur traductionnel global Hfq.
(Revues : 8. HENGGE-ARONIS, 2002, 9. REPOILA, MAJDALANI, GOTTESMAN, 2003,
Articles : 10. HIRSCH, ELLIOTT, 2005 et 11. LEASE, et al, 2004)
I) Identification des gènes régulés
par RpoS
L'approche la plus ancienne visant à déterminer
quels gènes sont sous le contrôle de RpoS consisterait à
cribler une banque de bactéries contenant des fusions lacZ
aléatoires en y mutant le gène rpoS. Les fusions
lacZ sont une bonne méthode de mesure de l'expression des
gènes puisque la â-galactosidase est stable et très facile
à doser, et particulièrement pour l'étude des gènes
en phase stationnaire, où la stabilité des ARN et
protéines étudiés est inconnue. (3. VIJAYAKUMAR, et al,
2004)
Une approche plus récente et plus globale est celle
des puces à ADN, comparant le transcriptome de bactéries
possédant ou non un gène rpoS fonctionnel soumises
à des stress. On observera donc des profils d'expression
différents, correspondant aux gènes régulés
directement ou indirectement par óS. (2. WEBER, et al, 2005)
Matériels et méthodes
Souches bactériennes et conditions de
croissance (pour l'études des fusions lacZ). Les
auteurs (3. VIJAYAKUMAR, et al, 2004) ont utilisé des souches
dérivées de K12 : HS1002-HS1100 (ÄlacU169) et
portant des fusions lacZ aléatoires construites comme
indiqué par Schellhorn et al., 1998, cité par 3. VIJAYAKUMAR, et
al, 2004), GC122 (ÄlacU169 rpoS::Tn10, Schellhorn et
al., 1988, cité par 3. VIJAYAKUMAR, et al, 2004). HS1002-HS1100 (les
souches de la banque de fusions lacZ) ont été
conjuguées avec une GC122 Hfr (rpoS::Tn10 est
transféré quelques minutes après l'origine de transfert).
L'allèle sauvage de rpoS est inactivé par recombinaison homologue
de rpoS::Tn10 chez les souches portant les fusions : cette
recombinaison est sélectionnée par une diminution de la
coloration bleutée des cellules sur LB lactose + Xgal, et la
dépendance à óS des gènes affectés par la
fusion a été vérifiée par complémentation
avec le plasmide pMMkatF3, portant l'allèle sauvage de
rpoS (Muvey et al. 1988). (figure 1)
Test enzymatique. L'activité
â-galactosidase a été testé avec de l'ONPG
(o-nitrophényl-â-D-galactopyranoside), et calculée
ainsi : (1000 x DO420nm)/(temps d'incubation en minutes x
volume en mL x DO600nm). (3. VIJAYAKUMAR, et al, 2004) Le test a
été effectué sur des cellules en phase stationnaire ou
exponentielle.
Identification des gènes en fusion avec
lacZ. Les zones proximales à l'insertion de lacZ ont
été séquencées comme indiqué par Roy et al.,
1995, (cité par 3. VIJAYAKUMAR, et al, 2004) et identifiées
grâce au logiciel BLASTN. Les fonctions de ces gènes ont
été prédites grâce aux logiciel SWISSPROT. (3.
VIJAYAKUMAR, et al, 2004)
Souches bactériennes et conditions de
croissance (pour les puces à ADN). Les auteurs (2. WEBER, et
al, 2005) ont utilisé un dérivé de la souche MC4100,
portant la mutation rpoS::Tn10 ou non (souches MC4100 et RH90). Les
souches sont cultivées en LB (milieu de Luria Bertani) à
37°C sous agitation. Pour l'expérience de choc hyperosmotique, on
transfère les cellules en milieu minimum M9 avec glycérol (0,4%)
pendant au moins 3 générations avant d'ajouter du NaCl à
0,3M. Pour l'expérience de choc acide (chute du pH), on cultive les
cellules pendant au moins 4 générations en LB avant d'ajouter
170mM d'acide 4-morpholine-methasulfonique (MES), ce qui acidifie le milieu
à pH=5. Les croissances sont mesurées par densité optique
à 578nm. (2. WEBER, et al, 2005)
Origine des puces à ADN de E. coli K12.
Les puces génomiques ont été fabriquées en
spottant avec un robot des produits de PCR obtenus avec des amorces permettant
l'extension d'ORF (cadres de lectures). On obtient donc toutes les ORF doubles
brins du génome. (détails de la procédure
utilisée : Polen et al., 2006, cité par 2. WEBER, et al,
2005)
Préparation de L'ARN et marquage des
ADNc. Les bactéries recueillies ont été
cultivées selon trois conditions différentes, qui permettent une
forte production de RpoS et une forte induction des gènes
contrôlés par RpoS : (i) durant la transition entre la phase
exponentielle et stationnaire de croissance en LB (DO578nm=4.0),(ii)
20 minutes après ajout de 0.3M de NaCl (ajouté à
DO578nm=0.3) en milieu M9+glycérol 0.4%, et (iii) 40 min
après chute du pH à 5 en LB (ajout de MES à
DO578nm=0.4). Les cellules sont centrifugées 2 min à
4500g à 4°C, et le culot est resuspendu dans 700ìL de tampon
RLT (RNeasy, QIAGEN, Allemagne) et lysées par choc grâce à
un appareil de lyse à microbilles (Mini-Bead Beater, Biospec products).
Après centrifugation, 500ìL d'éthanol est ajouté au
lysat, et le lysat est séparé en 2 lots. Les ARN totaux sont
extraits sur colonne RNeasy (QIAGEN) selon les instructions du fabriquant. Les
ARN isolés sont traités à la DNAse I (pour éliminer
l'ADN qui pourrait contaminer la solution) pendant 20min à 37°C,
puis incubés à 70°C pendant 10min pour inactiver la DNAse I,
et enfin extraits avec de l'alcool phénol-chlorformisoamyl et de
l'alcool chloroformisoamyl, puis par précipitation à
l'éthanol. (Polen et al., 2006, cité par 2. WEBER, et al, 2005)
Des quantités équivalentes d'ARN de chaque lots ont
été utilisés pour générer des ADNc
marqués au Cy3-dUTP et au Cy5-dUTP comme décrit par Polen et al.,
2006. (cité par 2. WEBER, et al, 2005)
Expérimentation avec les puces à ADN et
analyse des résultats. L'hybridation sur les puces est
réalisées comme indiquée par Polen et al., 2006,
(cité par 2. WEBER, et al, 2005) et la fluorescence à 532nm
(Cy3-dUTP) et à 635nm (Cy5-dUTP) a été
déterminée avec le scanner GenePix 4000 (Axon Inc.) et
analysée avec le logiciel GenePixPro 3.0. Chaque expérience a
été réalisée en triplicat. Les gènes ont
été considéré comme s'exprimant
différentiellement si (i) le ration signal/bruit excédait 3, (ii)
le signal été présent sur au moins 2 des triplicats, (iii)
un test de Student montre que le taux d'ARN relatif aux contrôles (ADN
génomique) est significatif, (iv) les taux moyens de variation
étaient supérieurs à 3 sur chaque réplicat. Les
gènes ont été regroupés en groupes fonctionnelles
à l'aide des logiciels GenProtEC (http://genprotect.mbl.edu), MEME
(http://meme.sdsc.edu) et BioProspector
(http://robotics.standford.edu/~xliu/BioProspector/) (Liu et al., 2001,
cité par 2. WEBER, et al, 2005).
Résultats et discussion
L'étude du régulon óS basée sur
les fusions transcriptionnelles avec le gène rapporteur lacZ a
permis l'identification de 48 fusions régulées par óS. De
nombreuses fusions correspondaient à des opérons, les auteurs (3.
VIJAYAKUMAR, et al, 2004) considèrent que le nombre de gènes
régulés par RpoS découverts dans cette étude est
d'environ 80, car si une fusion avec un gène au sein d'un opéron
montre une dépendance à óS, c'est que le promoteur des
gènes de l'opéron entier est reconnu par RpoS, et donc tous les
gènes de l'opéron sont sous sa dépendance. Ces
gènes n'avaient pas été auparavant identifiés comme
faisant partie du régulon RpoS (anciennement considéré
comme comprenant environ 100 gènes), ce qui porte le nombre de
gènes exprimés par EóS à approximativement 200. Les
auteurs ont quantifié cette variabilité d'expression en comparant
les activités â-galactosidase de souches rpoS+
ou rpoS- portant la même fusion. 80% des fusions
présentaient une expression variant au moins d'un facteur 5 en
présence ou non de RpoS en phase stationnaire. Toutes les souches
étudiées présentaient une dépendance d'expression
du gène fusionné à óS aussi bien en milieu riche
qu'en milieu minimum, sauf la souche comprenant une fusion sur le gène
argH-oxyR, qui n'est dépendante de óS qu'en milieu riche :
ces gènes présenteraient une dépendance à óS
différente selon les conditions extérieures. Toutes les souches
montraient une activation par RpoS spécifique de la phase stationnaire,
mais certaines conservaient une expression basale en phase exponentielle, ce
qui s'explique par le fait que certains promoteurs de ó70 sont aussi
reconnus par óS. (table 1)
Pour identifier les gènes régulés par
óS, les auteurs (2. WEBER, et al, 2005) ont comparé les
transcriptomes des souches rpoS+ et rpoS::Tn10 de
MC4100. Les trois conditions d'expérimentation (phase stationnaire, choc
hyperosmotique et choc acide à pH=5) ont été
utilisées afin d'identifier le plus grand nombre possible de
gènes. 481 gènes (annexes pages 14, 15 et 16) s'exprimant au
moins 2 fois plus chez la souche rpoS+ que chez la souche
mutante (leur expression est donc activée par óS), et 95
gènes s'exprimant au moins 2 fois plus chez la souche mutante que chez
la souche rpoS+ (dont l'expression est donc
réprimée en présence de óS, cette répression
sera discutée ultérieurement) ont été
identifiés dans au moins une condition expérimentale. 140 des
gènes activés par óS le sont dans les 3 conditions
testées. Les 341 autres ne sont activés que sous une ou deux
conditions, particulièrement 186 gènes qui ne s'expriment
différentiellement que dans le cas du choc hyperosmotique. (figure 2)
Ceci indique que l'expression des gènes contrôlés par RpoS
ne suit pas toujours simplement les variations d'expression de RpoS
lui-même, c'est-à-dire que l'expression de certains gènes
du régulon semble dépendre du signal externe, et donc pourrait
être aussi régulés par d'autres facteurs. Il est possible
que ces gènes ne puissent être dépendant de óS pour
leur expression qu'en présence d'autres régulateurs. Par
conséquent, les données de cette expérience indiquent que
le régulon RpoS s'étendrait à environ 10% des gènes
d'E. coli, et serait soumis à des régulations
internes.
Les gènes régulés par óS sont
souvent répartis un peu partout sur le chromosome, mais on peut tout de
même définir certains clusters : par exemple, une
région de 91kb à 79.3min, comprenant 29 gènes
contrôlés par óS, dont des gènes codant pour des
régulateurs ou des gènes impliqués dans la
résistance au stress acide. Un autre exemple est un cluster de 13kb
comprenant l'opéron csiD-ygaF-gabDTP. Cet
opéron est intéressant car il présente une
régulation différentielle selon les conditions, comme
déterminé par Metzner, Germer, et Hengge (référence
1., 2004). (figures 3 et 4) Il été déterminé les
fonctions physiologiques des gènes régulés par óS
identifiés par cette étude. (pour 57% d'entre eux des annotations
fonctionnelles existent). (figure 5) 8% des gènes identifiés
comme dépendants de óS dans les 3 conditions produisent des
protéines régulatrices, 14% sont des transporteurs ou des
protéines membranaires ( les trafics membranaires et
réorganisation de la membrane plasmique sont importants pour la
réponse à certains stress, comme le stress osmotique ou les chocs
acides), 19% sont des gènes impliqués dans le métabolisme
(glycolyse, fermentation, respiration anaérobie, transport
d'électrons, cycle des pentoses phosphate), et 11% des gènes de
réponse à différents stress. (2. WEBER, et al, 2005)
Les fonctions physiologiques de gènes
identifiés par ces deux approches sont semblables. Il est à noter
que l'étude par puces à ADN montre de meilleurs
résultats : en effet, les techniques de préparation des
fusions lacZ et de sélection des fusions
óS-dépendantes ont pour effet de ne pas prendre en compte des
gènes régulés par óS. La méthode de
préparation des souches portant les fusions risque de ne pas concerner
tous les gènes du régulon, et comme il a été vu que
certains gènes n'étaient dépendants de RpoS pour leur
expression que sous certaines conditions, le fait de n'avoir effectué le
criblage qu'en phase stationnaire a exclu tous les gènes qui sont
régulés par RpoS dans d'autres conditions, comme les 181
gènes ne s'exprimant différentiellement qu'en conditions de choc
hyperosmotique. L'approche « puce à ADN » procure
des données permettant la vérification in vivo par fusion avec un
gène rapporteur de la dépendance à RpoS des 481
gènes identifiés.
Les régulations différentes des gènes du
régulon RpoS selon les conditions, ou les inhibitions d'expression de
gènes en présence de RpoS peuvent s'expliquer en partie par le
fait que de nombreux gènes régulés pas óS sont des
protéines régulatrices : une partie des gènes s'exprimant
différentiellement identifiés grâces aux puces à ADN
sont donc indirectement dépendants de RpoS. Les gènes
apparaissant réprimés le sont sûrement par des
répresseurs dont l'expression est elle directement
contrôlée par EóS.
II) Caractérisation des promoteurs du
régulon RpoS
Le facteur óS reconnait in vitro des
séquences -10 presques identiques à celles du facteur ó70
(CTATA(c/a)T pour EóS, et TATAAT pour EóS, Tanaka et al., 1993,
cité par 5. LACOUR, KOLB, LANDINI, 2003), ce qui peut s'expliquer par
une similarité importante des « DNA Binding
domaine » des deux facteurs ó (Lonetto et al., 1992,
cité par 5. LACOUR, KOLB, LANDINI, 2003). Cependant, le facteur
óS est aussi capable de reconnaitre des promoteurs qui lui sont
spécifique et ne reconnait pas certains promoteurs spécifiques
à ó70. Cette reconnaissance spécifique est importante pour
la compréhension des phénomènes de régulations
dépendants de RpoS. On peut expliquer de plusieurs manières la
reconnaissance d'un promoteur par óS : par une
spécificité de séquence du promoteur (auquel cas
ó70 ne pourrai pas reconnaitre le même promoteur), par la
nécessité d'une protéine régulatrice
supplémentaire indispensable à la reconnaissance d'un promoteur
donné par óS, ou par des facteurs intracellulaires, comme le
degré de compaction de l'ADN, l'alarmone ppGpp, la concentration
élévée en sels (Hengge-Aronis 1998 et 2002, Ishihama,
2000, Vicente et al. 1999, cités par 5. LACOUR, KOLB, LANDINI, 2003).
Des études ont été menées afin de
définir par biologie moléculaire une séquence consensus
pour les promoteurs reconnus exclusivement par óS, mais aussi pour
déterminer quelles étaient les bases moléculaire de la
reconnaissance des promoteurs par óS et ó70.
Lacour, Kolb, et Landini (2003, référence 5.)
ont étudié l'importance des nucléotides dans la
région -10 du promoteur du gène aidB, impliqué
dans la réponse aux agents alkylants de l'ADN. Ce gène est
régulé par la protéine Ada (Volkert et al., 1986), qui,
sous sa forme méthylée, peut activer la transcription par
Eó70 et EóS, mais en l'absence de Ada, l'expression de
aidB est strictement dépendante de EóS (in
vitro, Eó70 se fixe au promoteur PaidB en raison de
l'excès d'ARN polymérase Eó70 mais est incapable de former
le complexe ouvert et donc d'activer la transcription, et EóS peut
activer la transcription in vitro sans protéine annexe). Ce
promoteur (PaidB) est donc un modèle potentiel d'étude
des promoteurs exclusifs de óS ne nécessitant pas l'intervention
d'une autre protéine. Dans d'autres études, 7. CHECROUN, et al,
2004, et 6. LACOUR, et al, 2004 ont déterminé quelles
étaient les caratéristiques de DNA Binding Domaine de óS
et de ó70 qui étaient impliqués dans cette
sélectivité.
Matériels et méthodes
Souches et plasmides. Les souches
utilisées par LACOUR, KOLB, LANDINI(référence 5., 2003),
et LACOUR, et al, (référence 6. 2004), sont MV1161 et son
dérivé rpoS- MV2792 (souches
dérivées de K12). Ces souches ont été
transformées par le plasmide pRS1274, portant une fusion lacZ
sous le contrôle d'un promoteur PaidB sauvage ou mutant (dont
PaidB(C12T)). (figure 6) Les mutants de PaidB ont
été produits par PCR avec des amorces permettant la substitution
contrôlée de certains nucléotides, et insérés
dans le plasmide pRS1274 (pour la transcription in vivo) ou pJCD01
(pour la transcription in vitro), grâce aux sites de restriction
BamHI-EcoRI. (5. LACOUR, KOLB, LANDINI, 2003, et 6. LACOUR,
et al, 2004)
Les souches utilisées par 7. CHECROUN, et al, 2004 sont
de nombreux dérivés de MC4100 mutées sur rpoS (Diaz et
al.1991, cité par 7. CHECROUN, et al, 2004) et portant
différentes mutations sur le promoteur du gène osmEP (obtenues de
la même manière que les mutants PaidB, fig 7) en fusion
avec lacZ. Les souches sont transformées par le plasmide
pBDArpoS décrit ci-dessous, et sont cultivées en LB
avant d'être déposées sur milieu de MacConkey (colonies
rouges si lac+)
Obtention de facteurs sigma mutants. Les
protéines ó70 mutés et taggés 12-Histidine ont
été obtenues en mutant directement le gène RpoD (codant
ó70) sur le plasmide pET-21ó grâce au kit QuickChange
(Strategene). Les mutations effectuées donnent pour phénotype
ó70(Q437H), ó70(T440I) et
ó70(T440E). Les óS mutants ont été
obtenus par amplification PCR du plasmide pBADrpoS (contenant
rpoS sous contrôle du promoteur araB) avec amorces
permettant l'introduction contrôlées de mutations. Ces mutations
ont été localisées dans les régions 2 et 4 de
óS grâce au choix des primers (6. LACOUR, et al, 2004, et 7.
CHECROUN, et al, 2004). Les mutants óS étudiés par 6.
Lacour et al., 2004 portent les acides aminés Q152A et E155A.
Transcription In Vivo. Les
bactéries ont été cultivées une nuit en LB à
37°C, puis les cultures ont été diluées à
DO600nm=0.02 pour réduire l'activité
â-galactosidase. L'activité â-galactosidase a
été mesurée comme indiqué
précédement, et comparée à celle mesurée
chez une souche transformée par un plasmide pRS1274 sans promoteur
(contrôle). (5. LACOUR, KOLB, LANDINI, 2003, 6. LACOUR, et al, 2004, et
7. CHECROUN, et al, 2004)
Purification des facteurs ó et de l'ARN
polymérase coeur, reconsitution de l'holoenzyme. Les
protéines ont été purifiées selon le protocole
décrit par Tanaka et al., 1995 et Gribskov et al., 1983 (cités
par 5. LACOUR, KOLB, LANDINI, 2003). L'holoenzyme (EóS ou Eó70) a
été reconstituée par incubation de l'enzyme coeur avec
soit óS soit ó70 au ratio 1:4 pendant 30 min à 37°C.
(5. LACOUR, KOLB, LANDINI, 2003, 6. LACOUR, et al, 2004, et 7. CHECROUN, et al,
2004)
Transcription In Vitro. L'expérience
de transcription unique a été réalisée dans un
tampon K-glu200 avec le plasmide pJCD01 (3nM) surenroulé portant soit le
PaidB sauvage soit un des mutants créés, et l'holoenzyme
EóS(50nM) ou Eó70(100nM), ainsi que les dNTP (500ìM) dont
du [á32P]UTP et incubés 15min à 37°C. La
réaction a été arrêtée par addition de
10ìL de tampon (formamide, EDTA, xylène cyanol et bleu de
bromophénol). Après chauffage à 65°C, les
échantillons ont été déposés sur un gel de
polyacrylamide de séquençage à 7%. La quantification des
produits de transcription a été réalisée avec un
PhosphorImager et normalisée à la production standart d'ARN. (5.
LACOUR, KOLB, LANDINI, 2003, 6. LACOUR, et al, 2004, et 7. CHECROUN, et al,
2004)
Gel Retard. L'holoenzyme reconstituée
Eó70 ou EóS (5 à 50 nM) et des fragments d'ADN de 180pb
correspondants aux promoteurs (1nM) ont été incubés
pendant 16min à 37°C, puis déposés sur une gel de
polyacrylamide non dénaturant à 5%. (5. LACOUR, KOLB, LANDINI,
2003, et 6. LACOUR, et al, 2004)
Dosage â-galactosidase. La mesure de
l'activité â-galactosidase a été effectuée
comme indiqué précédement, en absence d'arabinose. (7.
CHECROUN, et al, 2004)
Résultats et discussion
Un promoteur consensus ? Les auteurs Lacour,
Kolb, et Landini (référence 5., 2003) ont choisi de focaliser
les mutations du promoteur PaidB sur quatres
éléments : (i) un dinucléotide TG, typique des
promoteurs ó70-dépendants à boîte -10
étendue, mais ici à une position unusuelle (-16/-15 au lieu de
-13/-12, cette position ne permet pas la transcription par Eó70), (ii)
le résidu -13C (Eó70 a besoin absolument d'un T à cette
position), (iii) le résidu -12C, premier nucléotide de
l'hexamère -10, (iv) enfin, le -6A a été muté en T
car un T à cette position semble conservé dans les autres
promoteurs óS-dépendants. Globalement, il a été
montré grâce au gel retard que EóS (l'ARN polymérase
chargée du facteur sigma S) a plus d'affinité pour PaidB
que Eó70. In vivo, il a été confirmé
l'incapacité de Eó70 à activer la transcription si le
dinucléotide TG est en position -16/-15 au lieu de -13/-12, alors que
EóS en est capable : il est suggéré que ceci est dû
à la flexibilité du domaine 2.5 (reconnaissance de la boîte
-10) de óS. La substitution ou délétion du C en -13 a pour
effet la perte de spécificité de la reconnaissance du promoteur
par óS. Particulièrement, la substitution du C en G favorise
grandement la reconnaissance par ó70. On en conclue que ce
nucléotide -13C aide beaucoup à la fixation et à la
formation du complexe ouvert par EóS, ce qui confirme des observations
effectuées sur d'autres promoteurs de gènes du régulon
RpoS. La substitution du -6A n'a eu aucun effet sur la transcription de
aidB, ce qui prouve que les nucléotides apparaissant
conservés dans les analyses in silico des promoteurs n'ont pas
toujours de signification physiologique, ou alors ce nucléotide a une
importance chez d'autres promoteurs mais n'a pas de rôle à jouer
chez PaidB. La substitution du C en -12 en T améliore
grandement la transcription par Eó70 (quasi nulle si le
nucléotide -12 est un C) : la sélectivité de EóS
est perdue si cette position est occupée par un T. Les auteurs ont
étudiés les promoteurs de 57 autres gènes
dépendants à óS, et on constaté que 33% d'entre eux
avaient autre chose qu'un T à cette position (dont 16% de C). Des
expériences complémentaire de mutagénèse sur
certains de ces promoteurs leur on permis de déterminer deux classes de
promoteurs óS-dépendants : ceux qui possèdent autre chose
qu'un T en -12, et qui sont strictement dépendants de óS sans
nécessiter de facteurs additionnels, et ceux qui peuvent être
reconnus par ó70, possédants toujours un T en premier position de
l'hexamère, et pour lesquels la sélectivité
ó70/óS dépend d'un autre facteur (protéine
additionnelle).
Les expériences de transcription in vitro
confirment parfaitement ces observations, à la différence que
l'excès d'holoenzyme peut parfois forcer la fixation sur le promoteur,
les résultats ne sont donc pas du même ordre. Ces
expériences ont donc permis de déterminer l'importance
physiologique de la conservation de certains nucléotides des promoteurs
óS dépendants. Les auteurs proposent alors un nouveau consensus
pour les promoteurs strictement dépendants de RpoS :
TG(N)0-2CCATA(a/c)T, opposée à TGGTATAAT,
séquence optimale pour la reconnaissance par Eó70. Un recherche
dans la base de donnée Colibri montre que cette séquence est
retrouvée dans les 200pb précédent 14 ORF ou
opérons en plus de aidB.
La sélectivité sans facteurs additionnels peut
alors être expliquée par une tolérance accrue de óS
aux divergence de la séquence -10 des promoteurs par rapport à
ó70 : ó70 ne supporte pas la substitution du T en -12, et ne peut
pas permettre la formation du complexe ouvert si le dinucléotide TG
n'est pas en position -13/-12. óS lui peut reconnaitre les promoteurs
divergeants sur ces points, avec (dans le cas du TG) ou sans (dans le cas du
-12T) l'aide de protéines additionnelles. (5. LACOUR, KOLB, LANDINI,
2003).
Mécanismes moléculaires de la
sélectivité ó70/óS. Le gène
osmEP dépend de ó70 en phase stationnaire, et de
óS en phase exponentielle, et de mutants de ce promoteurs ont
été isolés. Parmi eux, osmEP13T, qui
possède un T à la place du C normalement présent en -13.
Ce mutants et d'autre mutants de osmEP (comme osmEP32G) ont
été utilisés par Checroun et al (référence
7., 2004) pour identifier de mutants de RpoS supprimant ces mutations, afin de
caractériser les interactions promoteur-acides aminés
impliquées dans la sélectivité de óS. Le criblage
est effectué sur milieu MacConkey : les colonies rouges (lac+)
portent un óS capable de transcrire le gène rapporteur sous
contrôle d'un promoteur osmEP muté. Les mutants
suppresseurs obtenus sont indiqué table 2. Les mutations supresseurs
n'affectent que trois acides aminés : Q152, E155 et R299, qui
appartiennent aux régions 2.4 (Q152 et E155), correspondant à la
zone impliquée dans la reconnaissance de la boîte -10 chez
ó70, et 4.2 (R299) semblable à la zone impliquée dans la
reconnaissance de la boîte -35 chez ó70. Ces acides aminés
correspondent aux Q437, T440 et R584 chez ó70. (figure 8) Les
activités de transcription des promoteurs mutants par ces óS
mutants ont été mesurées (figure 9). Des homologues de
óS chez 21 autres bactéries Gram- portent les même acides
aminés à ces mêmes positions. Il résulte de cette
étude que R299 interagit avec la guanine du dinucléotide CG en
position -31, mais cette interaction ne semble qu'amélorier la
reconnaissance du promoteur. En effet, de nombreux promoteurs óS
dépendants sont pourvus de boîte -35 très
dégénénées ou n'en ont pas (Espinosa-Urgel et al.
1996, cité par 7. CHECROUN, et al, 2004). De plus, il est
démontré que ce sont les acides aminés Q152 et E155 qui
procurent la capacité à óS de reconnaitre les C en
position -13, comme le montre la supression de la mutation osmEP13C
par le mutant rpoS Q152R. Des données structurales sur
l'analogue de óS chez Thermus aquaticus montre que les
équivalents de ces deux acides aminés appartiennent à deux
á-hélices formant une pince sur le grand sillon au niveau des
nucléotides -12/-13, ce qui expliquera leur rôle majeur dans la
reconnaissance de ces nucléotides (Protein Data Bank).
Une autre étude semblable, menée par Lacour, et
al (référence 6, 2004), utilisant le promoteur PaidB (sauvage ou
portant une substitution du -12C en T), visait à identifier des mutants
de ó70 capable de transcrire ce promoteur. (figure 10) Il en a
été conclu que les facteurs ó70 mutés Q437H et
T440E étaient capables de former un complexe ouvert compétent
avec le promoteur PaidB qui porte un C en -13, mais conservait une
infériorité par rapport à óS vis-à-vis de
l'affinité et de l'efficacité de transcription. La grande
spécificité de óS pour les promoteurs comprenant ce
nucléotide et sa capacité à les transcrire efficacement
dépend donc en majeure partie de ces deux nucléotides.
Conclusion :
Le régulon RpoS, de part son recouvrement partiel avec
le régulon ó70, et le fait qu'il comprenne de nombreuses
protéines régulatrices, certaines ayant même pour cible des
protéines du régulon RpoS, possède une architecture
très complexe, et dynamique puisque cette architecture elle-même
est soumise à régulation. (2. WEBER, et al, 2005) Le
régulon RpoS recoupe également ceux d'autres régulateurs
globaux, comme la protéine Lrp, ou CRP-AMPc, lui-même
régulateur de RpoS. Ce dynamisme permet un intégration fine des
signaux externes et une adaptation précise de la réponse
physiologique par une régulation génétique globale. La
compréhension accrue de ce mécanisme est importante puisqu'elle
permettra de mieux comprendre les états physiologiques des
bactéries, pour des applications dans la recherche fondamentale,
l'industrie ou encore la médecine. Par exemple, chez E.
coli, l'expression de RpoS ne semble pas dépendre d'une
perception de signaux de quorum sensing externes (elle ne possède pas de
système de quorum sensing mais est capable de percevoir ceux des autres
espèces), qu'en est-il chez d'autres espèces ? (8.
HENGGE-ARONIS, 2002)
De nombreuses questions restent à
élucider : ici il n'a été traité que de
E. coli, et des études sont menées sur le genre
Salmonella et sur Thermus
Aquaticus, mais pour l'instant presque rien n'est connu sur un
grand nombre de procaryotes d'intérêt publique.
Bibliographie :
1. MARTIN METZNER, JENS GERMER, REGINE HENGGE.
Multiple stress signal integration in the regulation of the
complex sigma-S-dependent csiD-ygaF-gabDTP operon in Escherichia
coli.
Molecular Microbiology (2004) 51 (3), 799-811
Regulation of the
2. HARALD WEBER, TINO POLEN, JOHANNA HEUVELING, VOLKER F.
WENDISCH, REGINE HENGGE.
Genome-Wide Analysis of the General Stress Response Network in
Escherichia coli: óS-Dependent Genes, Promoters, and Sigma
Factor Selectivity.
Journal of bacteriology, Mar. 2005, p. 1591-1603 Vol. 187, No.
5
3. SOMALINGA R. V. VIJAYAKUMAR, MARK G. KIRCHHOF, CHERYL L.
PATTEN, HERB E. SCHELLHORN.
RpoS-Regulated Genes of Escherichia coli Identified
by Random lacZ Fusion Mutagenesis.
Journal of bacteriology, Dec. 2004, p. 8499-8507 Vol. 186, No.
24
4. STEPHAN LACOUR, PAOLO LANDINI.
óS-Dependent Gene Expression at the Onset of Stationary
Phase in Escherichia coli : Function of óS-Dependent Genes and
Identification of Their Promoter Sequences.
Journal of bacteriology, Nov. 2004, p. 7186-7195 Vol. 186, No.
21
5. STEPHAN LACOUR, ANNIE KOLB, PAOLO LANDINI.
Nucleotides from -16 to -12 Determine Specific Promoter
Recognition by Bacterial óS-RNA Polymerase.
The journal of biological chemistry Vol. 278, No. 39, Issue of
September 26, pp. 37160-37168, 2003
6. STEPHAN LACOUR, OLIVIER LEROY, ANNIE KOLB, PAOLO
LANDINI.
Substitutions in Region 2.4 of ó70 Allow Recognition of
the óS-Dependent aidB Promoter.
The journal of biological chemistry Vol. 279, No. 53, Issue of
December 31, pp. 55255-55261, 2004
7. CLAIRE CHECROUN, PATRICIA BORDES, OLIVIER LEROY, ANNIE
KOLB, CLAUDE GUTIERREZ.
Interactions between the 2.4 and 4.2 regions of óS, the
stress-specific ó factor of Escherichia coli, and the -10 and -35
promoter elements.
Nucleic Acids Research, 2004, Vol. 32, No. 1 45-53
8. REGINE HENGGE-ARONIS
Signal Transduction and Regulatory Mechanisms Involved in
Control of the óS (RpoS) Subunit of RNA Polymerase.
Microbiology and molecular biology reviews, Sept. 2002, p.
373-395 Vol. 66, No. 3
9. F. REPOILA, N. MAJDALANI, S. GOTTESMAN.
Small non-coding RNAs, co-ordinators of adaptation processes
in Escherichia coli : the RpoS paradigm.
Molecular Microbiology (2003) 48 (4), 855-861
10. MATTHEW HIRSCH, THOMAS ELLIOTT.
Stationary-Phase Regulation of RpoS Translation in
Escherichia coli.
Journal of bacteriology, Nov. 2005, p. 7204-7213 Vol. 187, No.
21
11. RICHARD A. LEASE, DORIE SMITH, KATHLEEN MCDONOUGH, MARLENE
BELFORT.
The Small Noncoding DsrA RNA Is an Acid Resistance Regulator
in Escherichia coli.
Journal of bacteriology, Sept. 2004, p. 6179-6185 Vol. 186,
No. 18
Annexes :
Ratios d'expression des gènes contrôlés
par óS identifiés par puces à ADN. (2. WEBER, et al.
2005)

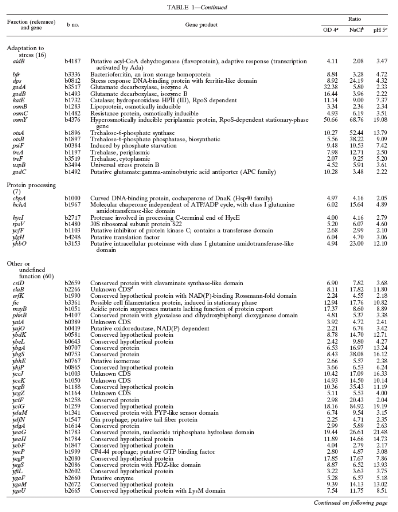
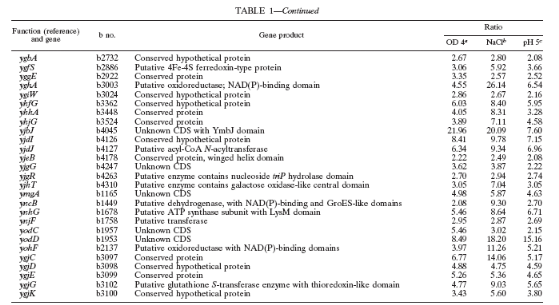

Figure 1. Crible génétique
utilisé pour identifier la dépendance à RpoS de
gènes en fusion avec lacZ. (3. VIJAYAKUMAR, et al, 2004)
Table 1. Gènes régulées
par RpoS identifiés grâce aux fusions lacZ, et leurs
fonction physiologiques. (3. VIJAYAKUMAR, et al, 2004)

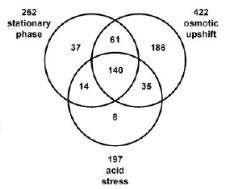
Figure 2. Répartition des fonctions
des gènes régulés par RpoS identifiés grâce
aux puces à ADN. (2. WEBER, et al, 2005)
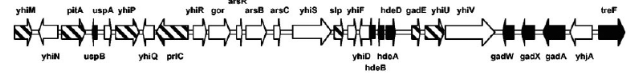
Figure 3. Le cluster
csiD-ygaF-gabDTP, situé à environ
79min sur le chromosome de E. coli. En noir, les gènes
régulés par RpoS dans les trois conditions,
hachurés : les gènes présentant une régulation
par RpoS seulement dans une ou deux conditions. (2. WEBER, et al, 2005)
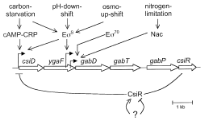
Figure 4. Régulations de
l'opéron csiD-ygaF-gabDTP. (1. METZNER,
GERMER, HENGGE, 2004)
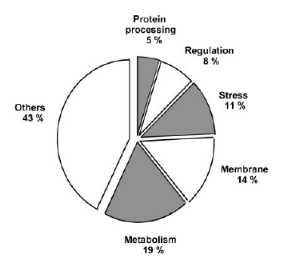
Figure 5. Fonctions physiologiques des
gènes identifiés par puces à ADN. (2. WEBER, et al,
2005)

Figure 6. Le promoteur PaidB. En
italique et avec astérisques, les nucléotides ciblés pour
la mutagénèse. (5. LACOUR, KOLB, LANDINI, 2003)

Figure 7. Le promoteur osmEP et les mutants
utilisés. (7. CHECROUN, et al, 2004)
Table 2. Mutants RpoS supresseurs obtenus
après criblage. (7. CHECROUN, et al, 2004)
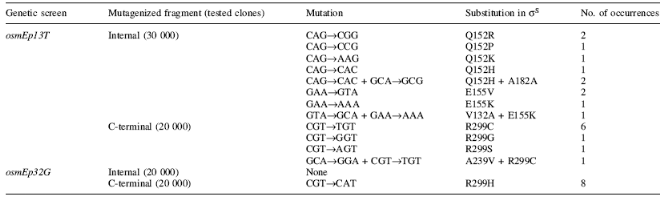
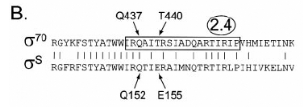
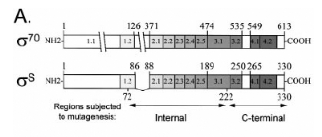
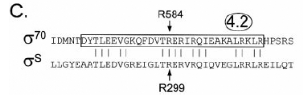 Figure 8. Organisation en domaines des protéines
ó70 et óS. (A) Organisation générale et indication
des zones soumises à la mutagénèse. (B) Alignement des
domaines 2.4 de ó70 et óS. La région 2.4 de ó70 est
encadrée. (C) Alignement des domaines 4.2 de ó70 et óS, la
région 4.2 de ó70 est encadrée. (7. CHECROUN, et al,
2004)
Figure 8. Organisation en domaines des protéines
ó70 et óS. (A) Organisation générale et indication
des zones soumises à la mutagénèse. (B) Alignement des
domaines 2.4 de ó70 et óS. La région 2.4 de ó70 est
encadrée. (C) Alignement des domaines 4.2 de ó70 et óS, la
région 4.2 de ó70 est encadrée. (7. CHECROUN, et al,
2004)
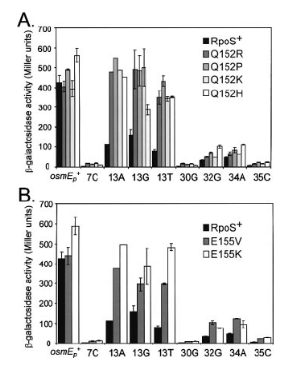
Figure 9. Effets des mutations de RpoS
affectant Q152 et E155 sur la transcription de promoteurs osmE
sauvages. Mesure des activités â-galactosidase des fusions
effectuées. (7. CHECROUN, et al, 2004)

Figure 10. Quantification de la transcription
de aidB (ration expression/expression du contrôle),
exprimée en pourcentages de l'activité observée avec
Eó70 sauvage. En blanc : PaidB sauvage, en noir,
PaidBC12T. (6. LACOUR, et al. 2004)
L'étude des mécanismes physiologiques et
génétiques de la réponse aux stress chez
les bactéries a de grand intérêts dans les domaines de la
recherche fondamentale, médicale, agro-alimentaire et industrielle. Chez
Escherichia coli, l'eubactérie la plus
étudiée, l'acteur majeur de cette réponse est le
facteur sigma alternatif RpoS (óS), une des sept sous
unité de l'ARN polymérase responsable de la reconnaissance des
promoteurs et de l'initiation de la transcription. Le
régulon RpoS est large et complexe, en partie parcequ'il
recoupe le régulon de ó70, le facteur sigma principal,
responsable de l'entretien cellulaire. Le présent rapport compile des
études visant à caractériser le régulon RpoS, tant
au niveau des gènes qui en font partie qu'au niveau des
mécanismes génétiques et protéiques de la
sélectivité RpoS/ó70.



